文献介绍

文献题目: 使用自适应图注意自动编码器从空间解析的转录组学中解读空间域
研究团队: 张世华(中国科学院数学与系统科学研究院)
发表时间: 2022-04-01
发表期刊: Nature Communications
影响因子: 17.6(2022年)
DOI: 10.1038/s41467-022-29439-6
摘要
空间分辨转录组学的最新进展使得能够全面测量基因表达模式,同时保留组织微环境的空间背景。破译组织中 spots 的空间背景需要仔细使用它们的空间信息。为此,作者开发了一个图注意力自动编码器框架 STAGATE,通过整合空间信息和基因表达谱来学习低维潜在嵌入,从而准确识别空间域。为了更好地表征空间域边界的空间相似性,STAGATE 采用注意力机制来自适应学习相邻点的相似性,并通过集成基因表达的预聚类来选择细胞类型感知模块。作者在具有不同空间分辨率的不同平台生成的不同空间转录组数据集上验证了 STAGATE。STAGATE 可以显着提高空间域的识别精度,并在保留空间表达模式的同时对数据进行去噪。重要的是,STAGATE 可以扩展到多个连续切片,以减少切片之间的批次效应,并有效地从重建的 3D 组织中提取三维 (3D) 表达域。
前言
复杂组织的功能从根本上与不同细胞类型的空间背景相关。组织中转录表达的相对位置对于理解其生物学功能和描述交互式生物网络至关重要。空间分辨转录组学 (STs) 的突破性技术,例如 10x Visium、Slide-seq、Stereo-seq 和 PIXEL-seq,能够以多个细胞甚至亚细胞分辨率对捕获位置(称为 spots)中的基因表达进行全基因组分析。
解读空间域(即具有相似空间表达模式的区域)是 STs 面临的巨大挑战之一。例如,人类大脑皮层的层状组织与其生物学功能尤其相关,其中位于不同皮层内的细胞通常在表达、形态和生理上有所不同。大多数现有的聚类方法都没有有效地利用可用的空间信息。这些非空间方法大致可以分为两类。第一类使用传统的聚类方法,如 k-means 和 Louvain 算法。这些方法根据 ST 技术的不同分辨率限制于少量的 spots 或稀疏性,并且聚类结果在组织切片中可能不连续。第二类利用单细胞 RNA 测序定义的细胞类型特征来反卷积 spots。它们不适用于细胞或亚细胞水平分辨率的 ST 数据。
一些最近的算法通过考虑相邻 spots 之间的相似性来调整聚类方法,以更好地解释基因表达的空间依赖性。这些方法在识别脑和癌症组织切片的空间域方面显示出显着的改进。例如,BayesSpace 是一种贝叶斯统计方法,它通过在先验中引入空间邻居结构来鼓励相邻点属于同一个聚类。Giotto 通过实现具有空间邻居先验的隐马尔可夫随机场 (HMRF) 模型来识别空间域。stLearn 根据从组织学图像中提取的特征定义形态距离,并利用这些距离以及空间邻居结构来平滑基因表达。SEDR 采用深度自动编码器网络来学习基因表示,并使用变分图自动编码器同时嵌入空间信息。SpaGCN 还应用图卷积网络来整合基因表达和空间位置,并进一步与自监督模块相结合以识别域。此外,最近开发的一种名为 RESEPT 的方法利用监督图像分割方法进行组织结构识别。尽管这些方法考虑了 ST 的空间结构,但它们定义的相邻点的相似性是在训练之前预先定义的,无法自适应地学习。此外,这些方法没有更详细地考虑空间域边界处 spots 的空间相似性,也不能很好地整合空间信息来推断和去噪基因表达。更重要的是,这些方法不能应用于多个连续的切片来重建三维 (3D) ST 模型并提取 3D 表达域(Supplementary Table S1)。
为此,作者开发了 STAGATE,通过集成空间信息和基因表达谱来学习低维潜在嵌入,使用自适应图注意力自动编码器从空间解析转录组学中准确识别空间域。对不同平台(例如 10x Visium、Slide-seq 和 Stereo-seq)生成的 ST 数据作为基准进行了广泛的测试和与现有方法的比较,证明了其在空间域识别、可视化、空间轨迹推断、数据去噪、3D 表达域提取等下游分析任务中的优越性。
研究结果
1. STAGATE 概述
STAGATE 首先根据 spots 的相对空间位置构建空间邻居网络 (SNN),并根据基因表达的预聚类选择性地通过修剪 SNN 来引入细胞类型感知 SNN(Fig. 1)。基因表达预聚类可以有效识别包含不同细胞类型的区域,因此这种细胞类型感知的 SNN 可以帮助更好地表征低空间分辨率的 ST 数据(例如 10x Visium)在这些不同空间域边界的空间相似性(参见 Methods 的“细胞类型感知 SNN 的构建(可选)”小节)。
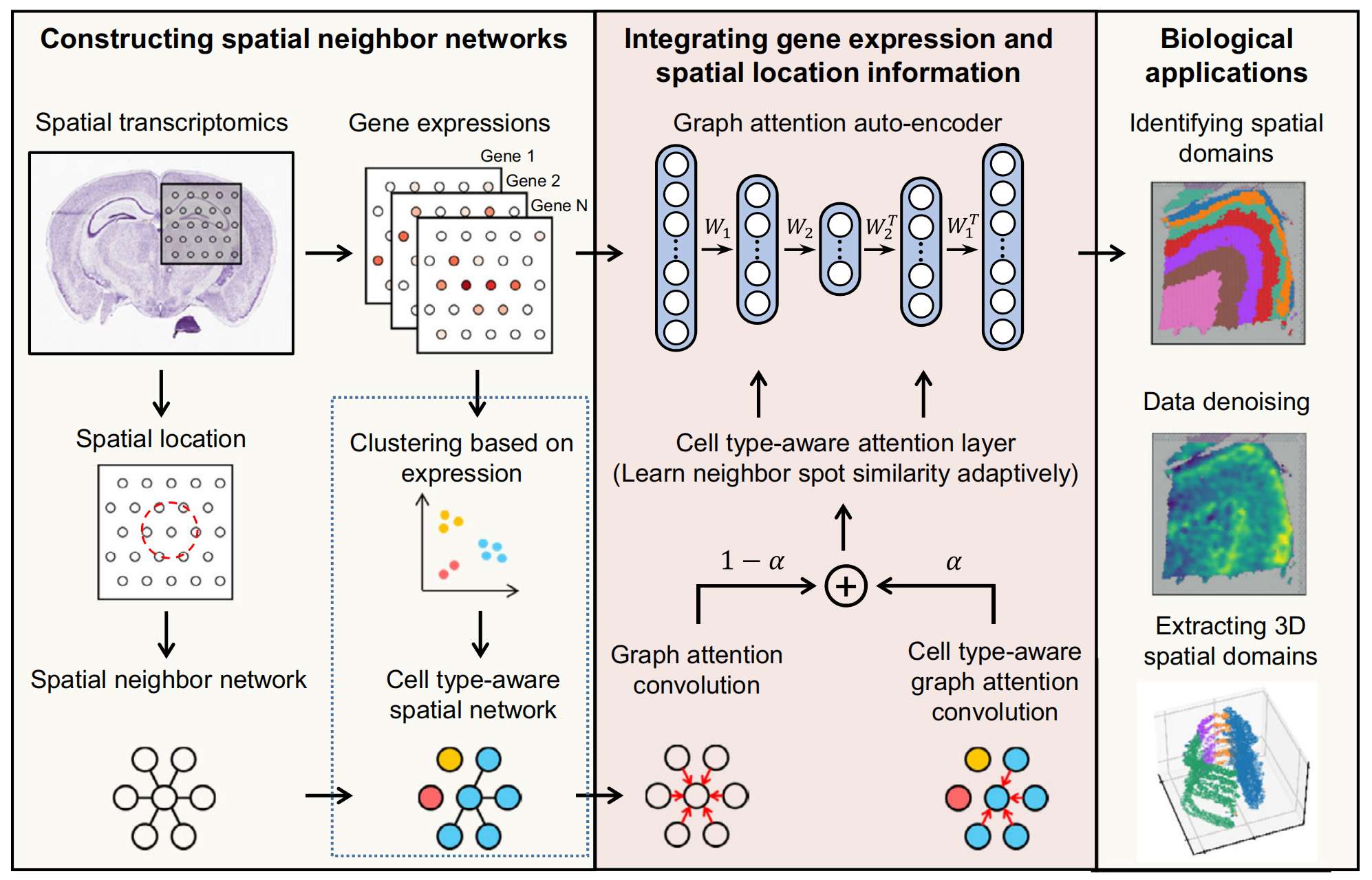
STAGATE 首先根据预定义的半径构建一个空间邻居网络(SNN),并在虚线框中为10x Visium 数据构建另一个可选的网络,根据基因表达的预聚类对其进行剪枝,以更好地表征空间相似性空间域的边界。STAGATE 通过图注意力自动编码器进一步学习具有空间信息和基因表达的低维潜在表示。自动编码器的输入是归一化的表达矩阵,编码器和解码器中间采用图注意力层。STAGATE 的输出可用于识别空间域、数据去噪和提取 3D 空间域。
然后,STAGATE 通过图注意力自动编码器学习具有空间信息和基因表达的低维潜在嵌入(Fig. 1)。每个 spot 的标准化表达首先由编码器转换为 d 维潜在嵌入,然后通过解码器反转回重建的表达谱。与经典的自动编码器不同,STAGATE 在编码器和解码器的中间层采用了注意力机制。它自适应地学习 SNNs 的边缘权重(即相邻点之间的相似性),并进一步使用它们通过集体聚合来自其邻居的信息来更新 spot 表示。最后,潜在嵌入用于通过 UMAP 可视化数据,并通过各种聚类算法(例如 mclust 和 Louvain)识别空间域(Fig. 1)。
2. STAGATE 改进了人类背外侧前额皮质数据集上已知层的识别
为了定量评估 STAGATE 的空间聚类性能,作者首先将其应用到包含 12 个人类背外侧前额叶皮层 (DLPFC) 切片的空间表达的 10x Visium 数据集。Maynard et al. 根据形态特征和基因标记手动注释 DLPFC 层和白质 (WM)(Fig. 2a)。考虑到这一点,作者将 STAGATE 的聚类精度与 SCANPY 实现的非空间聚类方法以及最近开发的五种空间聚类方法(Giotto、BayesSpace、stLearn、SpaGCN 和 SEDR)在调整后的兰德指数(ARI)、归一化互信息(NMI)、同质性得分(HS)方面进行了比较(参见 Supplementary Information 中的“与其他空间域识别方法的比较”小节)。
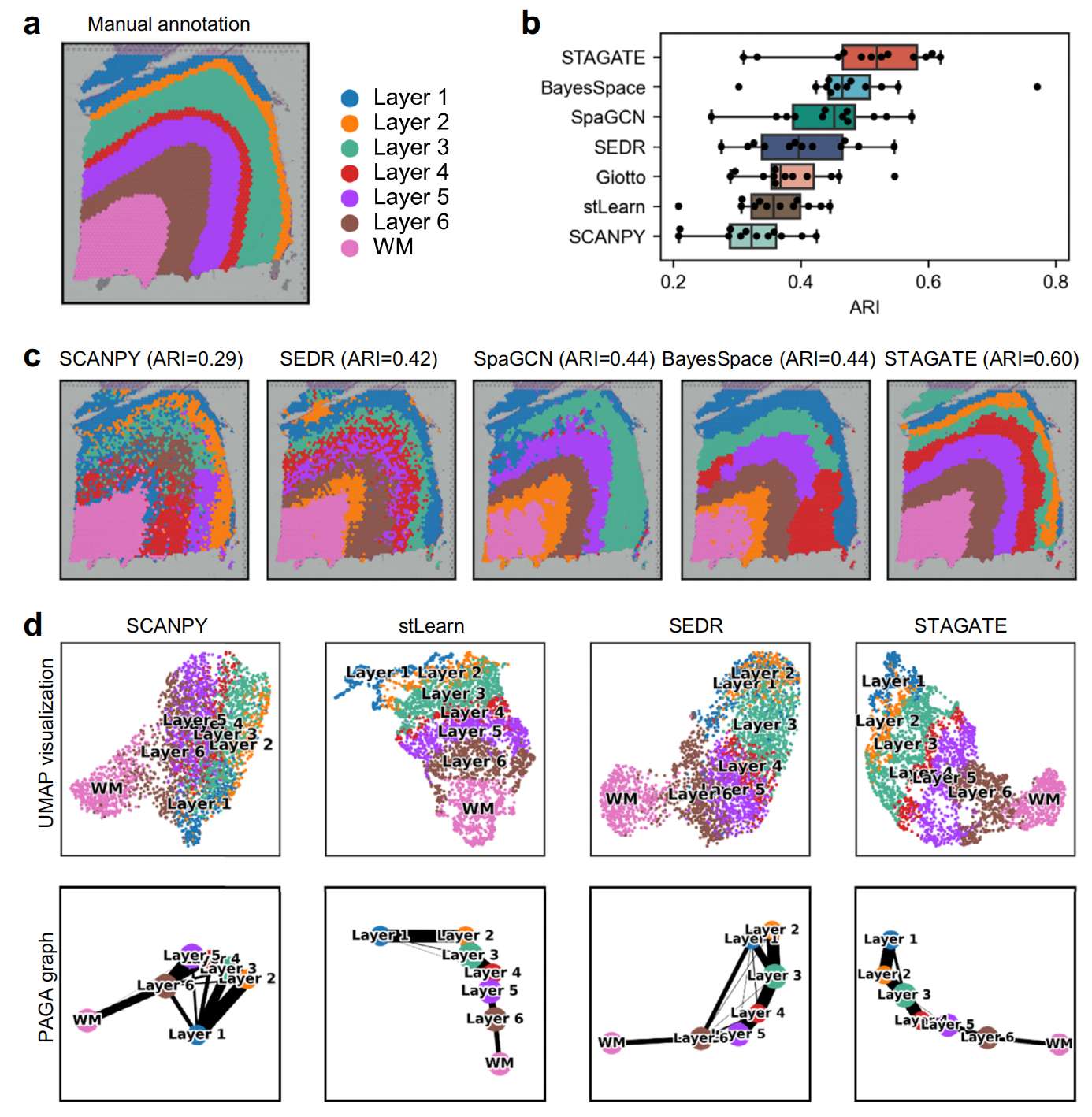
a. DLPFC 切片 151676 中皮质层和白质 (WM) 的 ground-truth 分割。
b. 七种方法的调整兰德指数 (ARI) 得分显示 DLPFC 数据集所有 12 张切片的聚类准确度箱线图。在箱线图中,中心线、箱线界限和晶须分别表示中位数、上四分位数、下四分位数和 1.5×四分位距。
c. SCANPY、SEDR、SpaGCN、BayesSpace、STAGATE 在 DLPFC 切片 151676 中生成的聚类分配。
d. SCANPY、stLearn、SEDR、STAGATE 嵌入分别在 DLPFC 切片 151676 中生成的 UMAP 可视化和 PAGA 图。作为端到端聚类方法,SpaGCN 和 BayesSpace 无法使用 UMAP 和 PAGA 进行可视化。
STAGATE 可以有效识别预期的皮质层结构,并且与其他方法相比取得了显著的改进(Fig. 2b and Supplementary Figs. S1, S2)。例如,在 DLPFC 切片 151676 中,STAGATE 清晰地划定了层边界并实现了最佳聚类精度(ARI = 0.60)(Fig. 2c)。相比之下,非空间方法 SCANPY 的聚类分配可以大致遵循本节中的预期层模式,但其聚类边界不连续且存在许多异常值,这损害了其聚类精度。有趣的是,利用空间信息的算法的性能明显优于非空间聚类方法 SCANPY。这些结果证明了 STAGATE 在空间域识别方面的优势以及使用空间信息的必要性。此外,作者通过比较不同超参数的聚类精度进一步测试了 STAGATE 的鲁棒性,发现 STAGATE 对编码器结构和潜在维度不敏感(Supplementary Fig. S2c–e)。
空间信息的整合使 STAGATE 能够揭示空间域之间的距离并在 UMAP 图中描绘空间轨迹。例如,在 DLPFC 切片 151676 中,皮质层组织良好,并在由 STAGATE 嵌入生成的 UMAP 图中显示出一致的空间轨迹(从第 1 层到第 6 层和白质)(Fig. 2d and Supplementary Fig. S3)。该结果与相邻皮质层之间的功能相似性以及时间顺序相一致。相比之下,在 SCANPY 嵌入的 UMAP 图中,属于不同层的 spots 没有被清楚地分开。对于另外两种空间聚类方法,stLearn 没有清楚地区分 WM 和皮质层,而 SEDR 混合了第 1 层和第 6 层的 spots。作者使用名为 PAGA 的轨迹推断算法进一步确认了推断的轨迹(Fig. 2d)。STAGATE 和 stLearn 嵌入的 PAGA 图均显示出从第 1 层到第 6 层的近似线性发展轨迹以及相邻层之间的相似性,而 SCANPY 和 SEDR 嵌入的 PAGA 结果则是混合的。
3. STAGATE 能够从不同空间分辨率的 ST 数据中识别组织结构
作者进一步测试了 STAGATE 是否可以应用于不同空间分辨率的 ST 数据。作者首先将 STAGATE 应用于来自小鼠海马的具有 10 μm 空间分辨率的 Slide-seqV2 数据集。与分辨率为 55 μm 的 10x Visium 平台相比,Slide-seqV2 可以在细胞水平的分辨率下分析空间表达,具有更多的 spots(每个切片 >10,000 个),但每个 spot 的测序深度更少(Supplementary Table S2)。正如预期的那样,使用具有相同参数的 Louvain 聚类算法,STAGATE 可以很好地表征组织结构并揭示空间域,而 SCANPY 和 SEDR 识别的聚类缺乏明显的空间分离(Fig. 3a and Supplementary Fig. S4)。例如,STAGATE 描绘了海马区域中清晰的“索状”结构以及“箭头状”结构,并确定了它的四个空间域。这一结果与 Allen Reference Atlas 对海马结构的注释一致(Fig. 3b),其中“索状”结构对应 Ammon 角的锥体层,可进一步分为 CA1、CA2 和 CA3 区(即 CA1sp、CA2sp 和 CA3sp),“箭头状”结构对应齿状回的颗粒细胞层(即 DG-sg)。虽然由于 spot 数较少,CA2sp 域没有单独聚类,但它在 STAGATE 嵌入的 UMAP 图中被分开(Supplementary Fig. S5)。此外,许多已知基因标记的表达也验证了 STAGATE 的聚类划分(Fig. 3c and Supplementary Fig. S6)。例如,Itpka 和 Bcl11b 在 Ammon 角域之间表现出差异表达,并且如预期的那样在 CA1sp 中高度表达。已知的海马 CA2 分子标记如 Amigo2 和 Pcp4 在已鉴定的 CA2sp 域中特异性表达。此外,已发现介导齿状回颗粒细胞兴奋性突触发育的 Lrrtm4 在已鉴定的 DG-sg 区域特异性表达。除了这些已知的组织结构之外,STAGATE 还鉴定了许多分离良好的空间域,并通过差异表达分析揭示了它们的空间基因表达模式(Supplementary Fig. S6)。例如,海马内除“索状”和“箭状”结构之外的区域(domain 6)表现出星形胶质细胞基因标记 Ddn 和 Camk2a 的强烈表达。海马区域周围的区域(domain 7)表达许多少突胶质细胞相关的基因标记,如 Trf 和 Mobp。此外,作者还在空间域 3 和 4 中观察到了显著的空间表达模式,其中分别以 Enpp2 和 Nwd2 表达为主。这些结果表明 STAGATE 可以解剖空间异质性并进一步揭示空间表达模式。作者还在通过 Slide-seq 和 10x Visium 技术分析的小鼠海马切片上测试了 STAGATE。作为 Slide-seqV2 的初始版本,Slide-seq 的转录本检测灵敏度相对较低(Fig. 3d)。STAGATE 分别在 Slide-seq 数据(Fig. 3e)和 10x Visium 数据(Fig. 3f)上很好地描绘了除 CA2sp 之外的已知组织结构。
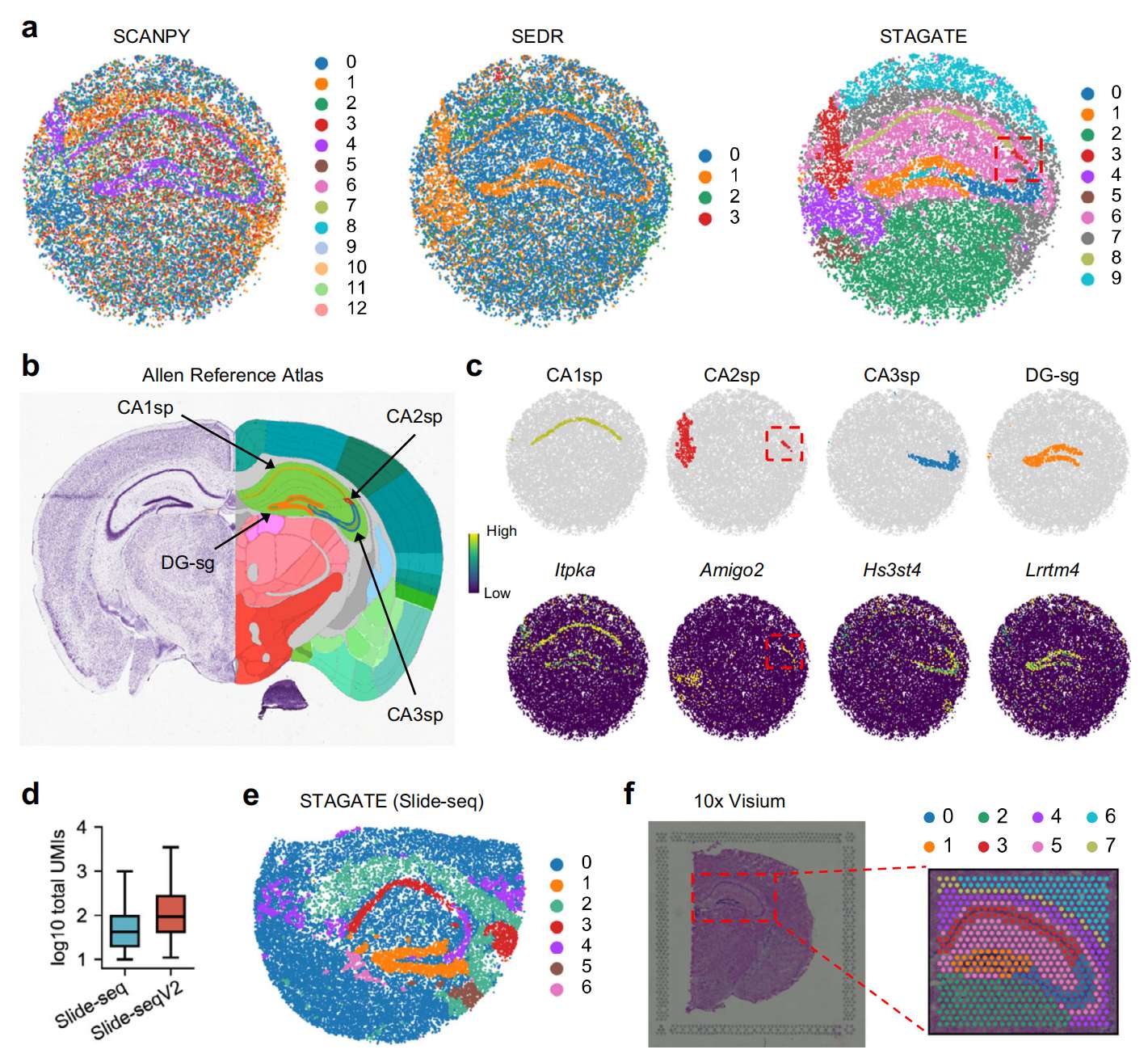
a. 在 Slide-seqV2 海马切片中,在低维 SCANPY、SEDR 和 STAGATE 嵌入上,通过分辨率 = 0.3 的 Louvain 聚类生成的空间域。
b. 来自成年小鼠大脑 Allen 参考图谱的海马结构注释。
c. STAGATE 识别的 CA1sp、CA2sp、CA3sp 和 DG-sg 域以及相应标记基因的可视化。空间域由 Allen 参考图谱中显示的结构注释进行注释。
d. Slide-seq(n = 18,509 spots)和 Slide-seqV2(n = 19,285 spots)分别生成的小鼠海马切片中每个 spot 的总 UMIs 数量。在箱线图中,中心线、箱线界限和须分别表示中位数、上四分位数和下四分位数以及 1.5× 四分位距。
e-f. 分别通过 Slide-seq(e)和 10x Visium(f)技术分析的海马切片上由 STAGATE 生成的空间域。
作者还验证了 STAGATE 在识别小鼠嗅球组织结构方面的性能,嗅球是一种广泛使用的层状组织模型组织。作者首先在 Stereo-seq 从小鼠嗅球组织生成的 ST 数据集上测试了 STAGATE。Stereo-seq 是一种新兴的空间组学技术,可以通过 DNA 纳米球图案阵列芯片实现亚细胞空间分辨率。这里使用的数据被分类为细胞水平的分辨率 (~14μm)。Fu et al. 在 DAPI 染色图像中注释了小鼠冠状嗅球的层状组织,包含头端迁移渠道(RMS)、颗粒细胞层(GCL)、内网状层(IPL)、僧帽细胞层(MCL)、外网状层(EPL)和嗅神经层(ONL)(Fig. 4a)。与 SCANPY 识别的 clusters 相比,使用 STAGATE 和 SEDR 嵌入识别的 clusters 更好地反映了层状组织,并且与注释层很好地对应(Fig. 4b and Supplementary Fig. S7)。重要的是,STAGATE 清楚地识别了狭窄的组织结构 MCL,这通过 MCL 的标记物 GABRA1 的表达得到了验证(Supplementary Fig. S8)。
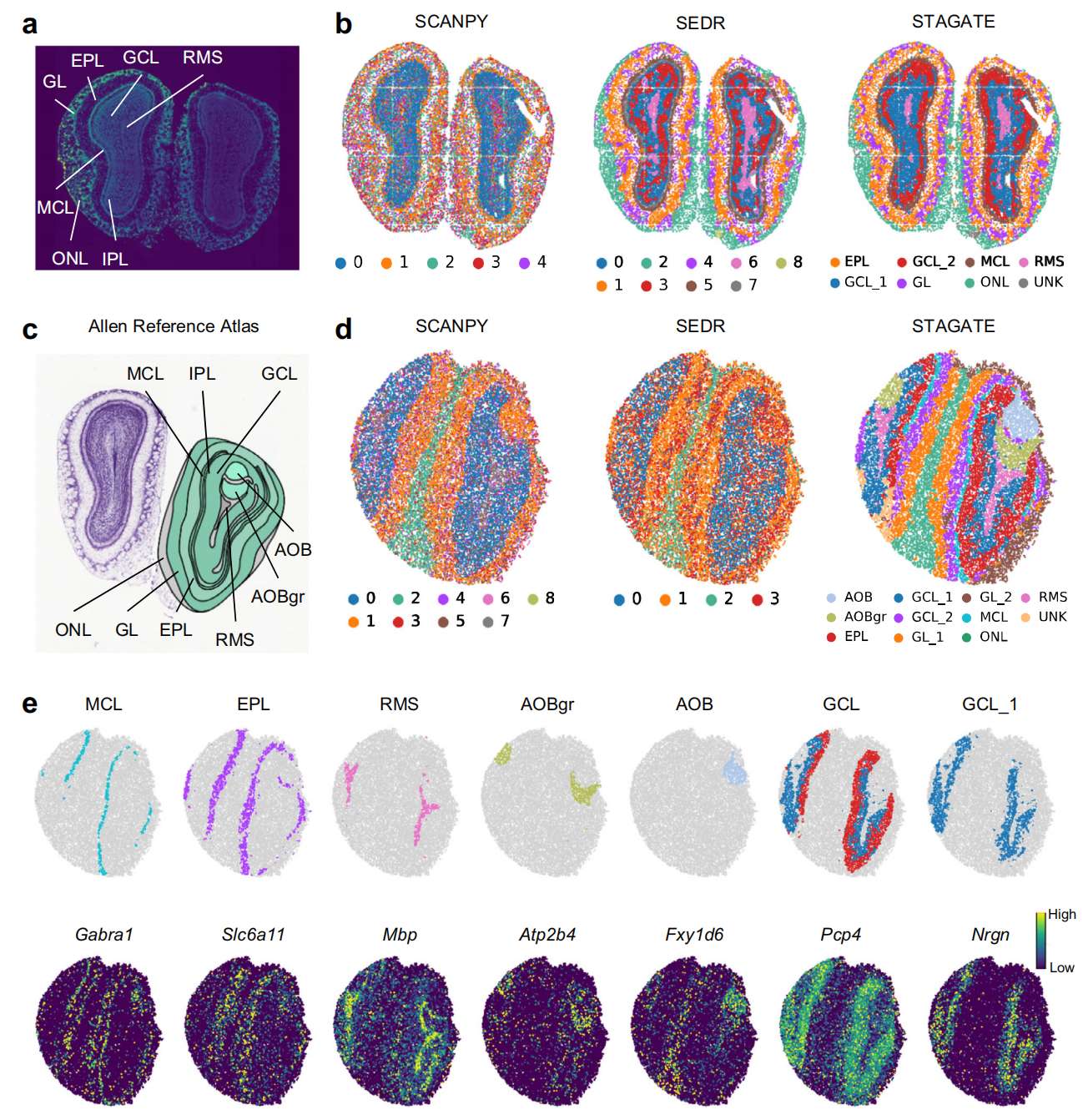
a. 在 Stereo-seq 生成的 DAPI 染色图像中注释的小鼠嗅球层状组织。
b. 在 Stereo-seq 小鼠嗅球组织切片中的低维 SCANPY、SEDR 和 STAGATE 嵌入上,通过分辨率 = 0.8 的 Louvain 聚类生成的空间域。
c. 由 Allen Reference Atlas 注释的小鼠嗅球层状组织。
d. 在 Slide-seqV2 小鼠嗅球组织切片中的低维 SCANPY、SEDR 和 STAGATE 嵌入上,通过分辨率 = 0.5 的 Louvain 聚类生成的空间域。
e. STAGATE 和相应标记基因识别的空间域的可视化。空间域由 Allen Reference Atlas 中显示的层状组织注释。
作者还将 STAGATE 应用于 Slide-seqV2 描绘的小鼠嗅球切片,发现 STAGATE 识别的空间域与 Allen Reference Atlas 中小鼠冠状嗅球的注释高度一致(Fig. 4c)。具体来说,与 SCANPY 和 SEDR 生成的 clusters 相比,STAGATE 识别出两个分别对应于副嗅球 (AOB) 和副嗅球颗粒层 (AOBgr) 的空间域(Fig. 4d and Supplementary Fig. S9)。STAGATE 发现的这些空间域明显得到已知基因标记的支持(Fig. 4e)。例如,Fxyd6 在识别的 AOB 域上表现出强烈表达,这与其免疫组织化学实验一致。颗粒细胞标记 Atp2b4 在识别的 AOBgr 域上表现出强烈表达。STAGATE 还鉴定出以 MCL 标志物 Gabra1 为主要表达的狭窄 MCL 结构。此外,STAGATE 还鉴定出 GCL 的空间亚群,名为 GCL_1,其主要表达为 Nrgn。Nrgn 是一种有据可查的精神分裂症风险基因,这意味着该域与认知功能有关。此外,作者发现 STAGATE 在 UMAP 图和 PAGA 图中描绘了小鼠嗅球之间的空间轨迹(从 AOBgr 到 RMS 再到 ONL)(Supplementary Fig. S10)。总之,这些结果说明了 STAGATE 能够识别组织结构并从不同空间分辨率的 ST 数据中揭示其组织。
4. 注意力机制和细胞类型感知模块有助于更好地表征相邻点之间的相似性
接下来,作者测试了 STAGATE 是否可以提供对包括更复杂的生物组织(例如整个大脑)在内的切片的洞察。作者将 STAGATE 应用于 10x Visium 数据集,该数据集描绘了小鼠冠状脑切片的空间表达(Fig. 5a)。作者发现 SCANPY 识别的聚类结果粗略地划分了包含不同细胞类型的组织结构,而缺乏对小空间域的识别(Fig. 5b and Supplementary Fig. S11)。例如,SCANPY 的聚类分配无法识别海马内的“索状”结构——阿蒙角和“箭状”结构——齿状回。此外,SEDR 仅平滑了域边界,但也不能描绘小的空间域(Fig. 5b)。直接应用 STAGATE 在空间域识别方面带来了一些改进(Fig. 5b)。具体来说,在海马区域,没有细胞类型感知模块的 STAGATE 识别出了阿蒙角的 CA1 区(domain 7)和 CA3 区(domain 8),但并没有描绘出齿状回结构。
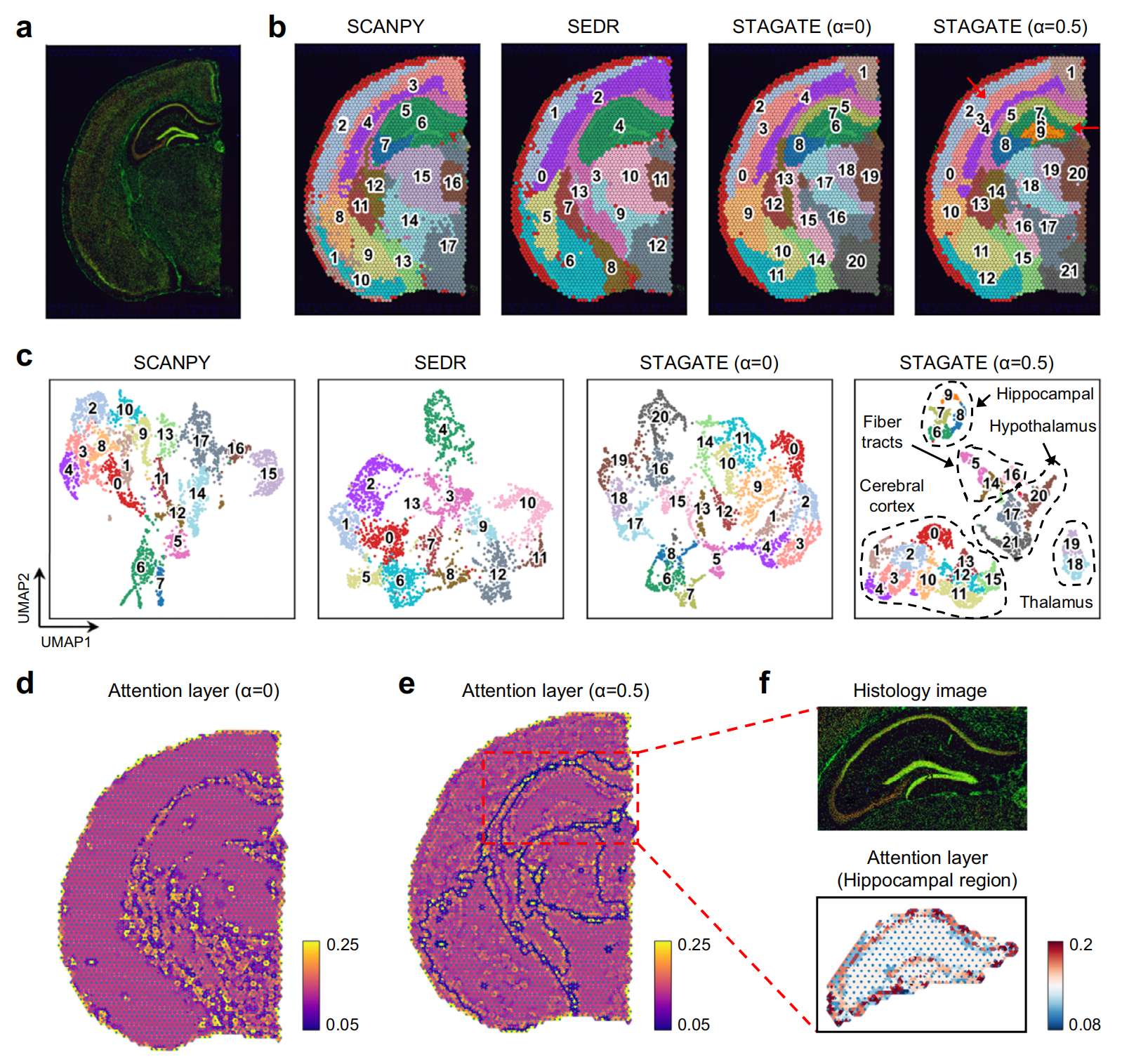
a. 用 DAPI 和 Anti-NeuN 染色的组织切片的免疫荧光成像。
b. 使用细胞类型感知模块,在 SCANPY、SEDR、STAGATE 和 STAGATE 的低维嵌入上通过分辨率 = 1 的 Louvain 聚类生成的空间域。α 表示细胞类型感知 SNN 的权重(见 Fig. 1)。
c. 分别使用细胞类型感知模块对 SCANPY、SEDR、STAGATE 和 STAGATE 的低维嵌入进行 UMAP 可视化。
d-e. 不使用(d)或使用(e)细胞类型感知模块的 STAGATE 注意力层的可视化。注意力层的节点根据 spots 的空间位置排列。注意力层的边缘用相应的权重着色。
f. 海马区域免疫荧光成像的放大视图和 e 中注意力层的可视化。
对于包含异质细胞类型且空间分辨率较低的 ST 数据,带有细胞类型感知模块的 STAGATE 可以更好地学习空间相似性(Fig. 1)。具体而言,预聚类过程基于具有较小分辨率参数(默认设置为 0.2)的 Louvain 算法(Supplementary Fig. S11b)。正如预期的那样,细胞类型感知模块的使用有助于识别空间域(Fig. 5b)。STAGATE 识别了海马中的 Ammon 角以及齿状回结构,并进一步描绘了 Ammon 角的空间域 CA1(domain 7)和 CA3(domain 8)。此外,STAGATE 更好地描绘了皮质区域的层结构(domain 2, 3, and 4)。值得注意的是,作者发现细胞类型感知模块也显著改善了 UMAP 图中组织结构的分离,而没有细胞类型感知模块的 SEDR 和 STAGATE 的分离更像是非空间方法 SCANPY 的平滑版本(Fig. 5c)。
作者进一步评估了注意机制的使用是否确实有助于更好地表征相邻 spots 间的异质相似性。作者通过按空间位置排列节点、按权重对边缘着色来可视化注意层,发现单独使用注意机制可以勾勒出皮质、海马、中脑等主要组织结构的边界(Fig. 5d)。将注意机制与细胞类型感知模块相结合,可以增强结构边界的描绘,进一步揭示小空间域内的空间相似性(Fig. 5e)。例如在海马区域,STAGATE 自适应地学习了阿蒙角和齿状回结构内的空间相似性(Fig. 5f)。总之,这些结果表明了注意机制和细胞类型感知模块对于描绘相邻 spots 间相似性的重要性。
作者还证实,使用细胞类型感知模块可以改善来自小鼠后脑的另一个 10x Visium 数据集中小空间域的识别(Supplementary Fig. S12)。具体而言,STAGATE 清楚地描绘了冠状结构周围的薄层,这通过 Calb1 的更高表达得到证实(Supplementary Fig. S12c)。相比之下,没有细胞类型感知模块的 STAGATE 和 SEDR 将更多 spots 归入该域。在 domains 2、12 和 15 中也发现了类似的情况,其中 STAGATE 是唯一可以清楚区分与组织学图像相对应的这三个域的方法(Supplementary Fig. S12d, e)。此外,使用细胞类型感知模块还有助于识别皮质区域内的层结构(Supplementary Fig. S12f)。通过可视化注意层,作者发现注意机制描绘了组织结构之间的异质空间相似性(Supplementary Fig. S12g)。
5. STAGATE 对基因表达进行去噪,以更好地表征空间表达模式
STAGATE 可以对基因表达进行去噪和估算。作者采用 STAGATE 来降低 DLPFC 数据集中的噪音,以更好地显示基因的空间模式。作者将原始数据的六个层标记基因的表达与 DLPFC 切片 151676 中 STAGATE 去噪后的基因表达进行了比较(Fig. 6a)。正如预期的那样,STAGATE 去噪后的基因清楚地显示了这些层标记基因的层状富集。例如,去噪后,ATP2B4 基因在第 2 层和第 6 层显示出差异表达,这与之前报道的结果一致,而其原始空间表达则完全混乱。作者根据来自 Allen Human Brain Atlas 的公开原位杂交 (ISH) 数据验证了 STAGATE 显示的层状富集(Fig. 6b)。此外,使用小提琴图比较原始表达和 STAGATE 去噪后的表达,表明 STAGATE 增强了层标记基因的空间模式(Fig. 6c, d)。值得注意的是,STAGATE 在 DLPFC 切片 151507 上获得了类似的性能(Supplementary Fig. S13)。总的来说,这些结果证明了 STAGATE 降低噪音和增强空间表达模式的能力。此外,作者还在下采样实验中比较了 STAGATE 与四种广泛使用的单细胞 RNA-seq 填充算法的填充性能,并显示其在填充效率和空间表达模式保存方面均更胜一筹(参见 Supplementary Information 的“STAGATE 在保留空间表达模式的同时填充基因表达”小节;Supplementary Fig. S14)。
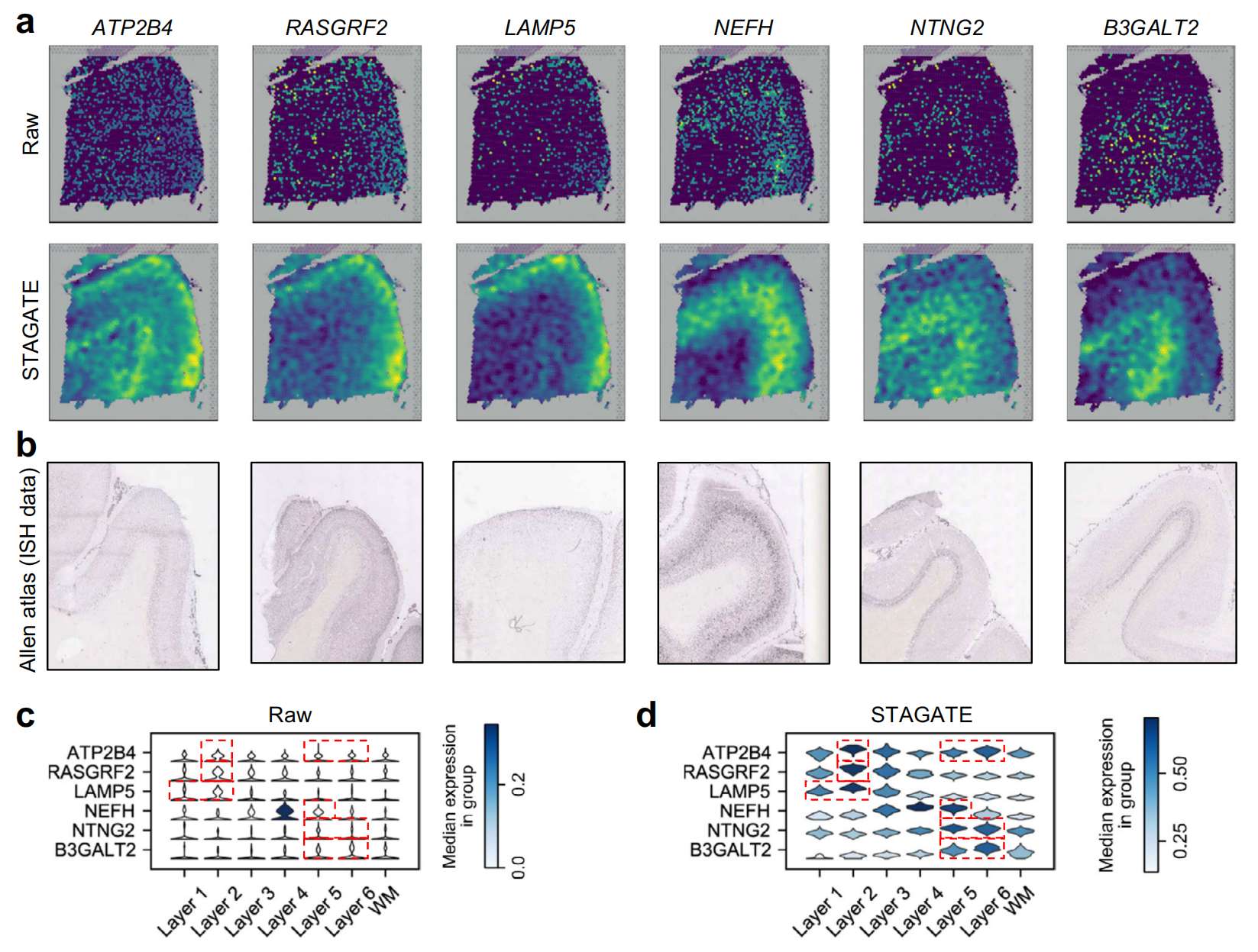
a. DLPFC 切片 151676 中的六个层标记基因的原始空间表达和 STAGATE 去噪表达的可视化。
b. 来自 Allen Human Brain Atlas 的成人大脑视觉皮层(ATP2B4、RASGRF2、NEFH、NTNG2 和 B3GALT2)或颞叶皮层(LAMP5)的 ISH 图像。
c. 层标记基因原始表达的小提琴图。
d. 层标记基因 STAGATE 去噪表达的小提琴图。与层标记基因对应的皮质层用红色框标记。
6. 3D SNN 的使用可以更好地提取 3D 空间模式
作者将 STAGATE 应用于 pseudo-3D ST 数据,该数据通过对齐由 Slide-seq 描绘的七个海马切片中的“索状”结构的 spots 而构建(Fig. 7a; Supplementary Table S3)。作者通过同时考虑每个切片内的 2D SNN 和相邻切片之间的相邻 spots,扩展了 STAGATE 以进行 3D 空间域识别(Fig. 7b;参见 Methods 中的“使用 STAGATE 识别 3D 空间域”小节)。由于数据稀疏,使用 SCANPY 生成的聚类结果混杂(Supplementary Fig. S15)。当仅采用 2D SNN 时,由于切片之间的批次效应,STAGATE 无法识别 CA2sp 域(Fig. 7c, d)。在相邻切片之间添加相邻边后,STAGATE 清晰地描绘了已知的组织结构,并且 spots 倾向于通过其空间结构而不是 UMAP 图中切片 ID 聚类(Fig. 7e, f)。作者利用已知的标记基因(包括 Itpka、Bcl11b、Amigo2 和 Lrrtm4)验证了基于 STAGATE 识别的组织结构(Supplementary Fig. S15d)。这些结果表明 STAGATE 可以帮助重建 3D 组织模型,并通过结合 3D 空间信息准确提取 3D 表达模式。
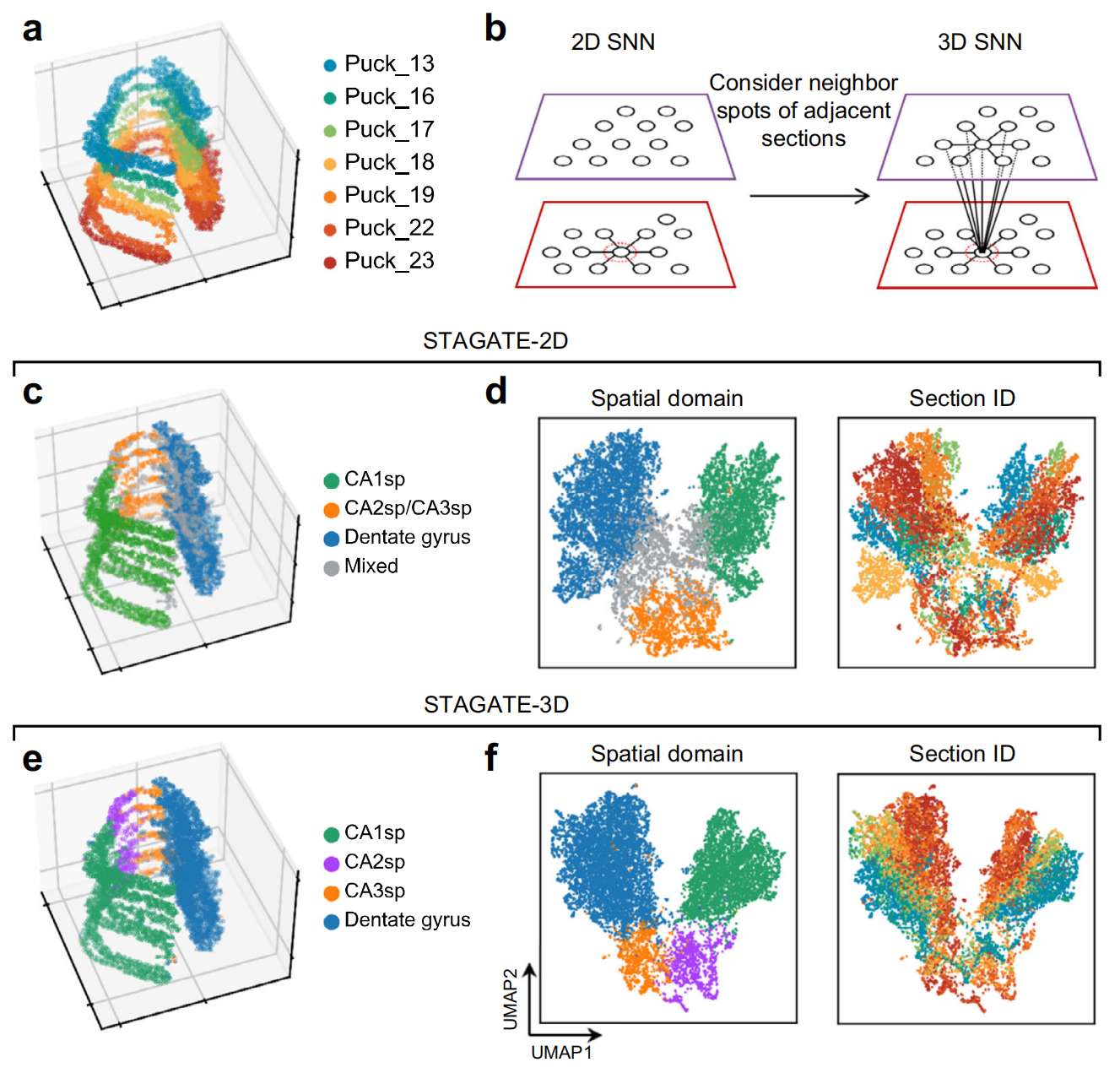
a. 由 Slide-seq 描绘的七个连续对齐切片堆叠而成的 3D 海马体积可视化。
b. 3D SNN 是每个切片内的 2D SNN 与连续切片之间的空间网络的组合。
c. STAGATE-2D 使用 2D SNN 生成的 cluster 分配。
d. STAGATE-2D 嵌入生成的 UMAP 图。这些 spots 分别由已识别的空间域(左)和切片 ID(右)着色。
e. STAGATE-3D 使用 3D SNN 生成的 cluster 分配。
f. STAGATE-3D 嵌入生成的 UMAP 图。这些 spots 分别由已识别的空间域(左)和切片 ID(右)着色。
讨论
准确识别空间域并进一步提取空间表达基因对于理解组织组织和生物功能至关重要。在这里,作者开发了一种快速且用户友好的空间域识别方法 STAGATE,该方法可以通过将 SCANPY 包的“anndata”对象作为输入无缝集成到标准分析工作流程中。STAGATE 将空间位置信息转换为 SNN,并进一步采用图注意自动编码器来整合 SNN 和表达谱。作者在由不同平台以不同空间分辨率生成的各种 ST 数据上测试了 STAGATE 的性能。作者发现 STAGATE 准确地揭示了 DLPFC 和小鼠嗅球的层状组织。此外,STAGATE 清楚地识别了已知的海马组织结构并揭示了它的空间域。作者还通过将 STAGATE 与 ISH 图像进行比较来展示其在表达去噪方面的能力。最后,作者说明了 STAGATE 缓解连续切片之间的批次效应并在 pseudo-3D ST 模型中提取 3D 表达域的能力。
STAGATE 的成功主要归功于使用图注意力机制来考虑空间邻居信息。然而,目前的 STAGATE 专注于表达谱和空间信息的整合,并没有利用组织学图像。现有的以组织学图像为输入的方法,如 stLearn,在作者的比较中没有取得良好的表现。stLearn 使用预训练的神经网络从图像中提取特征,并进一步通过余弦距离计算形态距离。作者认为这种预定义的方法没有利用深度学习的灵活性,注意力机制可以扩展以方便地自适应地整合组织学图像特征。
在本研究中,作者主要关注基于测序的 ST 数据,这些数据未在单细胞分辨率下进行表征。作者进一步将 STAGATE 应用于由 STARMAP 技术生成的基于图像的单细胞分辨率 ST 数据集,该数据集包括 1207 个细胞上 1020 个基因的表达(Supplementary Fig. S16)。以专家注释结构为金标准,与其他五种方法相比,STAGATE 实现了最佳聚类准确率(ARI = 0.544),而 SpaGCN 排名第二(ARI = 0.484)。此外,鉴于为基于图像的 ST 数据设计的空间域识别和单细胞分割(例如 ClusterMap 和 Baysor)之间的联系,作者预计 STAGATE 的理念可以在不久的将来扩展到正在进行的亚细胞分辨率技术(例如 Stereo-seq 和 PIXEL-seq)的单细胞分割任务。作者还希望通过使用新技术生成的数据集来提高其适用性。
STAGATE 可以处理不同空间分辨率的 ST 数据。一般来说,由于相邻 spots 之间的高度相似性,STAGATE 对于细胞或亚细胞分辨率的 ST 数据表现更好。对于空间分辨率相对较低的技术,作者引入了细胞类型感知模块来描述异构空间相似性。然而,STAGATE 的一个潜在限制是,它将一个部分的相邻 spots 与属于不同部分的相邻 spots 视为相同。未来的工作可能会采用异构网络来更好地描绘 3D 组织模型。
随着空间分辨率和数据规模的增加,计算方法应满足效率和可扩展性的基本要求。作者记录了 STAGATE 在真实数据集上花费的运行时间(Supplementary Fig. S17a)。在处理包含超过 50k spots 的最大真实数据集时,STAGATE 仅花费约 40 分钟。作者还在不同规模的模拟数据集上对 STAGATE 的运行时间和内存使用情况进行了基准测试,其中 spots 按照 10x Visium 芯片的位置排列。数值实验表明,STAGATE 速度很快,处理包含 50k spots 的数据集仅花费不到 40 分钟,GPU 内存使用量约为 4GB(Supplementary Fig. S17b)。然而,GPU 内存使用量与 spots 数几乎呈线性相关,这可能是限制 STAGATE 应用于海量数据集的瓶颈(Supplementary Fig. S17c)。未来的工作有望通过引入基于子图的训练策略来提高 STAGATE 的可扩展性。
此外,STAGATE 能够在空间域内检测空间变异基因。现有的空间变异基因识别算法(如 SPARK-X)不考虑空间域信息,这使得很难识别小组织结构内空间特异性表达的基因。为了说明这一点,作者比较了小鼠嗅球组织的 Slide-seqV2 数据集上 STAGATE 空间域的差异表达基因与 SPARK-X 的差异表达基因。具体来说,STAGATE 识别了 959 个域特异性基因,而 SPARK-X 搜索了 2479 个空间变异基因,FDR < 0.01。作者发现 SPARK-X 识别的许多基因在空间域之间没有表现出显著差异(Supplementary Fig. S18a)。此外,STAGATE 识别的基因组和 SPARK-X 的前 1,000 个基因之间用 Moran's I 统计量测量的空间自相关性相似(Supplementary Fig. S18b)。两种方法鉴定出的基因集具有很大的重叠性,但 SPARK-X 忽略了一些小组织结构的特定基因(Supplementary Fig. S18c)。例如,MCL 标记物 Gabra1 在 MCL 结构域中表现出显著的富集(Fig. 4e; FDR = 1e-34),但 SPARK-X 未能鉴定出其空间模式(FDR = 0.018)。此外,Nefh 基因也在 MCL 结构域中表现出强烈的表达(Supplementary Fig. S18d; FDR = 1e-12),而 SPARK-X 忽略了它(FDR = 1)。作者期望 STAGATE 能够促进组织组织的识别和相应基因标记的发现。
注:本文为个人学习笔记,仅供大家参考学习,不得用于任何商业目的。如有侵权,请联系作者删除。

本文由 mdnice 多平台发布



















