目录
数据读入
每个样本各细胞比例
两个组间细胞比例
亚组间细胞比例差异分析(循环)
单个细胞类型亚新间比例差异
①单细胞学习-数据读取、降维和分群-CSDN博客
比较各个样本间的各类细胞比例或者亚组之间的细胞比例差异
①数据读入
#各样本细胞比例计算
rm(list = ls())
library(Seurat)
load("scedata1.RData")#这里是经过质控和降维后的单细胞数据
table(scedata$orig.ident)#查看各组细胞数
table(Idents(scedata))#查看各种类型细胞数目
#prop.table(table(Idents(scedata)))
table(Idents(scedata), scedata$orig.ident)#每个样本不同类型细胞数据> table(scedata$orig.ident)#查看各组细胞数BM1 BM2 BM3 GM1 GM2 GM3 2754 747 2158 1754 1528 1983
> table(Idents(scedata))#查看各种类型细胞数目Fibroblast Endothelial Immune Other Epithelial 2475 4321 2688 766 674 > #prop.table(table(Idents(scedata))) > table(Idents(scedata), scedata$orig.ident)#每个样本不同类型细胞数据BM1 BM2 BM3 GM1 GM2 GM3Fibroblast 571 135 520 651 312 286Endothelial 752 244 619 716 906 1084Immune 1220 145 539 270 149 365Other 142 161 194 55 79 135Epithelial 69 62 286 62 82 113
②每个样本各细胞比例
#换算每样样本每种细胞占有的比例:绘制总的堆积图
Cellratio <- prop.table(table(Idents(scedata),scedata$orig.ident),margin = 2)# margin = 2按照列计算每个样本比例
Cellratio <- as.data.frame(Cellratio)#计算比例绘制堆积图
library(ggplot2)#绘制细胞比例堆积图
colourCount = length(unique(Cellratio$Var1))
p1 <- ggplot(Cellratio) + geom_bar(aes(x =Var2, y= Freq, fill = Var1),stat = "identity",width = 0.7,size = 0.5,colour = '#222222')+ theme_classic() +labs(x='Sample',y = 'Ratio')+#coord_flip()+ #进行翻转theme(panel.border = element_rect(fill=NA,color="black", size=0.5, linetype="solid"))
p1
dev.off()> head(Cellratio)Var1 Var2 Freq 1 Fibroblast BM1 0.20733479 2 Endothelial BM1 0.27305737 3 Immune BM1 0.44299201 4 Other BM1 0.05156137 5 Epithelial BM1 0.02505447 6 Fibroblast BM2 0.18072289

③两个组间细胞比例
这里比较BM和GM两个组间的细胞比例
##分成两个组进行比较:先查看每个样本的具体细胞数量
library(tidyverse)
library(reshape)
clusdata <- as.data.frame(table(Idents(scedata), scedata$orig.ident))
#进行长宽数据转换
clusdata1 <- clusdata %>% pivot_wider(names_from = Var2,values_from =Freq )
clusdata1 <- as.data.frame(clusdata1)
rownames(clusdata1) <- clusdata1$Var1
clusdata2 <- clusdata1[,-1]#[1] "BM1" "BM2" "BM3" "GM1" "GM2" "GM3"
#分别计算每个组每种细胞和
BM <- c("BM1","BM2","BM3")
clusdata2$BMsum <- rowSums(clusdata2[,BM])
GM <- c("GM1","GM2","GM3")
clusdata2$GMsum <- rowSums(clusdata2[,GM])#然后绘制堆积图
clus2 <- clusdata2[,c(7,8)]
clus2$ID <- rownames(clus2)
clus3 <- melt(clus2, id.vars = c("ID"))##根据分组变为长数据
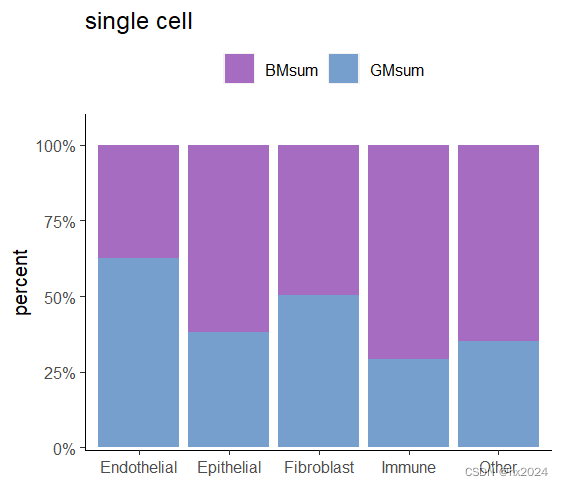
p <- ggplot(data = clus3,aes(x=ID,y=value,fill=variable))+#geom_bar(stat = "identity",position = "stack")+ ##展示原来数值geom_bar(stat = "identity",position = "fill")+ ##按照比例展示:纵坐标为1scale_y_continuous(expand = expansion(mult=c(0.01,0.1)),##展示纵坐标百分比数值labels = scales::percent_format())+scale_fill_manual(values = c("BMsum"="#a56cc1","GMsum"="#769fcd"), ##配色:"BMsum"="#98d09d","GMsum"="#e77381"limits=c("BMsum","GMsum"))+ ##limit调整图例顺序theme(panel.background = element_blank(), ##主题设置axis.line = element_line(), legend.position = "top")+ #"bottom"labs(title = "single cell",x=NULL,y="percent")+ ##X,Y轴设置guides(fill=guide_legend(title = NULL,nrow = 1,byrow = FALSE))
p
dev.off()> head(clus3)ID variable value 1 Fibroblast BMsum 1226 2 Endothelial BMsum 1615 3 Immune BMsum 1904 4 Other BMsum 497 5 Epithelial BMsum 417 6 Fibroblast GMsum 1249
④亚组间细胞比例差异分析(循环)
#组间差异分析:仍然是使用这个比例数据进行分析,不过却是在各个样本中进行比例比较
table(scedata$orig.ident)#查看各组细胞数
table(Idents(scedata))#查看各种类型细胞数目
table(Idents(scedata), scedata$orig.ident)#各组不同细胞群细胞数
Cellratio <- prop.table(table(Idents(scedata), scedata$orig.ident), margin = 2)#计算各组样本不同细胞群比例
Cellratio <- data.frame(Cellratio)
#需要进行数据转换,计算每个样本比例后进行差异分析
library(reshape2)
cellper <- dcast(Cellratio,Var2~Var1, value.var = "Freq")
rownames(cellper) <- cellper[,1]
cellper <- cellper[,-1]
###添加分组信息dataframe
sample <- c("BM1","BM2","BM3","GM1","GM2","GM3")
group <- c("BM","BM","BM","GM","GM","GM")
samples <- data.frame(sample, group)#创建数据框
rownames(samples)=samples$sample
cellper$sample <- samples[rownames(cellper),'sample']#R添加列
cellper$group <- samples[rownames(cellper),'group']#R添加列###作图展示
pplist = list()##循环作图建立空表
library(ggplot2)
library(dplyr)
library(ggpubr)
library(cowplot)
sce_groups = c('Endothelial','Fibroblast','Immune','Epithelial','Other')
for(group_ in sce_groups){cellper_ = cellper %>% select(one_of(c('sample','group',group_)))#选择一组数据colnames(cellper_) = c('sample','group','percent')#对选择数据列命名cellper_$percent = as.numeric(cellper_$percent)#数值型数据cellper_ <- cellper_ %>% group_by(group) %>% mutate(upper = quantile(percent, 0.75), lower = quantile(percent, 0.25),mean = mean(percent),median = median(percent))#上下分位数print(group_)print(cellper_$median)pp1 = ggplot(cellper_,aes(x=group,y=percent)) + #ggplot作图geom_jitter(shape = 21,aes(fill=group),width = 0.25) + stat_summary(fun=mean, geom="point", color="grey60") +#stat_summary添加平均值theme_cowplot() +theme(axis.text = element_text(size = 10),axis.title = element_text(size = 10),legend.text = element_text(size = 10),legend.title = element_text(size = 10),plot.title = element_text(size = 10,face = 'plain'),legend.position = 'none') + labs(title = group_,y='Percentage') +geom_errorbar(aes(ymin = lower, ymax = upper),col = "grey60",width = 1)###组间t检验分析labely = max(cellper_$percent)compare_means(percent ~ group, data = cellper_)my_comparisons <- list( c("GM", "BM") )pp1 = pp1 + stat_compare_means(comparisons = my_comparisons,size = 3,method = "t.test")pplist[[group_]] = pp1
}#批量绘制
plot_grid(pplist[['Endothelial']],pplist[['Fibroblast']],pplist[['Immune']],pplist[['Epithelial']],pplist[['Other']],#nrow = 5,#列数ncol = 5)#行数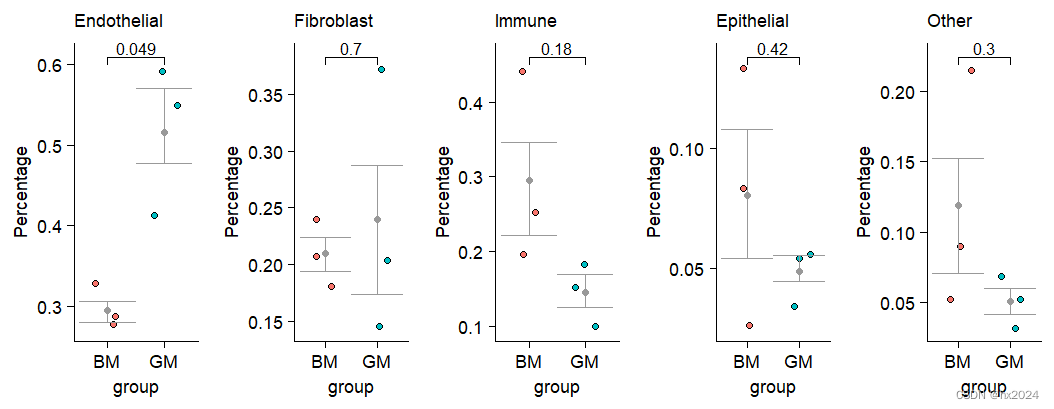
⑤单个细胞类型亚新间比例差异
##数据处理
##单个细胞类型比例计算
rm(list = ls())
library(Seurat)
library(tidyverse)
library(reshape2)
library(ggplot2)
library(dplyr)
library(ggpubr)
library(cowplot)
load("scedata1.RData")#计算各个样本细胞,各种类型细胞
Cellratio <- prop.table(table(Idents(scedata), scedata$orig.ident), margin = 2)#计算样本比例
Cellratio <- data.frame(Cellratio)
cellper <- dcast(Cellratio,Var2~Var1, value.var = "Freq")##长数据转宽数据
rownames(cellper) <- cellper[,1]
cellper <- cellper[,-1]
sample <- c("BM1","BM2","BM3","GM1","GM2","GM3")###添加分组信息dataframe
group <- c("BM","BM","BM","GM","GM","GM")
samples <- data.frame(sample, group)#创建数据框
rownames(samples)=samples$sample
cellper$sample <- samples[rownames(cellper),'sample']#R添加列
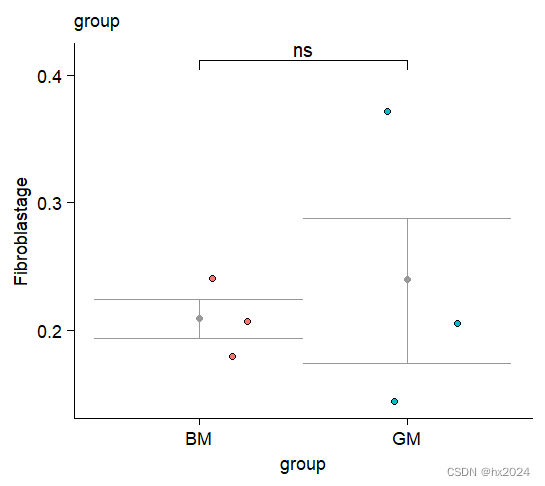
cellper$group <- samples[rownames(cellper),'group']#R添加列dat <- cellper[,c(1,7)]#提取需要分析的细胞类型"Fibroblast" "group"
#根据分组计算四分位及中位数
dat1 <- dat %>% group_by(group) %>% mutate(upper = quantile(Fibroblast, 0.75), lower = quantile(Fibroblast, 0.25),mean = mean(Fibroblast),median = median(Fibroblast))
#table(dat1$group)#BM GM 作图
#pdf("单个细胞类型组间比较.pdf",width = 4,height = 4)##一定添加大小
my_comparisons =list( c("BM","GM"))
P <- ggplot(dat1,aes(x=group,y= Fibroblast)) + #ggplot作图geom_jitter(shape = 21,aes(fill=group),width = 0.25) + stat_summary(fun=mean, geom="point", color="grey60") +theme_cowplot() +theme(axis.text = element_text(size = 10),axis.title = element_text(size = 10),legend.text = element_text(size = 10),legend.title = element_text(size = 10),plot.title = element_text(size = 10,face = 'plain'),legend.position = 'none') + labs(title = "group",y='Fibroblastage') +geom_errorbar(aes(ymin = lower, ymax = upper),col = "grey60",width = 1)+#误差棒#差异检验stat_compare_means(comparisons=my_comparisons,label.y = c(0.4),method="t.test",#wilcox.testlabel="p.signif")
P
dev.off()
感谢: TS的美梦-CSDN博客
参考:跟着Cell学单细胞转录组分析(六):细胞比例计算及可视化 (qq.com)
跟着Cell学单细胞转录组分析(十四):细胞比例柱状图---连线堆叠柱状图_单细胞细胞占比图怎么画-CSDN博客