小罗碎碎念
有关于生存曲线的基本概念(例如删失事件的定义)和绘图的详细教程我已经在5月的推文中介绍过了,有需求的同学欢迎前去考古。
R语言绘制生存分析曲线·从概念到实战的保姆级教程|2024-05-12
https://mp.weixin.qq.com/s/Z6gBbpBlALT_KKgEW29Stg
今天这期推文的目的是把主要的代码框架梳理出来,方便快速调用,省去浏览知识点分析的过程。
成品展示
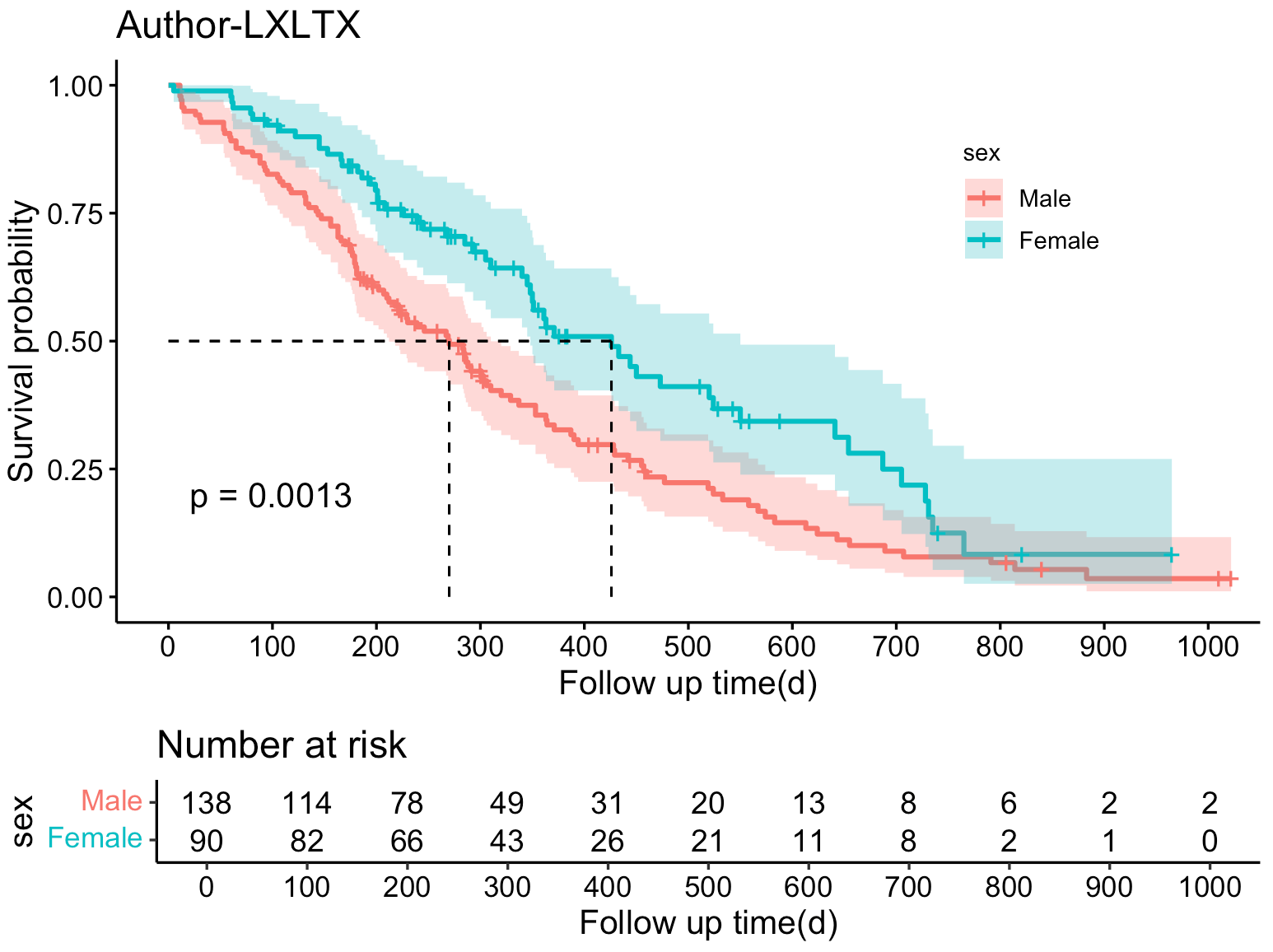
跟着这期推文,你最后能实现的效果如下。
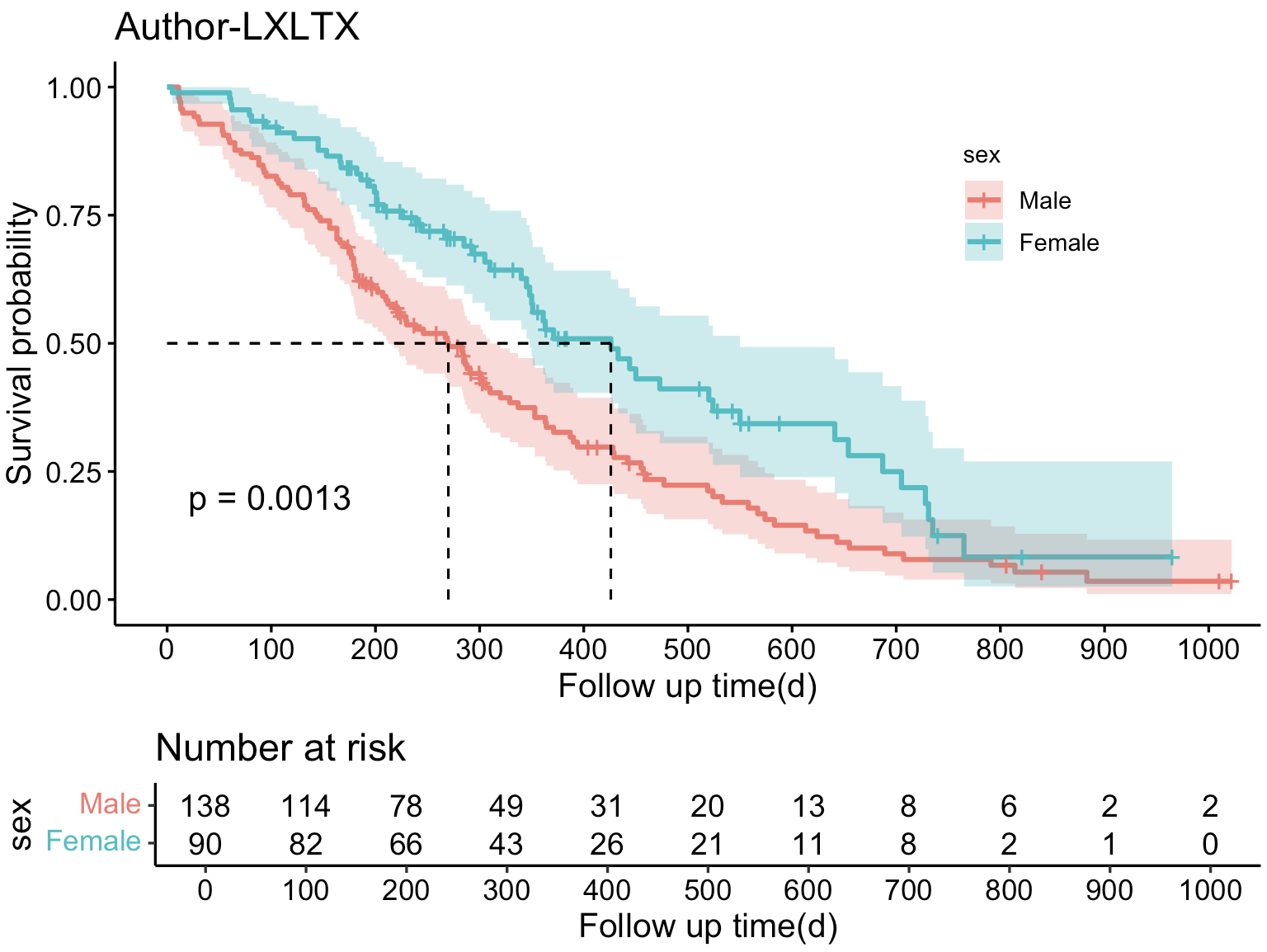
一、曲线解读
假设我们已经掌握了一定的基础知识,那么可以看下列这个曲线。
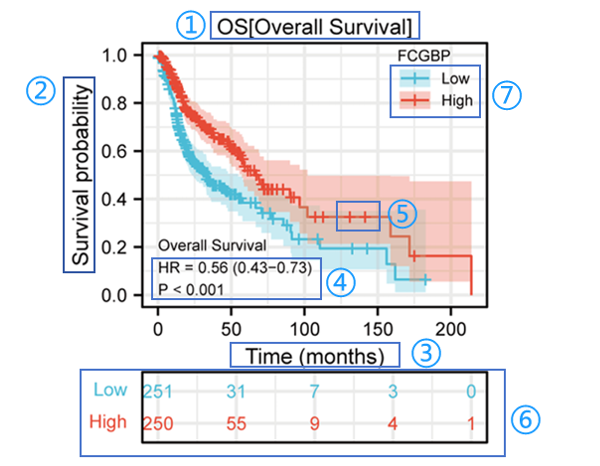
①OS:总生存期
②纵坐标:患者生存率
③横坐标:观察时间
④HR:目标gene高表达组相对于低表达组的死亡风险比;HR<1,代表gene高表达抑制死亡;P<0.05说明两组间差异有统计学意义
⑤代表删失数据
⑥两条曲线在各个时间点上对应的人数
⑦组别:代表不同组别对应曲线
二、曲线绘制
先调用自带的数据做一些分析,然后我再介绍如何利用自己的数据进行分析。
2-1:装包
install.packages("survminer") # 安装survminer包
install.packages("survival") # 安装survival包
library(survminer) # 加载包
library(survival) # 加载包
2-2:导入内置数据集
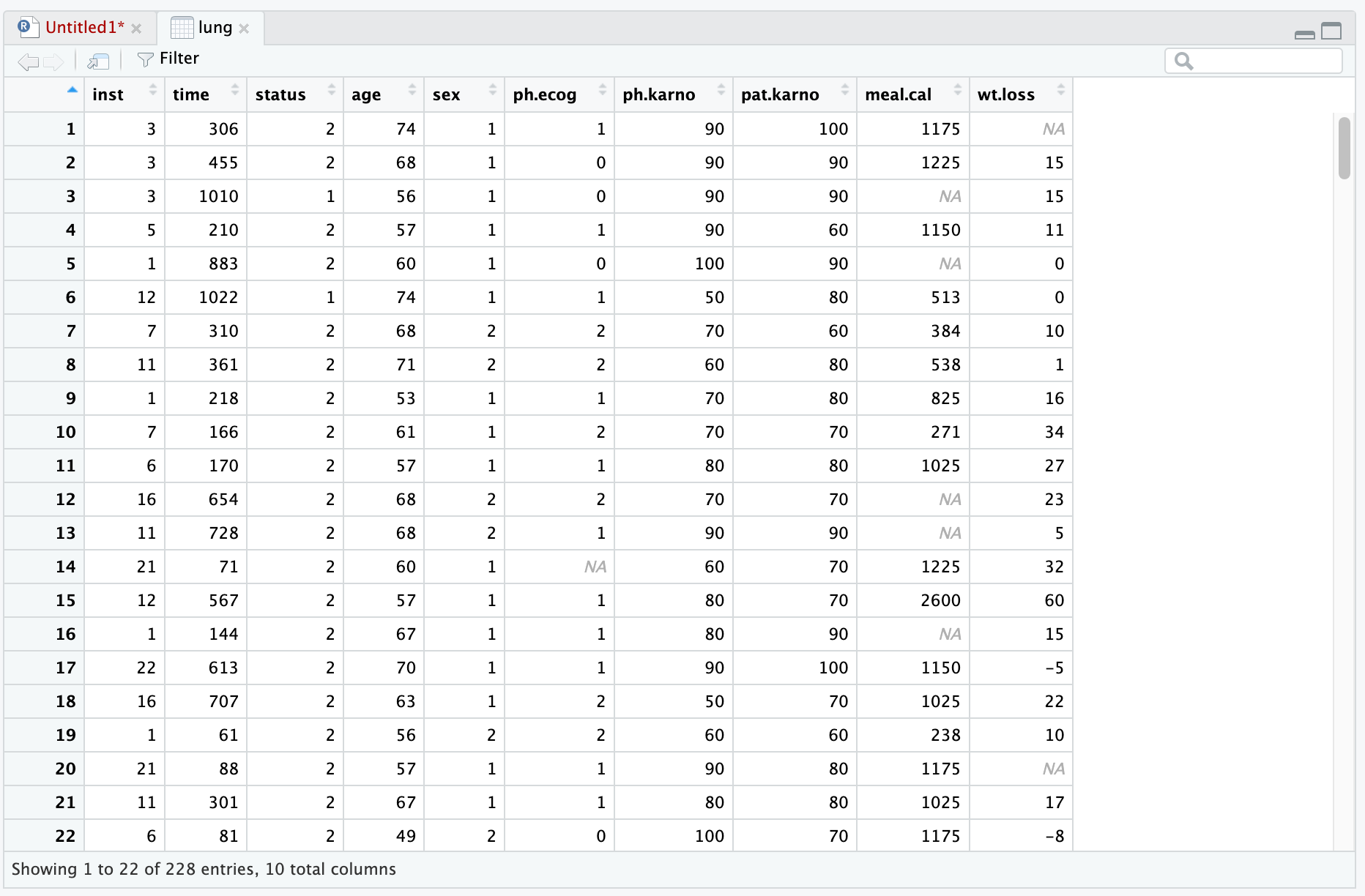
使用survival包的lung数据集进行演示。
View(lung) # 查看数据集
-
inst # 机构代码;
-
time # 生存天数;
-
status # 生存状态;
-
age # 年龄;
-
sex # 性别,1为男性,2为女性;
-
ph.ecog、ph.karno、pat.karno # 为病人和患者评分,这里用不到;
-
meal.cal # 进食时消耗的卡路里;
-
wt.loss # 最近6个月内的体重下降。
2-3:拟合生存曲线
创建生存对象
在survival包中先使用Surv()函数创建生存对象,生存对象是将事件时间和删失信息合并在一起的数据结构。
attach(lung) # 绑定数据集
Surv(time,status) # 创建生存对象
这个生存对象通常用于后续的生存分析模型中,如Cox比例风险模型等,以估计生存函数、风险比或者进行时间至事件的分析。
拟合曲线
在R语言中,生存分析通常涉及使用survival包中的survfit()函数来拟合生存数据。
fit <- survfit(Surv(time,status) ~ sex,data = lung) # 数据集来源
fit # 查看拟合曲线信息
plot(fit)#绘制曲线
现在其实就可以绘制一个非常粗糙的曲线了,后续会介绍如何完善。
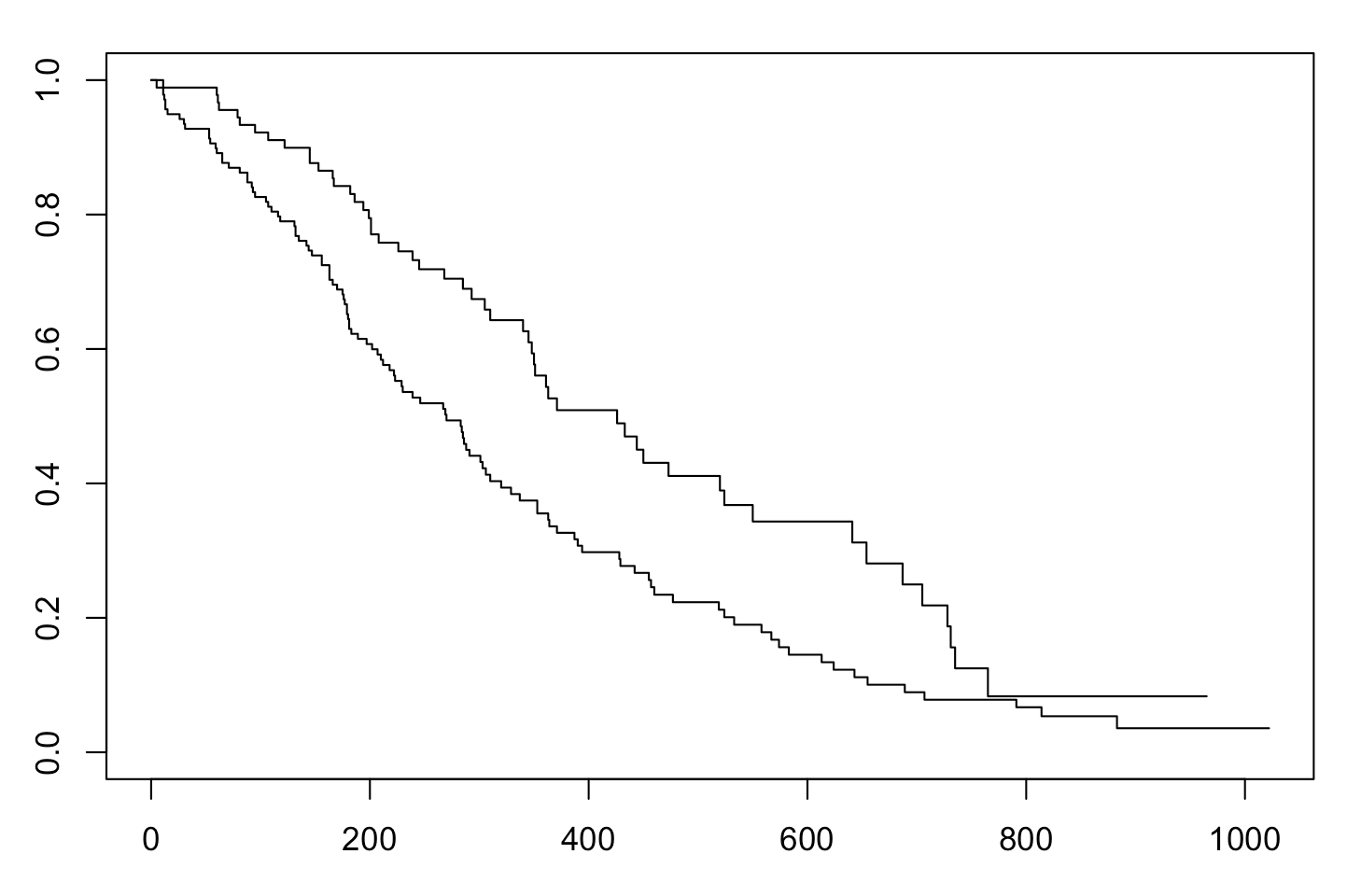
绘制基础曲线
在R语言中,ggsurvplot() 函数是 survminer 包提供的一个用于绘制生存曲线的高级函数,它基于 ggplot2 包的图形语法。
ggsurvplot(fit, data = lung)
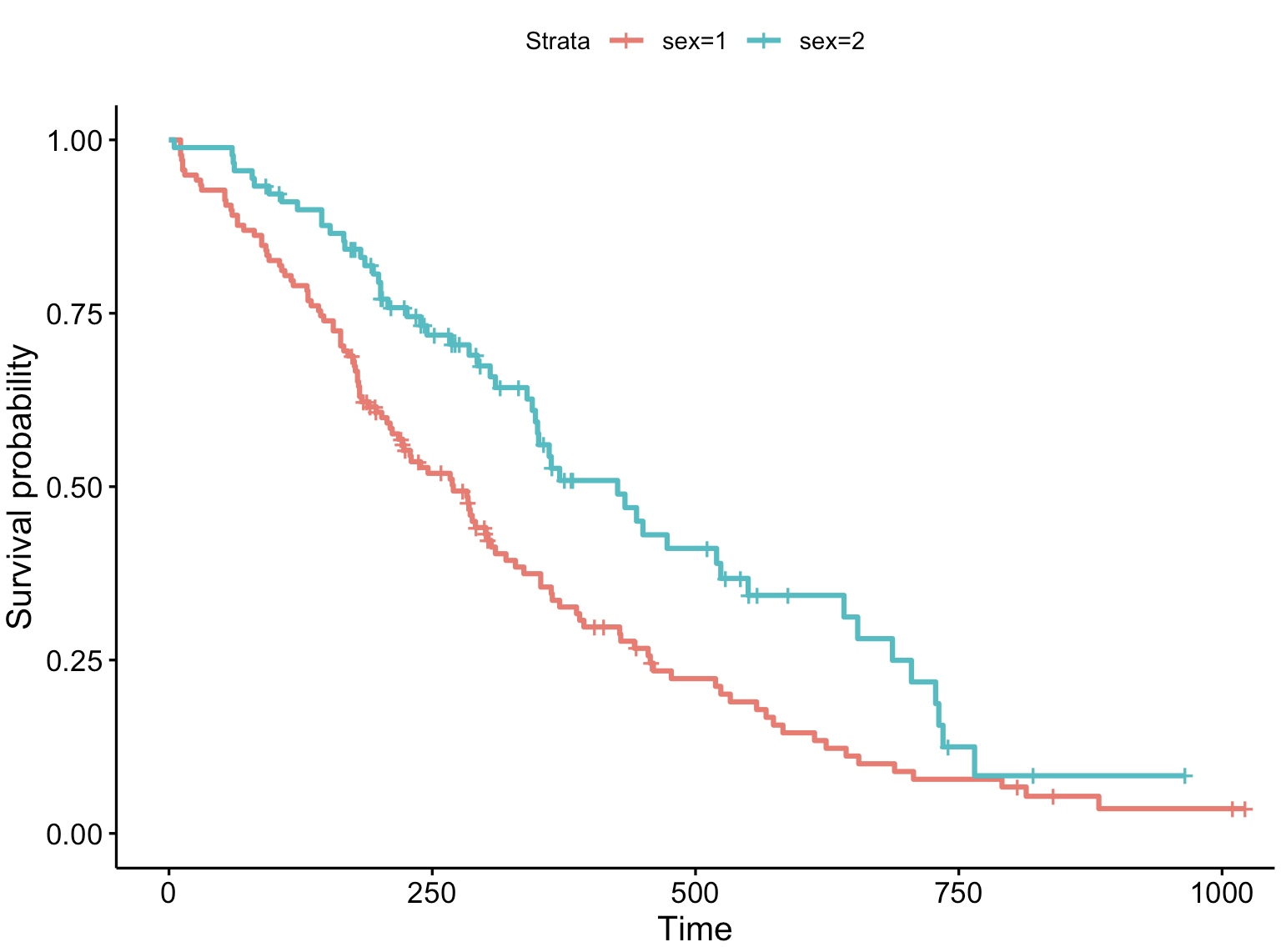
三、自定义曲线
3-1:增加中位生存时间
中位生存时间(median survival time)又称为生存时间的中位数,表示刚好有50%的个体其存活期大于该时间,是生存分析中常用的概括性统计量。
图解法是计算中位生存时间的方法。其利用生存曲线图,从纵轴生存率为50%处画一条与横轴平行的线,并与生存曲线相交,然后自交点画垂线与横轴相交,此交点对应的时间即为中位生存时间。
ggsurvplot(fit, data = lung, surv.median.line = "hv") # 增加中位生存时间
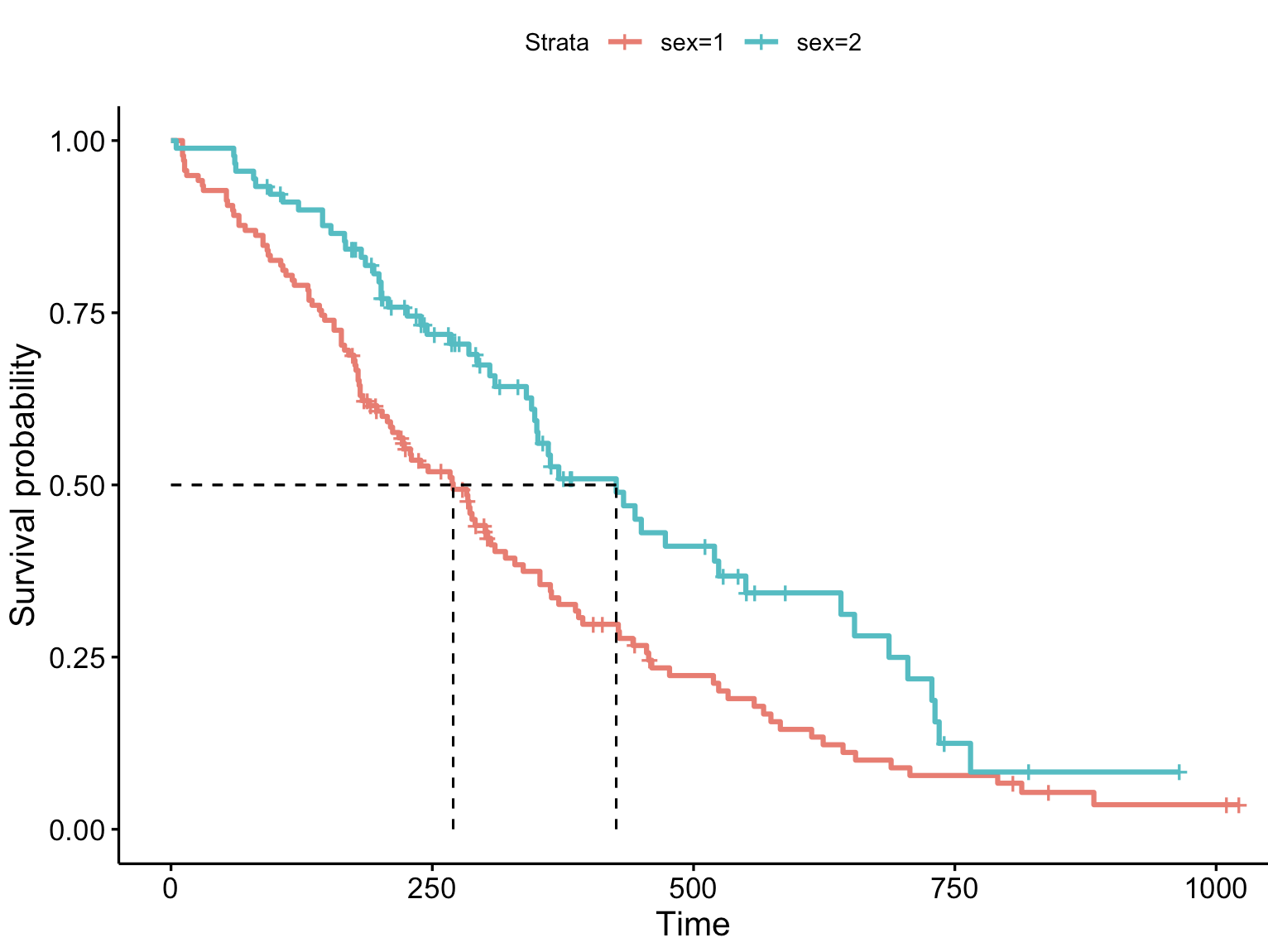
从图上可以看出,男性的中位生存时间小于女性。由之前的结果可知具体时间——男性为270,女性为426。
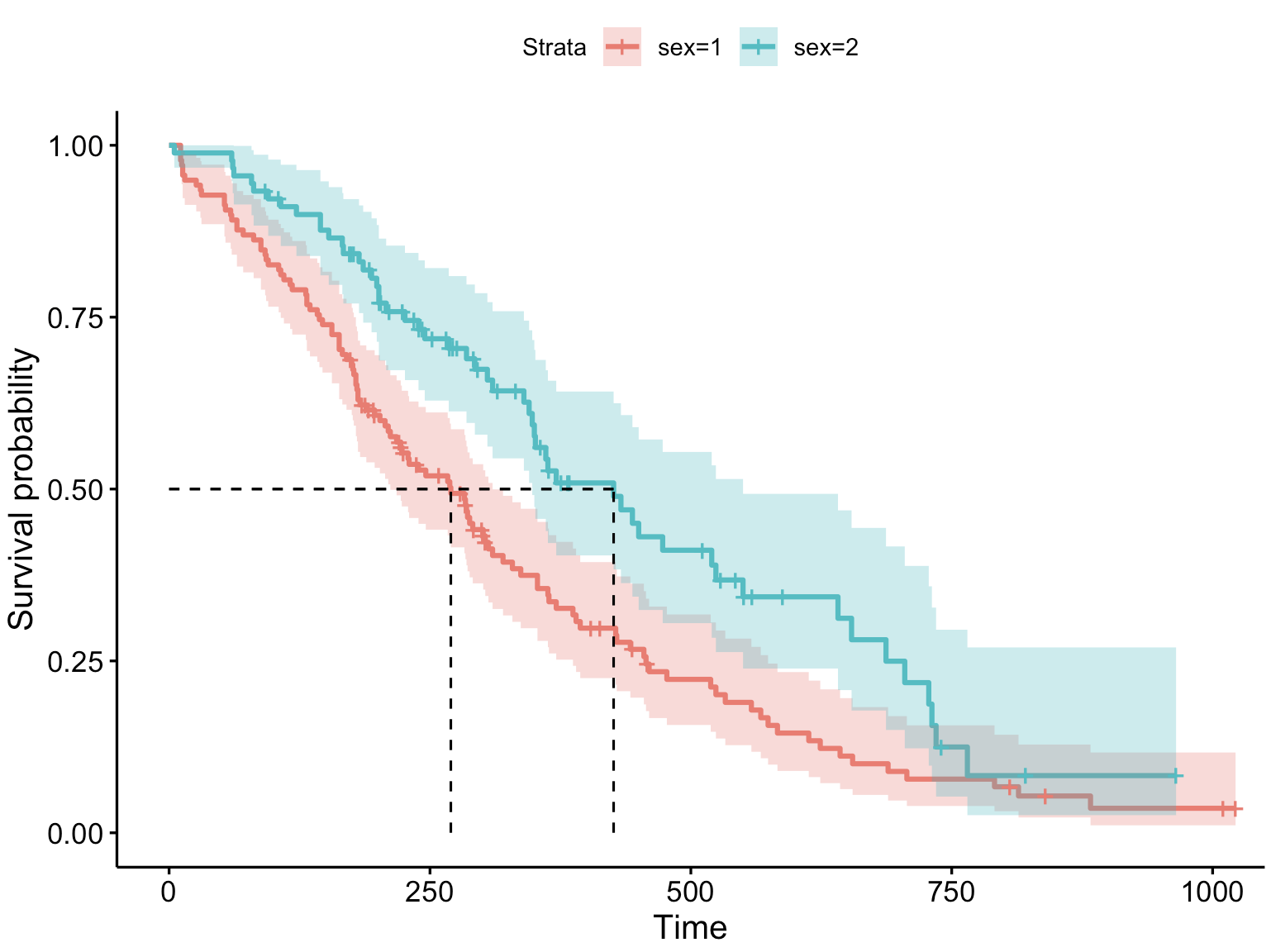
3-2:增加置信区间
ggsurvplot(fit, data = lung, surv.median.line = "hv",conf.int = TRUE) # 增加置信区间
3-3:累计风险曲线
当你在 ggsurvplot() 函数中设置 fun = "cumhaz" 时,函数会生成累积风险曲线,而不是默认的生存曲线。累积风险曲线通常用于展示在不同时间点之前至少一次发生事件的累积概率。
ggsurvplot(fit, data = lung, conf.int = TRUE,fun = "cumhaz") # 绘制累计风险曲线
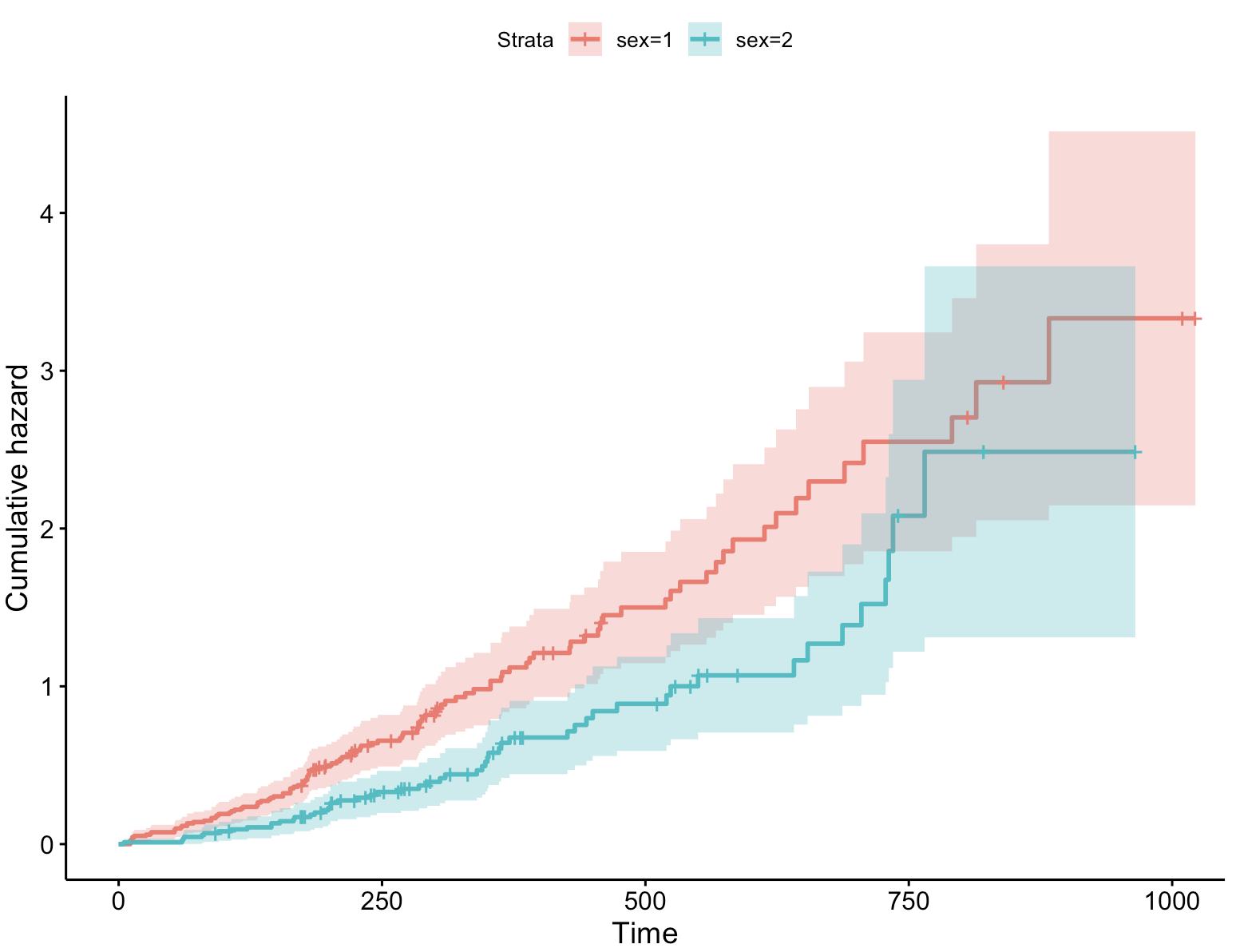
⚠️累积风险曲线在某些情况下比生存曲线更有用,尤其是在想要强调事件发生风险随时间的累积效应时。然而,累积风险曲线的解释可能比生存曲线更复杂,因此在选择使用时需要考虑分析的目的和受众。
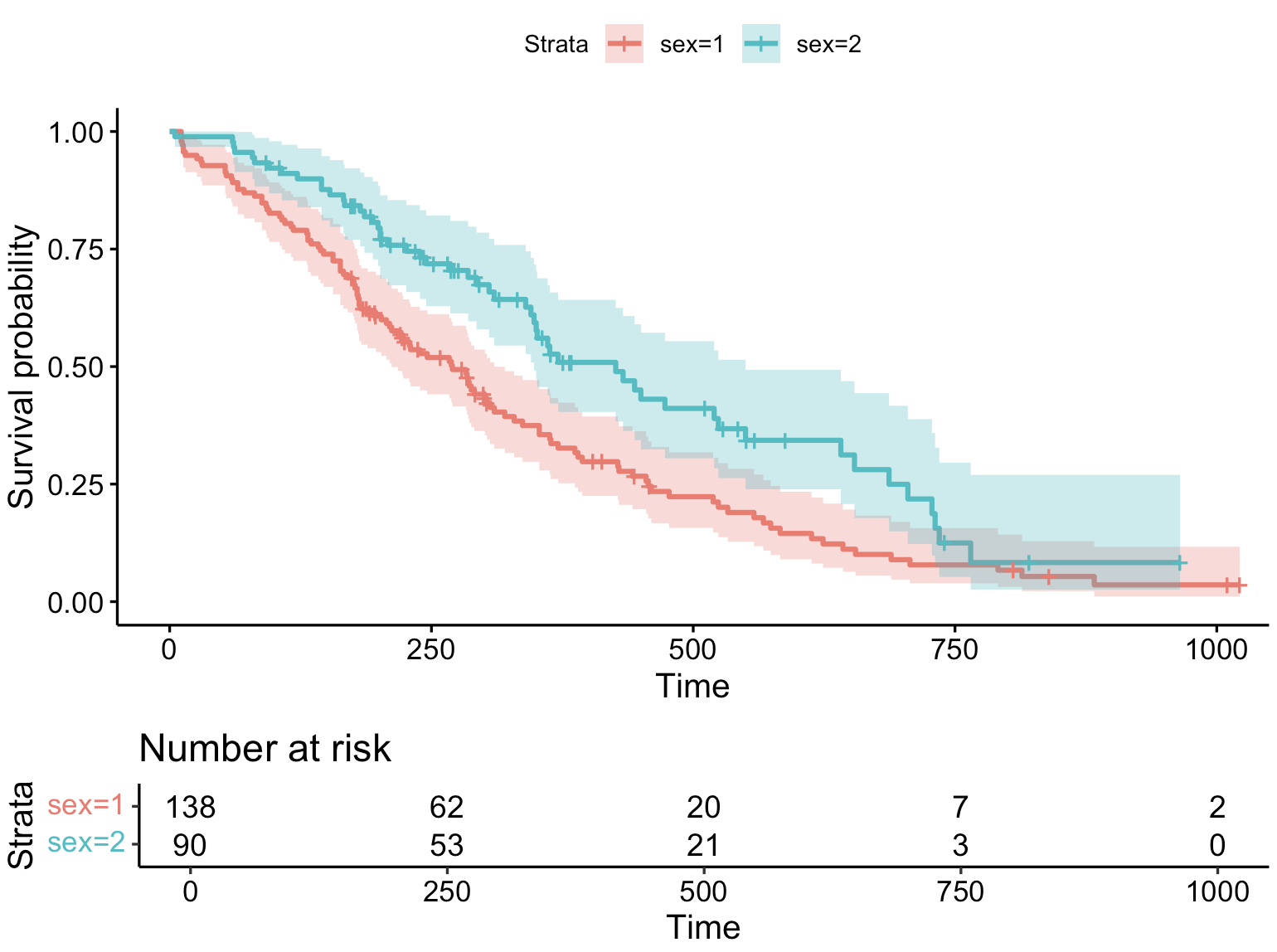
3-4:风险表
ggsurvplot(fit, data = lung, conf.int = TRUE,risk.table = TRUE) # 绘制累计风险曲线
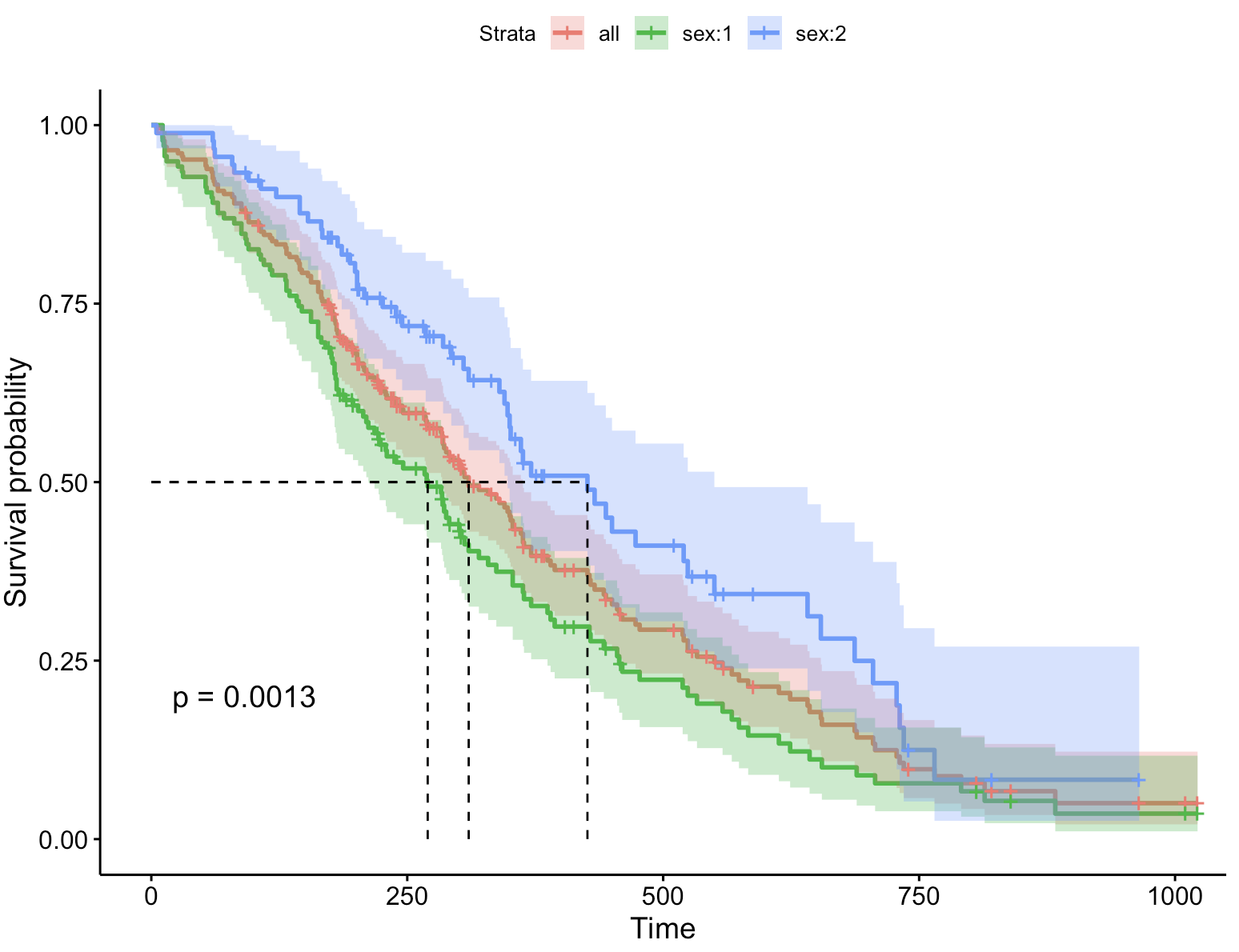
3-5:总患者生存曲线
ggsurvplot(fit, # 创建的拟合对象 data = lung, # 指定变量数据来源 conf.int = TRUE, # 显示置信区间 pval = TRUE, # 添加P值 surv.median.line = "hv", # 添加中位生存时间线 add.all = TRUE) # 添加总患者生存曲线
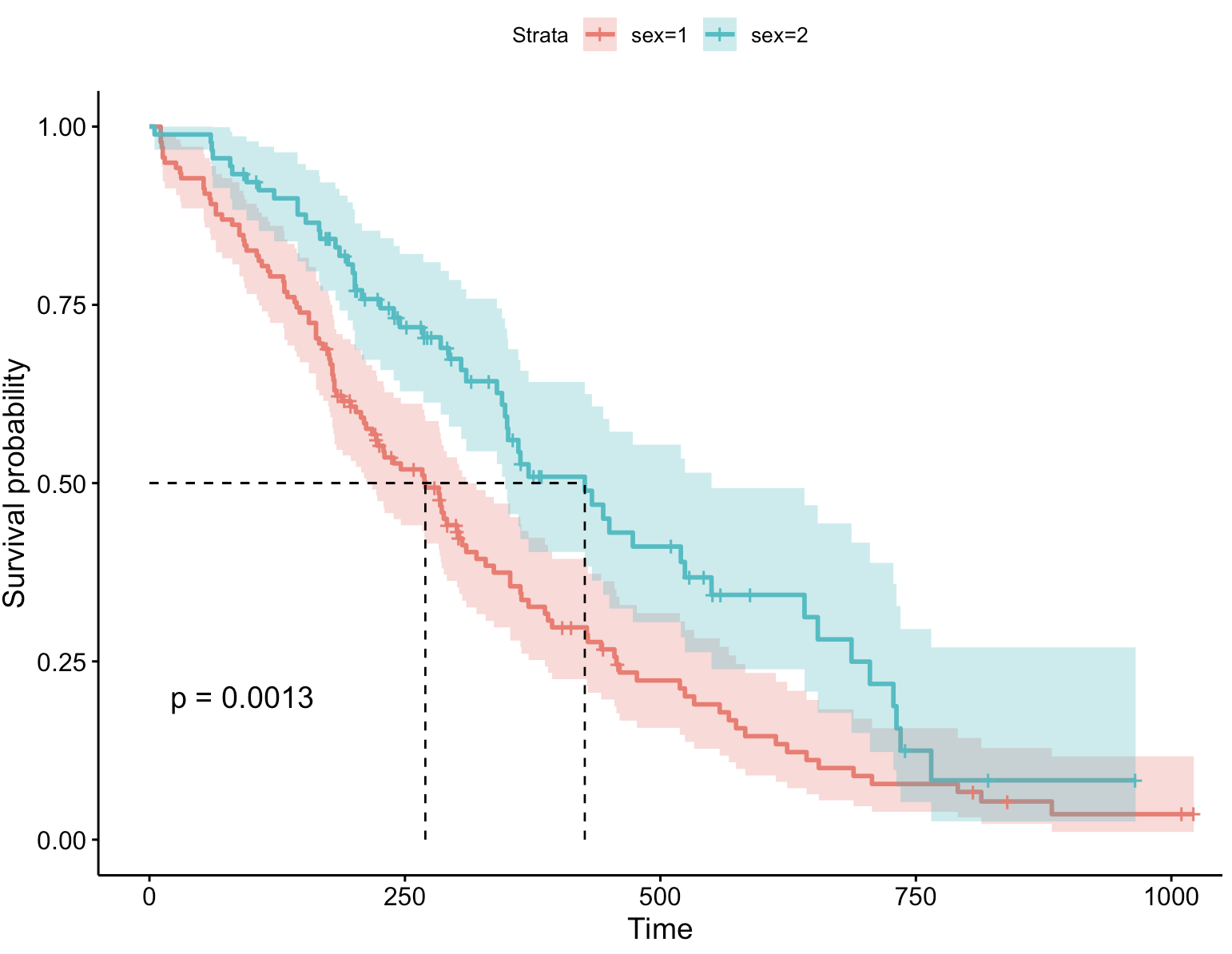
3-6:自定义调色板
ggsurvplot(fit, # 创建的拟合对象 data = lung, # 指定变量数据来源 conf.int = TRUE, # 显示置信区间 pval = TRUE, # 添加P值 surv.median.line = "hv", # 添加中位生存时间线 palette = "hue") # 自定义调色板
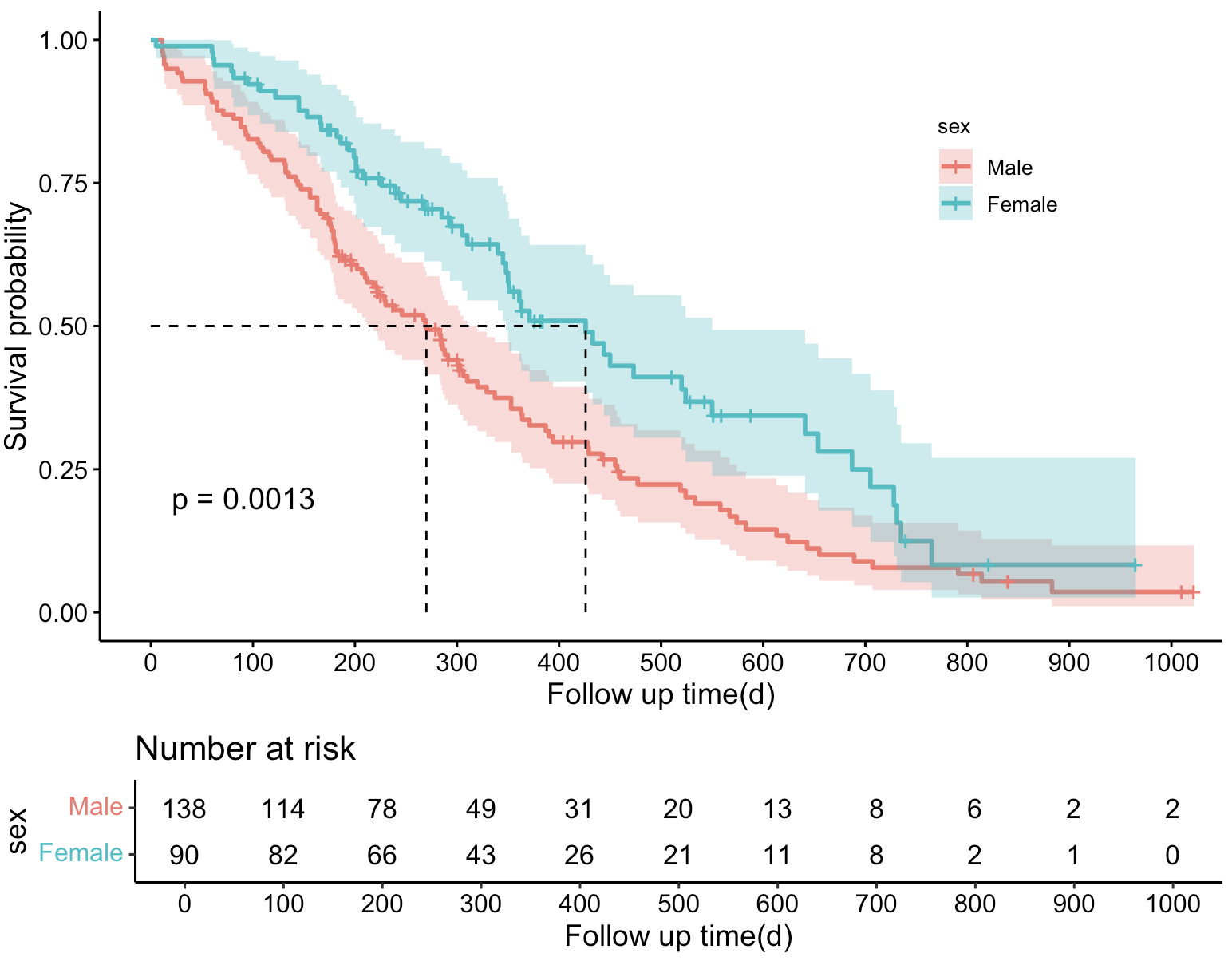
3-7:美化生存曲线
ggsurvplot(fit, # 创建的拟合对象 data = lung, # 指定变量数据来源 conf.int = TRUE, # 显示置信区间 pval = TRUE, # 添加P值 surv.median.line = "hv", # 添加中位生存时间线 risk.table = TRUE, # 添加风险表 xlab = "Follow up time(d)", # 指定x轴标签 legend = c(0.8,0.75), # 指定图例位置 legend.title = "sex", # 设置图例标题 legend.labs = c("Male", "Female"), # 指定图例分组标签 break.x.by = 100) # 设置x轴刻度间距
3-8:添加标题
ggsurvplot(fit, data = lung, conf.int = TRUE, pval = TRUE, surv.median.line = "hv", risk.table = TRUE, xlab = "Follow up time(d)", legend = c(0.8,0.75), legend.title = "sex", legend.labs = c("Male", "Female"), break.x.by = 100,title = "Author-LXLTX") # 添加图表标题