之前写过一篇文章,介绍了NetCDF文件格式,并详细讲解了如何使用Python对NetCDF文件进行读写操作,进而介绍了NetCDF文件的地理参考,最后以两个数据为例讲解了怎么将NetCDF格式的数据转GeoTIFF格式的数据。
但是上次介绍的NC文件的地理参考是比较标准的,即数据都有经度和维度这两个维度来表示其在地理空间的位置,并且这两个维度都有与其对应的坐标变量。在标准的NC文件中,坐标变量都是一维的。而不标准的NC文件的坐标变量则会是二维的,比如经度坐标变量存储的是每个像元的经度。通常情况下,这种NC数据甚至不能称为是栅格数据,因为需要这么存储的NC数据基本上每个像元的大小是不一致。而以TIF格式存储的栅格数据必须要求每个像元大小一致,所以这种数据处理起来就比较麻烦。下面介绍一下我碰到这种数据是怎么处理的,也欢迎有更好的方法的同学交流。关注公众号GeodataAnalysis,回复20230818获取示例数据和代码。
首先,既然知道每个像元的坐标值,那么可以把它转为点数据看一下。这是整体的每个点的分布,这种分布的情况下即使不考虑像元大小不一致的问题,我也没找到方法计算它的GeoTransform。

再看一下局部,显然点分布的密度差异很大,说明像元大小是不一致的。
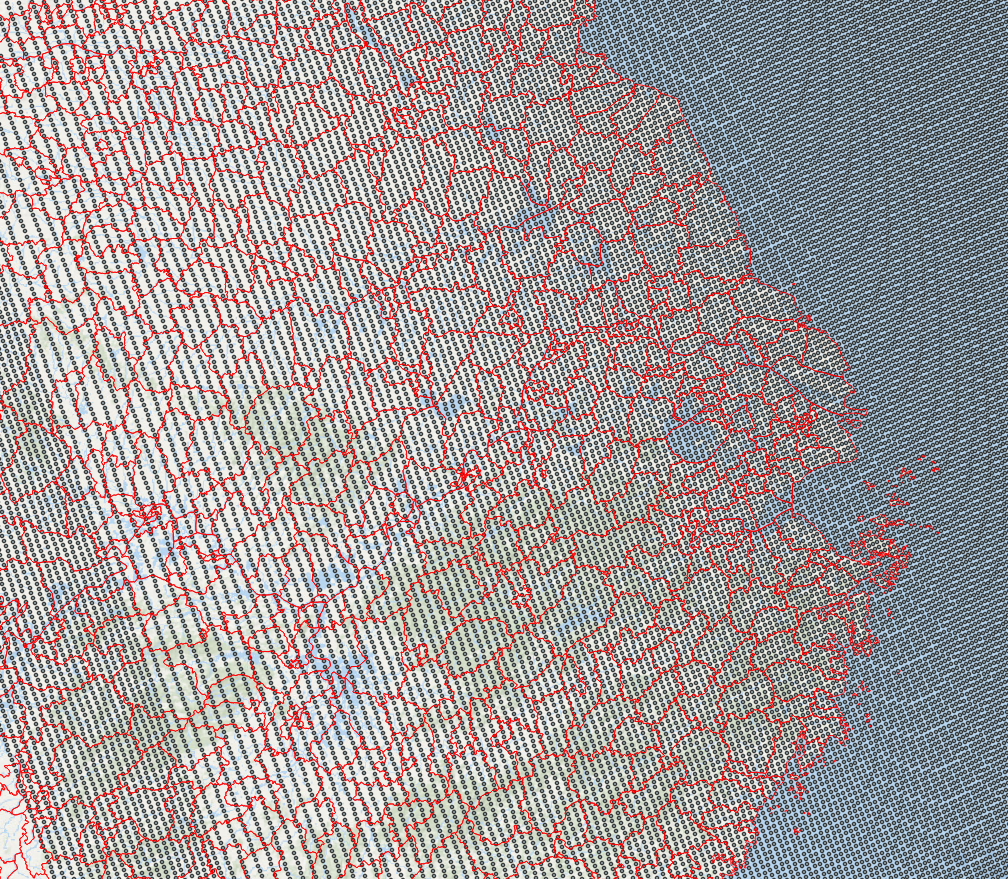
综上,在处理这种NC数据时,我是将其转为CSV格式,三列分别表示经度、维度和像元值,再使用GDAL的Grid函数把它插值为TIF格式的栅格数据。
下面以从哨兵 5 下载SO2数据为例,分享一下处理的代码(关注公众号GeodataAnalysis,回复20230818获取示例数据和代码):
import os
import numpy as np
import netCDF4 as nc
from osgeo import gdalds = nc.Dataset('./0228SO2.nc')
data = ds['PRODUCT']['sulfurdioxide_total_vertical_column'][0, ...].filled(np.NAN)lon = ds['PRODUCT']['longitude'][0, ...].data
lat = ds['PRODUCT']['latitude'][0, ...].datawith open('./0228SO2.csv', 'w') as fp:fp.write('x,y,z\n')for x, y, z in zip(lon.flatten(), lat.flatten(), data.flatten()):if not np.isnan(z):fp.write(f'{x},{y},{z}\n')vrt_path = './0228SO2.vrt'
with open(vrt_path, 'w') as fn_vrt:layer_name = '0228SO2'fn_vrt.write('<OGRVRTDataSource>\n')fn_vrt.write('\t<OGRVRTLayer name="%s">\n' % layer_name)# 下面这个路径最好使用绝对路径fn_vrt.write('\t\t<SrcDataSource>%s</SrcDataSource>\n' % './0228SO2.csv')fn_vrt.write('\t\t<GeometryType>wkbPoint</GeometryType>\n')fn_vrt.write('\t\t<GeometryField encoding="PointFromColumns" x="x" y="y" z="z"/>\n')fn_vrt.write('\t</OGRVRTLayer>\n')fn_vrt.write('</OGRVRTDataSource>\n')# 命令中的各个命令最好都用绝对路径
# 自行设置输出的分辨率或者输出的行列数,以及输入的坐标系
tif_path = './0228SO2.tif'
order = f'gdal_grid -a invdist:power=2.0:smoothing=1.0:nodata=-10000 -txe {lon.min()} {lon.max()} -tye {lat.min()} {lat.max()} -outsize 594 416 -a_srs EPSG:4326 -l {layer_name} {vrt_path} {tif_path} --config GDAL_NUM_THREADS ALL_CPUS'
result = os.system(order)
print(result)
结果如下图所示:

![java八股文面试[JVM]——JVM参数](https://img-blog.csdnimg.cn/aad554f27ae740339b5c2167bc5cf9b6.png#pic_center)




![java八股文面试[java基础]——接口和抽象类的区别](https://img-blog.csdnimg.cn/398c13d3972444f7b64cf221cdece643.png)