帕金森病(PD)是一种常见的神经系统退行性疾病,其特征是黑质(SNpc)中的多巴胺能神经元死亡。越来越多的证据表明,铜(Cu)参与了髓鞘的形成,并通过调节突触活动和神经营养因子诱导的兴奋毒性参与细胞死亡。
1.研究流程
图 1 显示了所有分析过程。首先,从GEO数据库中检索了PD-脑黑质测序数据,并利用GSE49036数据集筛选CR-DEGs,探索其相关性和单基因GSEA通路分析。然后探讨了单个基因与免疫细胞之间的相关性,构建了CR-DEGs mRNA-miRNA-lncRNA网络,构建了基因-药物网络,并进行了分子对接以探讨其结合的可行性。然后探索了存在于帕金森病中的 CR-DEGs 分子亚型,构建了具有临床和聚类分型的 WGCNs,并将聚类分型与临床模块基因交叉。然后利用交叉基因通过机器学习构建诊断模型,并利用 GSE7621 和 GSE20141 验证了该诊断模型。
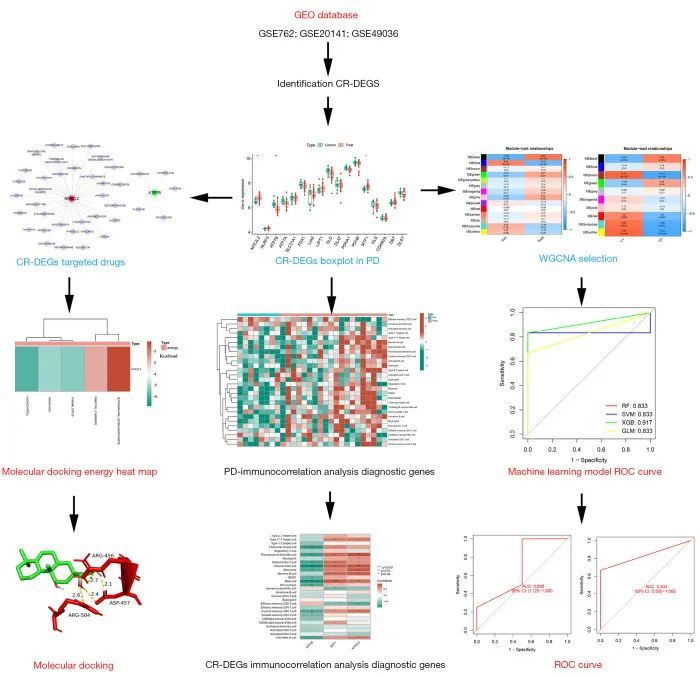
图1 与杯突症相关的诊断基因和药物治疗的系统分析流程图
2.鉴别帕金森病的 CR-DEG 及基因间的相关性分析
对 GSE49036 数据进行归一化处理,使用 R 软件的 limma 包提取 CRGs,并进行 DEG 分析筛选。根据筛选标准:根据筛选标准:P<0.001,用 "***"表示;P<0.01,用 "**"表示;P<0.05,用 "*"表示,使用pheatmap和ggpubr软件包绘制热图(图2A)和箱形图(图2B),直观显示差异基因表达结果。
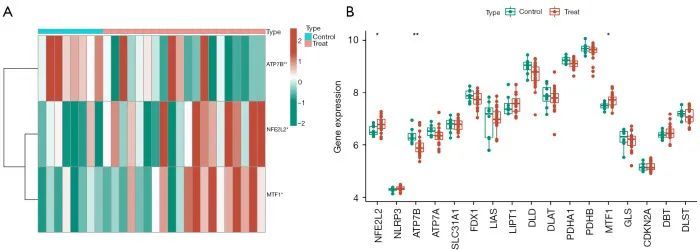
图2 PD中的CR-DEGs
利用 R 软件的 corrplot 软件包对上述基因在 PD 患者中的表达进行了相关性分析,结果显示 ATP7B 与 NFE2L2 和 MTF1 负相关,NFE2L2 与 MTF1 正相关。利用circlize软件包还可视化了基因间表达相关性的结果(图3)。
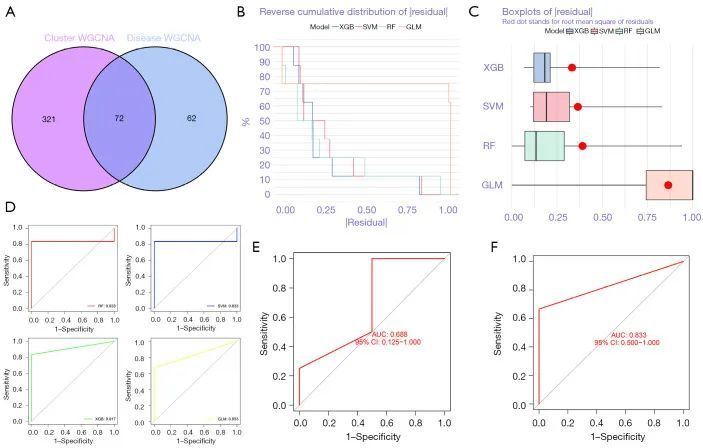
图3 CR-DEG 的相关性
3.PD 组织和 CR-DEG 的 GSEA
如图 4 所示,为了探索杯突相关基因如 NFE2L2、MTF1 和 ATP7B 在帕金森病中的潜在作用机制,作者进行了单基因 GSEA 富集分析。有趣的是,虽然NFE2L2和MTF1下调,ATP7B下调,但这3个基因都与突触囊泡循环有关。相反,ATP7B上调的信号通路还与柠檬酸循环(TCA循环)、尼古丁成瘾、视黄醇代谢、淀粉和蔗糖代谢等有关,而NFE2L2和MTF1则共同与蛋白酶体有关,MTF1下调的信号通路则与2-氧代羧酸代谢、糖磷脂生物合成-球蛋白和异球蛋白系列等有关。NFE2L2 下调与氨基酰-tRNA 生物合成、糖磷脂生物合成-球型和等球型系列等有关。
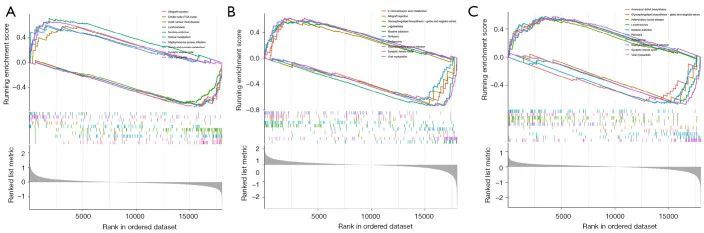
图4 ATP7B、MTF1 和 NFE2L2 通路相关性探索
图5 图表格式相同
这些结果表明,NFE2L2、MTF1 和 ATP7B 可能参与了突触小泡循环的调控,进而影响脑神经递质的传递,并可能参与脑组织的有氧代谢,导致疾病进展。
4.PD组织和CR-DEG的免疫相关性分析
为了探讨PD中CR-DEGs的免疫细胞浸润,作者使用免疫基因组 "immune.gmt "对数据集GSE49036进行了GSEA,如图5A、5B所示。CD8 T细胞在正常对照组与PD组织中高表达,而质体树突状细胞(DC)、巨噬细胞、未成熟B细胞、髓源抑制细胞(MDSCs)、肥大细胞、中性粒细胞、自然杀伤(NK)T细胞、2型T辅助细胞、T滤泡辅助细胞和其他免疫细胞在PD黑质与正常脑组织中高表达。作者还探讨了 CR-DEGs 的个别基因与免疫细胞之间的关系(见图 6)。ATP7B 与活化的 B 细胞、CD56 dim NK 细胞、中央记忆 CD4 T 细胞、中央记忆 CD8 T 细胞、巨噬细胞、肥大细胞、MDSC、单核细胞、NK 细胞、NK T 细胞、浆细胞 DC 和 T 滤泡辅助细胞相关、和其他免疫细胞呈负相关,而 NFE2L2 与 MTF1 和 17 型 T 辅助细胞、类浆细胞 DC、NK T 细胞、中央记忆 CD8 T 细胞、NK 细胞、单核细胞呈负相关。记忆B细胞、肥大细胞和活化B细胞呈正相关,有趣的是,它们与效应记忆CD8 T细胞的表达呈负相关;这些免疫细胞可能参与了PD中脑黑质多巴胺能神经元的变性死亡,而NFE2L2、MTF1和ATP7B等CR-DEGs可能是PD免疫治疗的有希望的靶点。
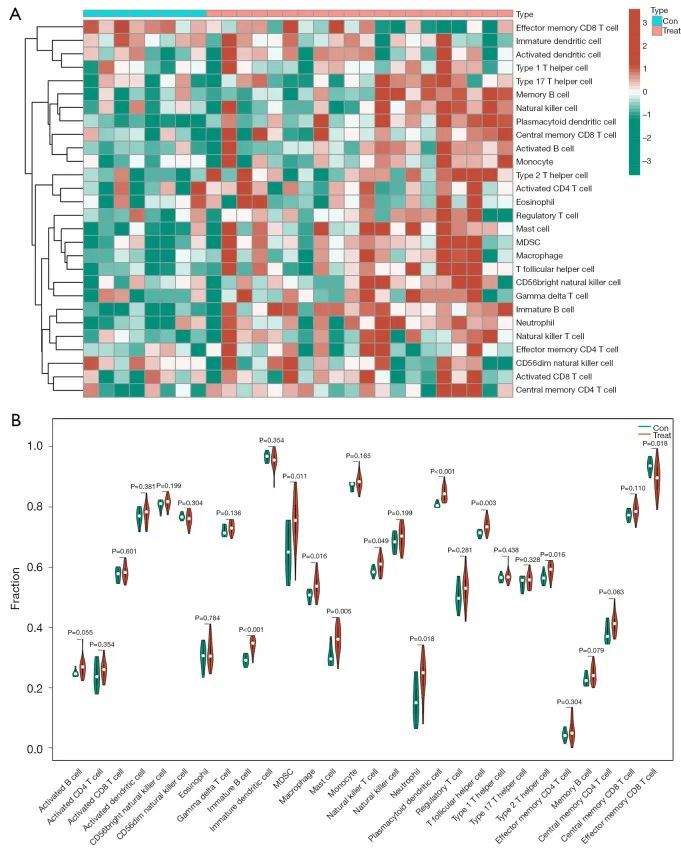
图5 PD和正常情况下免疫浸润模式的免疫基因组分析
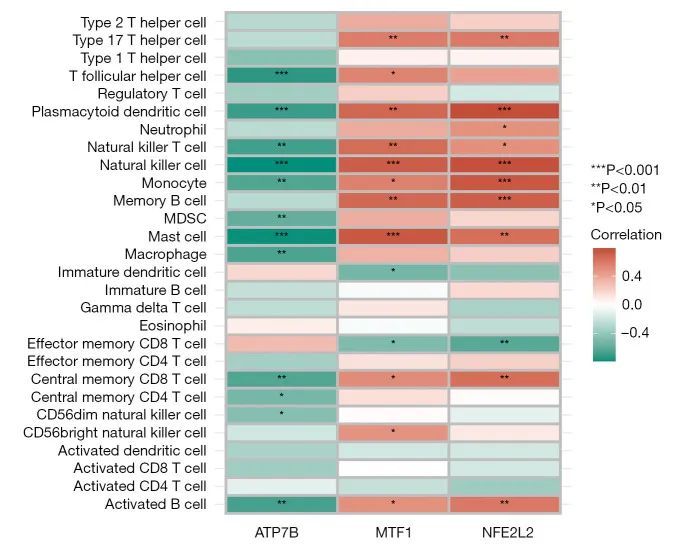
图6
5.PD-CR-DEGs ceRNA 网络搜索
为了进一步探讨NFE2L2、MTF1和ATP7B基因之间复杂的分子相互作用机制,作者绘制了一个mRNA-miRNA-lncRNA网络关系图,如图7所示。这个复杂的网络有60个节点和99个相互作用,其中包括40个miRNA和17个lncRNA,这可能为作者探索杯突相关性脊髓灰质炎的发病机制提供了一个新的思路。
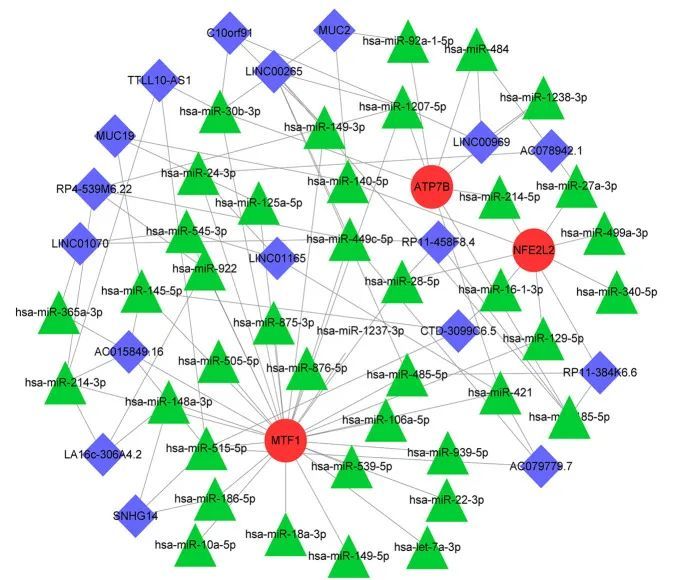
图7
6.治疗帕金森病的前瞻性小分子药物
为了探索用于PD治疗的前瞻性小分子药物,作者从DGIdb数据库中下载了NFE2L2、MTF1和ATP7B靶向药物的结果,并选择了有记录结果的药物,用Cytoscape展示了基因-药物结果(图8A)。最后,作者选择了 NFE2L2 基因-药物相互作用得分前 5 位的药物,利用 AutoDock 进行小分子药物-大分子蛋白质对接。对接所需的能量如图 8B 所示,其中 LAGASCATRIOL 与 NFE2L2 的对接所需的能量最低。图 9A 显示了 NFE2L2 蛋白与 LAGASCATRIOL 分子对接结果的全貌,图 9B 显示了小分子药物与蛋白质分子对接结果的局部视图,见图 9C。LAGASCATRIOL 能与 NFE2L2 第 456 位和第 504 位的精氨酸分别形成 1 个离子键,与第 457 位的天冬氨酸形成 2 个离子键。
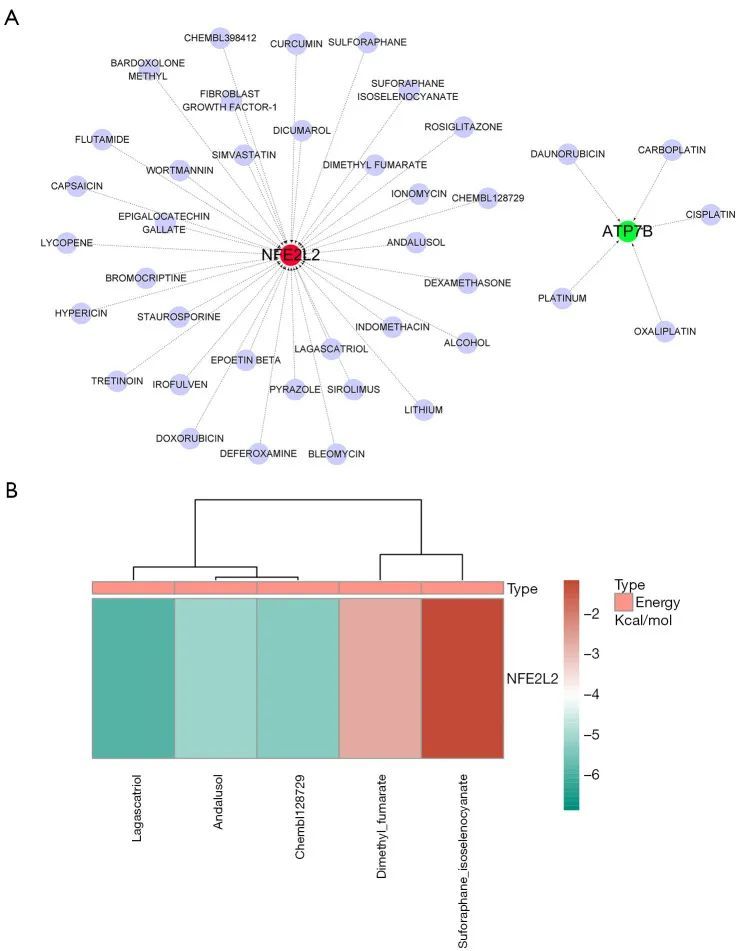
图8
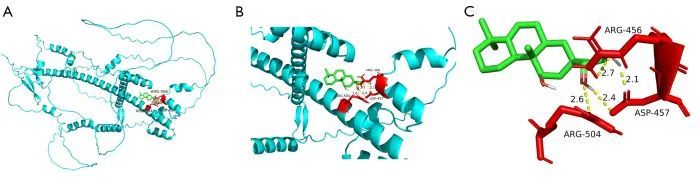
图9 NFE2L2 蛋白与 LAGASCATRIOL 的对接
7.基于 PD 样本中 CR-DEG 的聚类
在 GSE49036 中,作者进行了聚类分析,将 PD 样本分为不同的分子亚型,并根据上述选定的 3 个 CR-DEGs 的表达情况使用 PCA 对亚型进行了检验(图 10A、10B)。作者将这两个聚类分别命名为 CRGClusters C1 和 C2。对这 2 个 CRGClusters 的差异分析表明,C1 中 ATP7B 的表达量明显高于 C2,而 C1 中 NFE2L2 和 MTF1 的 ATP7B 表达量明显高于 C2,C2 的 ATP7B 表达量则明显低于 C1(图 10C)。在《京都基因组百科全书》(KEGG)中,比较 GSEA 中的两个群组,也发现了明显不同的 KEGG 通路富集谱,有趣的是 PARKINSONS_DISEASE 上,泛素介导的蛋白酶解、氧化磷酸化和柠檬酸循环 tca 循环也出现了上调,而 tgf beta 信号通路和剪接体则出现了下调(图 10D)。相反,在基因本体(GO)生物学下调的通路中:免疫突触形成、造血的正调控、b 细胞分化的正调控、内胚层细胞分化的调控、突触小泡内吞、树突延伸、突触前密核小泡外吞、突触后小泡外吞的调控和突触后细胞膜钙离子浓度的调控(图 10E)。这些结果表明,神经递质可能参与了大脑中神经递质的产生和传输,也可能参与了与帕金森病进展有关的树突状突触的产生,导致纹状体 DA 含量显著减少。
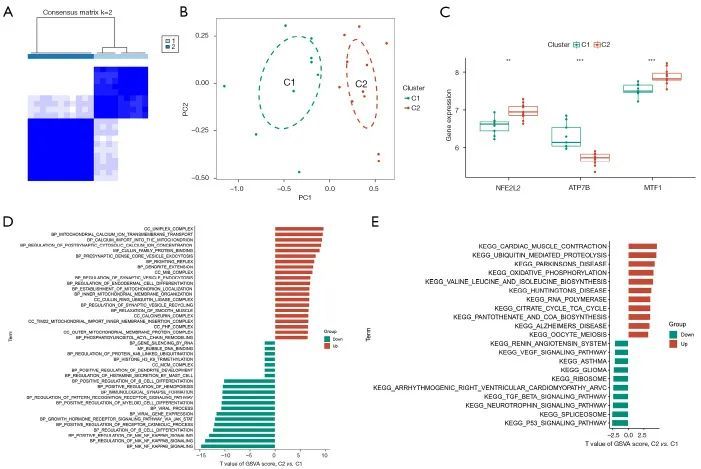
图10
8.脊髓灰质炎样本的 WGCNA 共表达分析
作者首先对临床性状相关性进行了 WGCNA 分析,以选择波动最大的前 25% 基因进行 WGCNA 分析,并切断离群样本,将其余样本纳入分析(图 11A)。如图 11B 所示,当功率 =8 且平均一致性较高时,标度独立性达到 0.9。因此,使用功率 =8 来构建共表达模块,以获得初步的模块划分结果,WGCNA 的结果显示,不同的模块被识别为不同的颜色(图 11C)。为了检测离群值,利用模块的特征值构建树,然后合并属于同一分支的距离很近的树,并将截距值设为 0.5(图 11D)。共表达模块的构建如图 11E 所示,合并相似模块后得到结果。根据每个模块中样本的特征值和样本的特征,进行相关性分析以确定与特定性状相关的模块,结果发现以 MEblack 为代表的基因在 PD 中高度正相关(图 11F)。
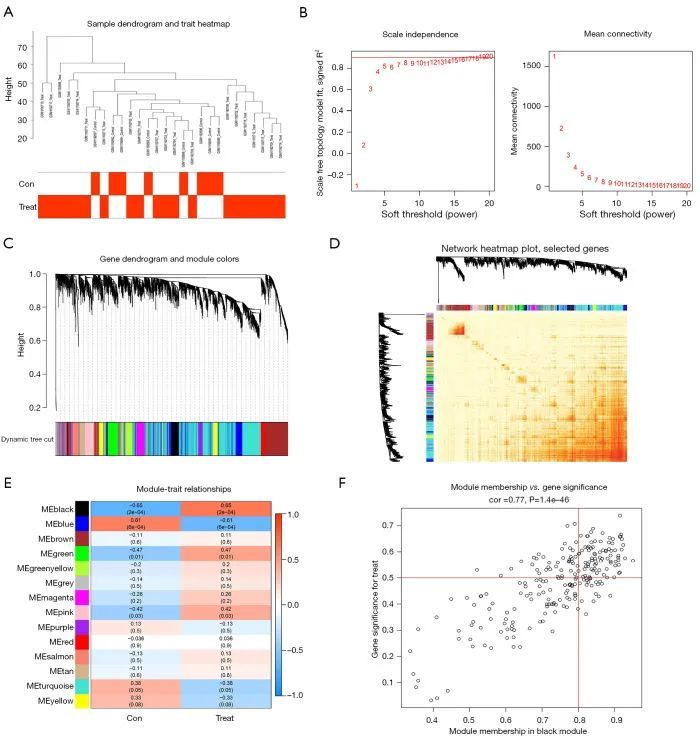
图11 PD 疾病相关模块共表达基因的 WGCNA 选择
与作者在分型后进行 WGCNA 相关性分析的方法相同,当幂次 =7、标度独立性达到 0.9 时,作者像图 12B 一样切掉离群样本(图 12A),通过颜色识别模块,最终发现分型后的特征(图 12C-12F),以 MEyellow 为代表的基因在 CRGCluster C1 中高度正相关(图 12F)。
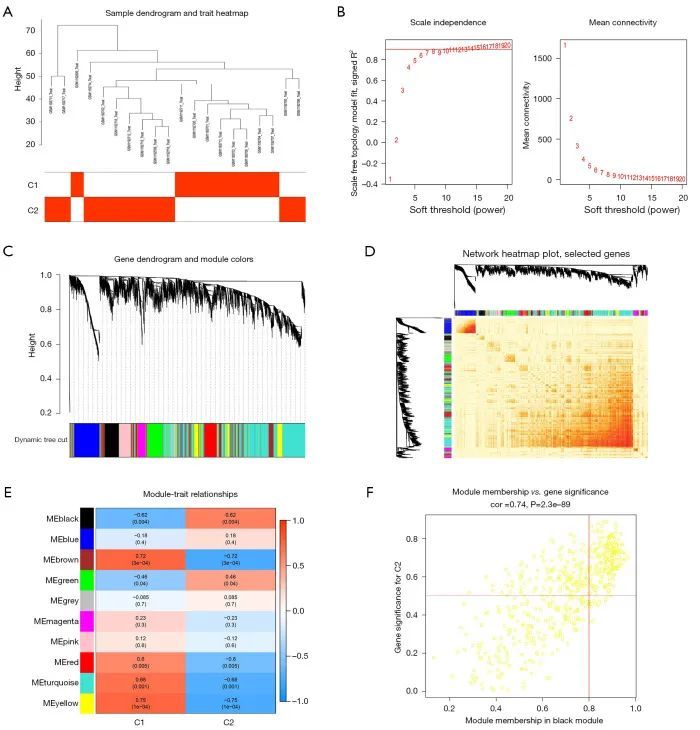
图12 WGCNA 挑选 PD 群相关模块共表达基因
9.机器学习建立诊断模型
作者将临床性状和分型的常见性状基因模块进行交叉,共找到 72 个性状基因(图 13A)。作者使用 GSE49036 数据集提取了这 72 个性状基因,并使用 RF 树、SVM、XGB 和 GLM 算法建立了性状诊断模型。作者首先分析了数据样本的残差,绘制了四种算法残差的反向累积分布图(图 13B)。XGB 模型在大多数样本中的残差较小,而 GLM 在许多样本中的残差高于 XGB 模型。4 种算法残差的箱形图(图 13C)中,红点表示残差的均方根,排序如下:XGB < SVM < RF < GLM,并用 ROC 曲线验证了这 4 个模型。四个模型的 AUC 值如下:RF =0.833;SVM =0.833;XGB =0.917;GLM =0.833(图 13D)。
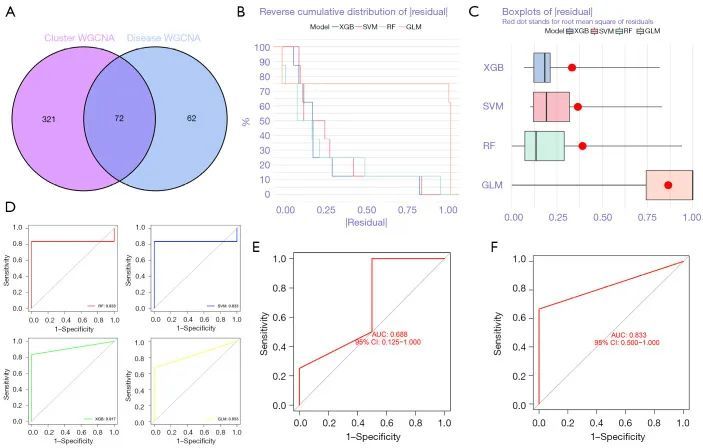
图13
最后,作者选择了 XGB 模型,并使用 GSE7621 和 GSE20141 基因集验证了 XGB 模型的 ROC 曲线(图 13E、13F),相应的 AUC 值分别为 0.688 和 0.833。最后,将模型中的基因绘制成提名图(图 14),结果显示模型中的基因有 SLC35D3、FLJ22184、SYT17184、SLC35D3、FLJ22184、FLJ22184、SYT17 和 RIMS3。
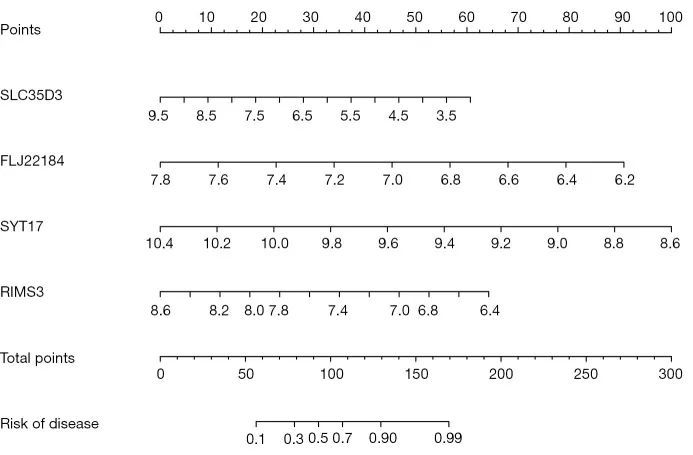
图14 作者构建了一个提名图来预测帕金森病进展的概率
总结
通过生物信息学分析,在帕金森病患者的黑质中发现了 CR-DEG。ATP7B以及NFE2L2和MTF1被认为是有待进一步研究的候选基因。这项研究提供了在帕金森病发病机制中与铜积累导致细胞死亡相关的新基因,并为相关的神经退行性疾病探索了有前景的治疗靶点。