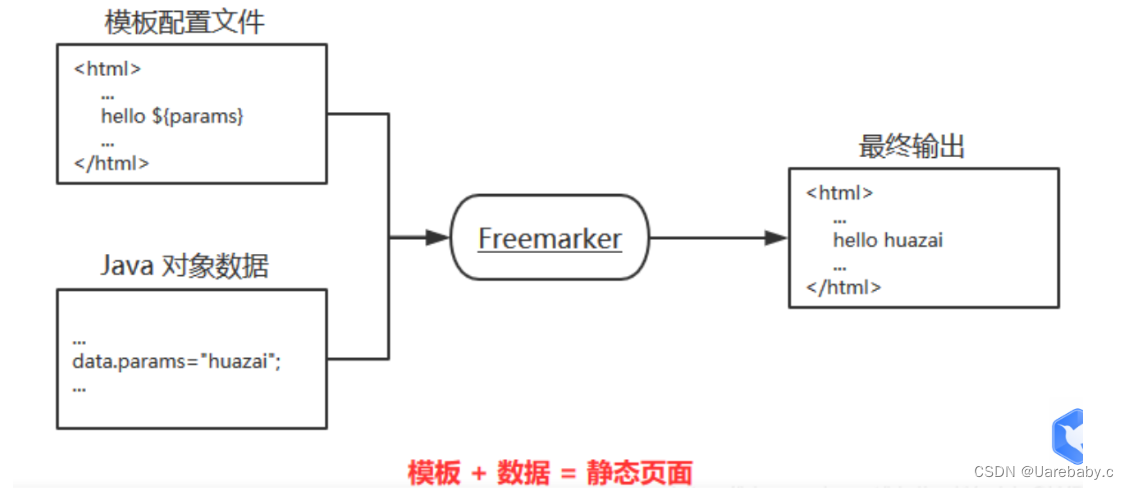
大家好,今天我们来画单细胞中最简单的细胞比例图~
1.老规矩,先加载pbmc数据
dir.create("~/gzh/细胞比例")setwd("~/gzh/细胞比例")subset_data=readRDS("~/gzh/pbmc3k_final.rds")table(stringr::str_split(string = colnames(subset_data),pattern = "-",simplify = TRUE)[,2])subset_data$stim=ifelse(subset_data$nCount_RNA>2000,yes = "control",no = "exp")library(Seurat)library(ggplot2)
2.为了画出组别,人为在pbmc中添加一列组别信息,并看一下metadata信息
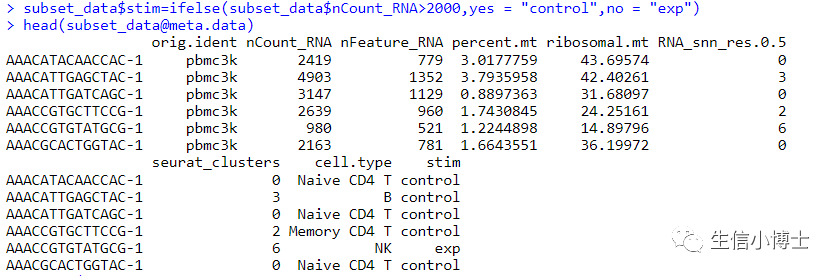
如果你不理解这一步的话,可以看我的数据结构视频讲解,会帮助你理解的
单细胞直播一理解seurat数据结构与pbmc处理流程
单细胞直播三seurat数据结构与数据可视化
3.画出来的草图
ggplot(subset_data@meta.data,aes(x=Idents(subset_data), fill=stim)) + geom_bar(position = "fill")+RotatedAxis()

4.另外一种ggplot图更好看
ggplot(subset_data@meta.data,aes(x=stim, fill=Idents(subset_data))) + geom_bar(position = "fill")+RotatedAxis()

5 如果还不满意,就是使用ggplot2美化单细胞图表的问题啦,大家可以各显神通,我在这里抛砖引玉
方法一
ggplot(subset_data@meta.data, aes(x=stim, fill=Idents(subset_data))) + geom_bar(position = "fill")+RotatedAxis()+theme_classic()
方法二 图形横轴和纵轴的字体更清晰些
gplot(subset_data@meta.data,aes(x=stim, fill=Idents(subset_data))) + geom_bar(position = "fill")+RotatedAxis()+theme_classic()+ theme(axis.text.x=element_text(angle=90,hjust = 1,vjust=0.5,size = 20),axis.text.y=element_text(size = 19),axis.text = element_text(color = 'black', size = 12))+# scale_fill_gradient(low="red",high="blue")+labs(x=NULL,y=NULL)
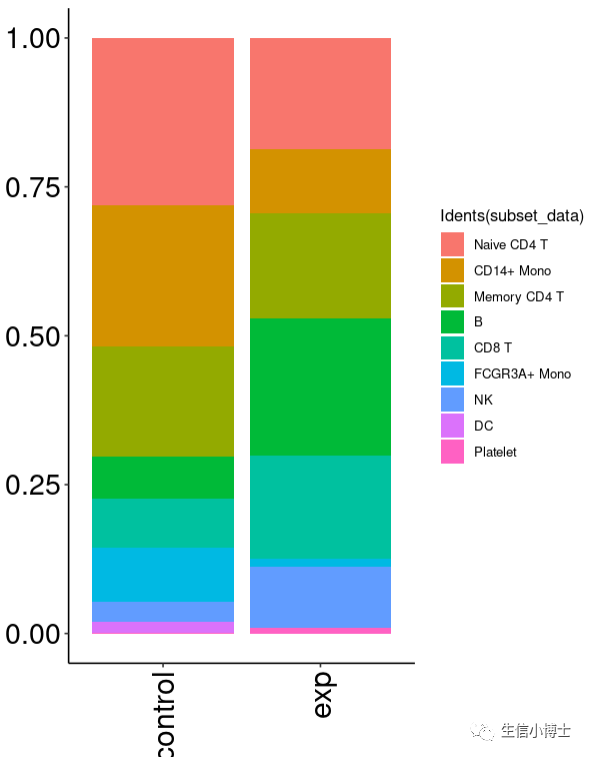
更多美图可以关注我微信公众号:生信小博士
单细胞featureplot美化修改-自定义修改图片样式-umap密度图画等高线





![[密码学]DES](https://img-blog.csdnimg.cn/direct/35cb8bb4644e4840a37ff4d27f13b99f.png)