随机森林(Breiman 2001a)(RF)是一种非参数统计方法,需要没有关于响应的协变关系的分布假设。RF是一种强大的、非线性的技术,通过拟合一组树来稳定预测精度模型估计。随机生存森林(RSF)(Ishwaran和Kogalur,2007;Ishwaraan,Kogalur、Blackstone和Lauer(2008)是Breimans射频技术的延伸从而降低了对时间到事件数据的有效非参数分析。
接着文章《机器学习系列–R语言随机森林进行生存分析(1)》, 咱们继续分析
上一节,咱们已经介绍了通过VIMP来绘制变量的重要性。
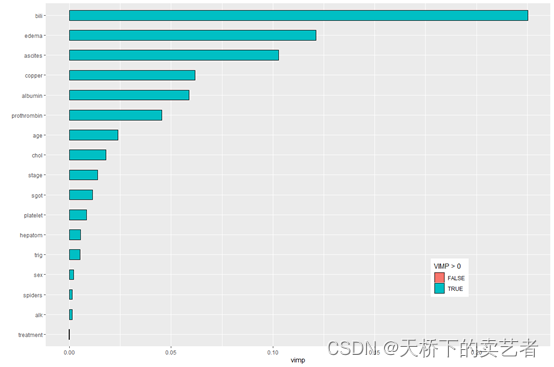
在 VIMP 中,预后风险因素是通过在其他数据设置下测试森林预测来确定的,根据对森林预测能力的影响对最重要的变量进行排序。
randomForestSRC包中还有另一种方法是就是利用对森林构建的检验来对变量进行排序,就是最小森度。最小深度(Ishwaran 等人,2010 年;Ishwaran、Kogalur、Chen 和
Minn 2011)假定,对预测影响大的变量是那些最频繁地分割离根节点最近的节点的变量,它们在根节点上分割了最大的群体样本。在每棵树中,节点级别根据其与树根的相对距离进行编号(树根为 0)。最小深度是通过对森林中所有树的每个变量的第一次分割深度取平均值来衡量重要的风险因素。
该指标的假设是,较小的最小深度值表明该变量分离了大组观测值,因此对森林预测的影响较大。
一般来说,要根据 VIMP 选择变量,我们要检查 VIMP 值,寻找 VIMP 测量值差异较大的排序点。但是最小深度是森林构建的定量属性,Ishwaran 等人(2010 年)还推导出了变量影响证据的分析阈值。规则使用最小深度分布的平均值,将最小深度低于该阈值的变量归类为森林预测中的重要变量。
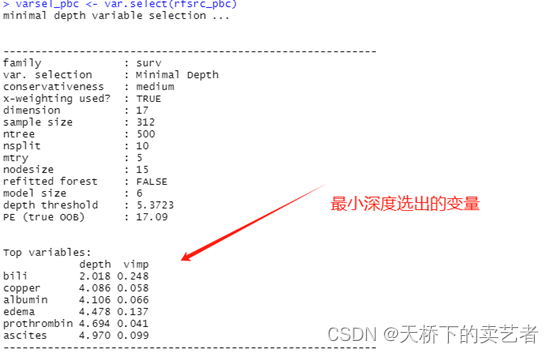
varsel_pbc <- var.select(rfsrc_pbc)
topvars <- varsel_pbc$topvars
gg_md <- gg_minimal_depth(varsel_pbc, lbls = st.labs)
print(gg_md)

综合上面两图,咱们可以得到,最小深度阈值(depth threshold)为5.2757,共筛选了15个变量,第一个进行分裂的变量就是bili,在深度2.144就开始分裂了,接着就是albumin和copper。
绘制深度节点和变量图
plot(gg_md)
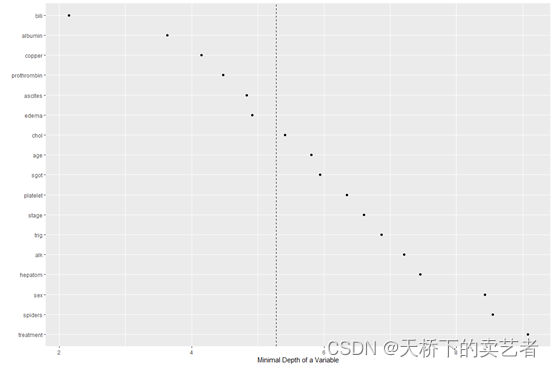
从上图可以看出虚线就是最小深度,越往右深度越大,其中较小的最小深度值表示较高的重要性,较大的值表示重要性较低。
由于我们现在有两个指标来判定,那选哪个好呢?我们可以使用gg_minimal_vimp函数来进行综合比较,
plot(gg_minimal_vimp(gg_md)) +theme(legend.position=c(0.8, 0.2))
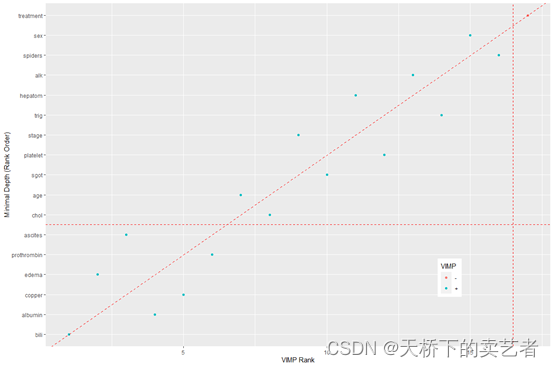
这个图形是这个包中的一个核心图形,我要好好解释一下。因为这张图使用两个方法,vimp和最小深度法。这条斜着的虚线是这两种方法的分界点,蓝色的点代表vimp大于0的,红色的点代表vimp小于0。红色斜着的虚线上的点,代表这个变量在两种分类方法排名相同,高于红色虚线上的点,代表它的vimp的排名更加高,低于红色虚线上的点,表明它的最小深度排名更高。
看它生成的表格也可以看出来
out2<-gg_minimal_vimp(gg_md)
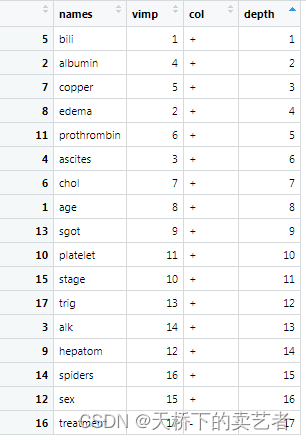
我们可以看到两种方法有些排名是一样的,有些是不一样的。如果我们根据阈值5.2757进行筛选,那么最终可以选出"bili" ,“albumin” ,“copper” ,“prothrombin” ,"edema"这5个变量,有些文章介绍有临床意义的变量也是可以选进来的。
接下来绘制部分依赖图(PDP),假设咱们想了解"bili"这个变量,对1年和3年生存结局的影响(也就是依赖性),咱们先生成这个结局治疗的数据
gg_v <-gg_variable(rfsrc_pbc, time = c(1, 3),time.labels = c("1 Year", "3 Years"))
进行绘图,注意形状散点代表的意义不一样
plot(gg_v, xvar = "bili", alpha = 0.4) + #, se=FALSElabs(y = "Survival", x = "bili") +theme(legend.position = "none") +scale_color_manual(values = c("red", "blue"), labels = c("1 Year", "3 Years")) +coord_cartesian(ylim = c(-0.01, 1.01))+xlab("Serum Bilirubin")
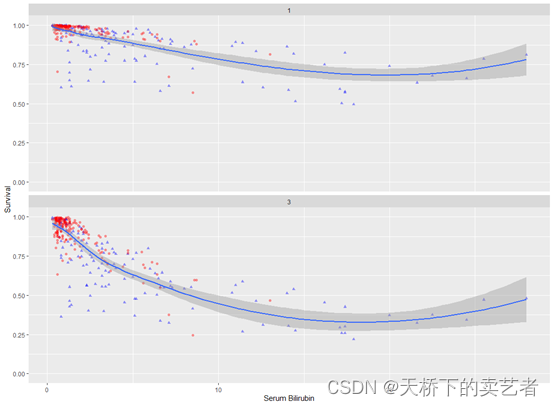
ggRandomForests包的绘图函数画起来不咱们美观,我们可以根据结局数据自己来画
ggplot(gg_v) + geom_point(aes_string(x = "bili", y = "yhat", color = "event", shape = "event"))+geom_smooth(aes_string(x = "bili", y = "yhat", color = "time",fill="time"))+theme_classic()+xlab("bili")+ylab("yhat")

当然咱们也可以分面
ggplot(gg_v) + geom_point(aes_string(x = "bili", y = "yhat", color = "event", shape = "event"))+geom_smooth(aes_string(x = "bili", y = "yhat", color = "time",fill="time"))+facet_wrap(~time,ncol = 1)
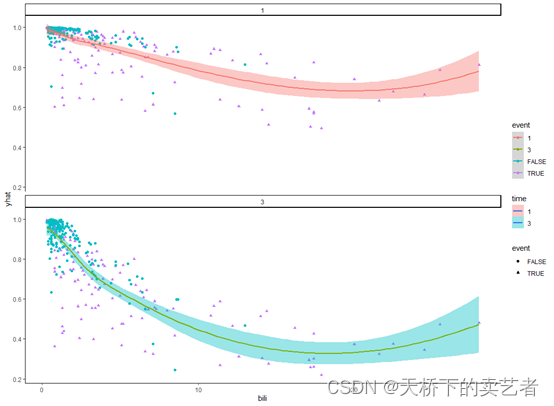
上图表明胆红素超过20后,随着胆红素增加存活率上升。
部分依赖图(Partial Dependence Plot)显示了一个或两个特征对机器学习模型的预测结果的边际效应,由于机器学习算法非参数的特性使得部份依赖图可以揭示线性以及非线性特征,容易理解并且有较高的解释力。但是对于生存数据,我们还要考虑时间的影响,
咱们可以使用parallel包的mclapply函数,结合plot.variable函数来处理时间数据,我们先定义要观察的变量和3个时间节点(1年,3年和5年)
xvar <- c("bili", "albumin", "copper", "prothrombin", "age", "edema")
time_index <- c(which(rfsrc_pbc$time.interest > 1)[1]-1,which(rfsrc_pbc$time.interest > 3)[1]-1,which(rfsrc_pbc$time.interest > 5)[1]-1)
time_index装有3个时间点数据,下面导入包来分析mclapply函数类似于平时咱们的lapply函数,就是对多个时间点使用plot.variable函数来跑循环
library(parallel)
partial_pbc <- mclapply(rfsrc_pbc$time.interest[time_index],function(tm){plot.variable(rfsrc_pbc, surv.type = "surv",time = tm, xvar.names = xvar,partial = TRUE ,show.plots = FALSE)})
时间点的预测值存在partial_pbc列表里面,3个数据代表3年

咱们把数据提取出来,咱们这里只提取1年和3年
gg_dta <- mclapply(partial_pbc, gg_partial)
pbc_ggpart <- combine.gg_partial(gg_dta[[1]], gg_dta[[2]],lbls = c("1 Year", "3 Years"))
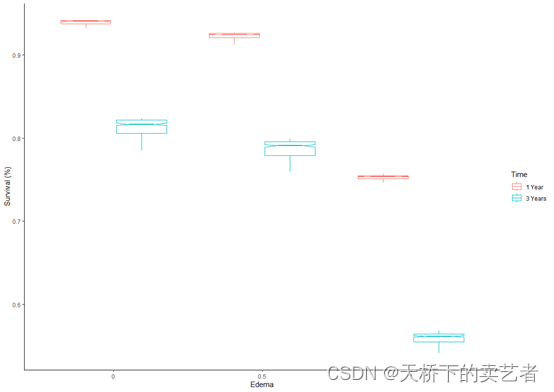
提取数据后就可以绘图了,先绘制一个箱线图
ggplot(pbc_ggpart[["edema"]], aes(y=yhat, x=edema, col=group))+geom_boxplot(notch = TRUE,outlier.shape = NA) + # panel=TRUE,labs(x = "Edema", y = "Survival (%)", color="Time", shape="Time") +theme_classic()
绘制时间依赖图(文章是这么叫的)
ggplot(pbc_ggpart[["bili"]], aes(y=yhat, x=bili, col=group))+geom_smooth() + # panel=TRUE,labs(x = "bili", y = "Survival (%)", color="Time", shape="Time") +theme(legend.position = c(0.1, 0.2))+theme_classic()
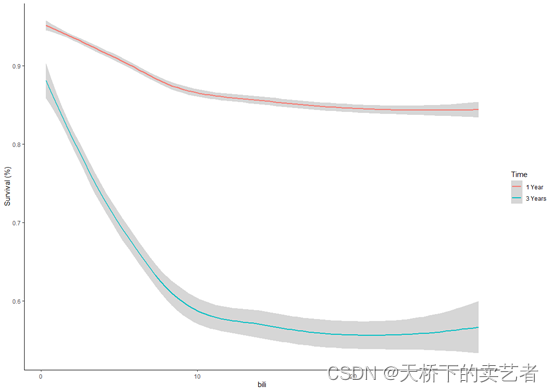
咱们可以看到和部分依赖图还是有点区别的,结论也不一样了。咱们也可以按我上面的方法从pbc_ggpart提取数据来自己绘制,有兴趣的可以试一下,这样更加灵活,更加好看。
接下来咱们来做下亚组的依赖关系,也就是亚组分析,亚组关系需要按年提取,咱们提取第一年的数据
ggvar<- gg_variable(rfsrc_pbc, time = 1)
咱们把edema改成分组的显示
ggvar$edema <- paste("edema = ", ggvar$edema, sep = "")
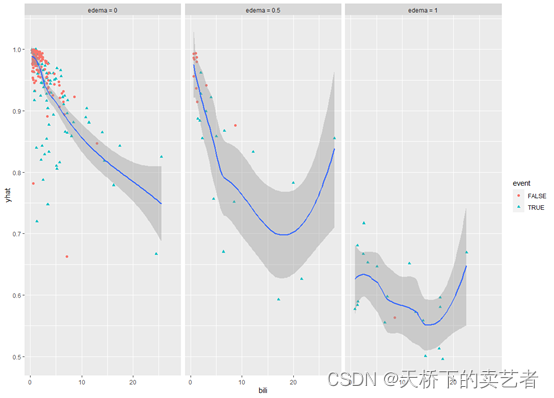
绘制亚组关系图,表明了每个亚组对生存结局的影响
ggplot(ggvar) + geom_point(aes_string(x = "bili", y = "yhat", color = "event", shape = "event"))+geom_smooth(aes_string(x = "bili", y = "yhat"))+facet_wrap(~edema)
咱们也可以对连续变量进行分组后在绘制
bili_cts <-quantile_pts(ggvar$bili, groups = 6, intervals = TRUE)
bili_cts[1] <- bili_cts[1] - 1.e-7 #我们需要移动最小值,以便包含该观察结果
##创建条件组并添加到gg_variable对象
bili_grp <- cut(ggvar$bili, breaks = bili_cts)
ggvar$bili_grp <- bili_grp
levels(ggvar$bili_grp) <- paste("bilirubin =", levels(bili_grp)) #调整面的命名
绘图
ggplot(ggvar) + geom_point(aes_string(x = "albumin", y = "yhat", color = "event", shape = "event"))+geom_smooth(aes_string(x = "albumin", y = "yhat"))+facet_wrap(~bili_grp)
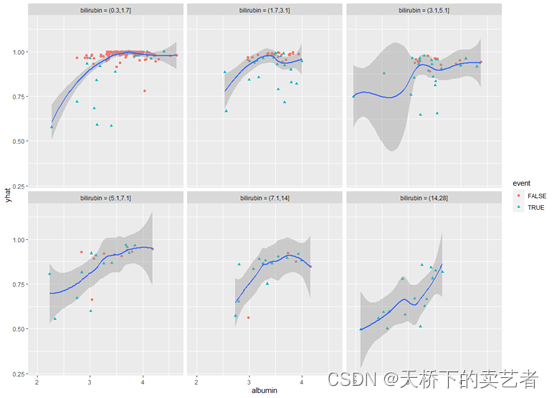
也可以使用gg_partial_coplot来绘制亚组的图,前面步骤是一样的,先生成分组变量
albumin_cts <-quantile_pts(ggvar$albumin, groups = 6, intervals = TRUE)
albumin_cts[1] <- albumin_cts[1] - 0.01 #我们需要移动最小值,以便包含该观察结果
##创建条件组并添加到gg_variable对象
albumin_grp <- cut(ggvar$albumin, breaks = albumin_cts)
ggvar$albumin_grp <- albumin_grp
使用g_partial_coplot生成绘图数据
coplotpbc <- gg_partial_coplot(rfsrc_pbc, xvar = "bili",groups = ggvar$albumin_grp,surv_type = "surv",time = rfsrc_pbc$time.interest[time_index[1]],show.plots = FALSE)
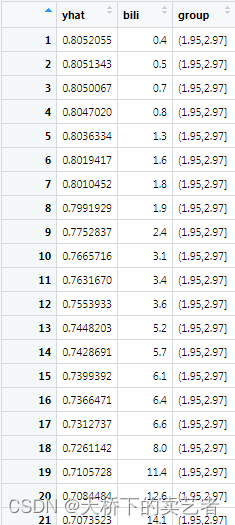
绘图
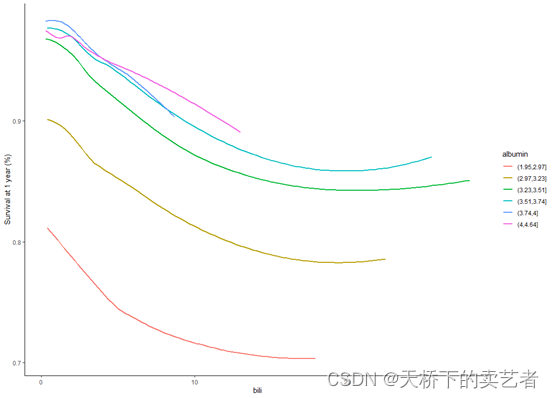
ggplot(coplotpbc, aes(x=bili, y=yhat, col=group, shape=group)) +geom_smooth(se = FALSE) +labs(x = "bili", y = "Survival at 1 year (%)",color = "albumin", shape = "albumin")+theme_classic()
除此之外还可以做决策曲线和roc曲线,这里就不弄了,我的既往文章都有。这两章内容比较多,代码我自己跑是没问题,但是怕有时候贴出来有时候会少贴一段,我把这两章代码进行了打包,公众号回复:随机森林生存分析代码,可以获得,回复要一模一样才行。