实验11 支持向量机
一、实验目的
1:了解支持向量机的结构和原理。
2:应用支持向量机建立训练模型,对模型进行评估。
二、实验内容和要求
【实验内容】
选择支持向量机,对花卉图像或玉米果穗图像进行分类。花卉图像包括玫瑰,向日葵和蒲公英;玉米果穗图像包括虫蛀,损伤,发霉。
(1)可针对这些特点对图像的颜色、纹理、形状进行分析,从而进行特征选择与提取;
(2)也可以尝试不提取特征,直接将图像作为输入,对比效果;
(3)调整人工神经网络的参数,观察不同的参数对分类结果的影响;
(4)实现对花卉图像的分类。
【实验要求】
1:完成对花卉数据集的分类任务。
2:调整不同的参数,通过对分类结果的对比,选取合适的参数。
3:使用不同的样本数量,查看是否对分类结果产生影响。可对样本进行扩增(例如:反转、平移、缩放、亮度变化)。
4:进行人工神经网络和支持向量机的方法对比。
三、实验结果与分析
1:基于sklearn依赖的SVM多分类
【1】代码任务解析:
(1)初始化数据集路径和分类标签

(2)读取数据集图像信息

(3)将图像提取的像素转成numpy数组

(4)按照8:2的比例划分训练集和测试集

(5)调用sklearn中的SVM-Classifier,实例化模型

(6)输入训练集进行训练,并输出训练集的准确率

(7)输入测试集进行预测,并输出测试集的准确率

【2】程序输出结果:

上述程序打印了SVM多分类的训练集准确率和测试集准确率,在花卉数据集上的结果如下图所示。

在玉米数据集上的结果如下图所示。
综上所述,在花卉数据集上SVM的训练集准确率是1.00、测试集准确率是0.83;在玉米数据集上SVM的训练集准确率是1.00、测试集准确率是0.50。
2:基于数学原理手动实现的SVM多分类代码
【1】代码任务解析:
(1)构建SimpleSVM类,定义初始化函数init、训练函数fit(包含损失曲线的绘制)、预测函数predict,各个函数的代码如下图所示。
init函数:

fit函数:

predict函数:

(2)读取数据集图像信息
(3)将图像提取的像素转成numpy数组
(4)按照8:2的比例划分训练集和测试集
(5)调用SimpleSVM类,实例化模型![]()
(6)输入训练集进行训练,并输出训练集的准确率
(7)输入测试集进行预测,并输出测试集的准确率
(8)从测试集中随机抽样,输出真实标签和预测标签

【2】程序输出结果:
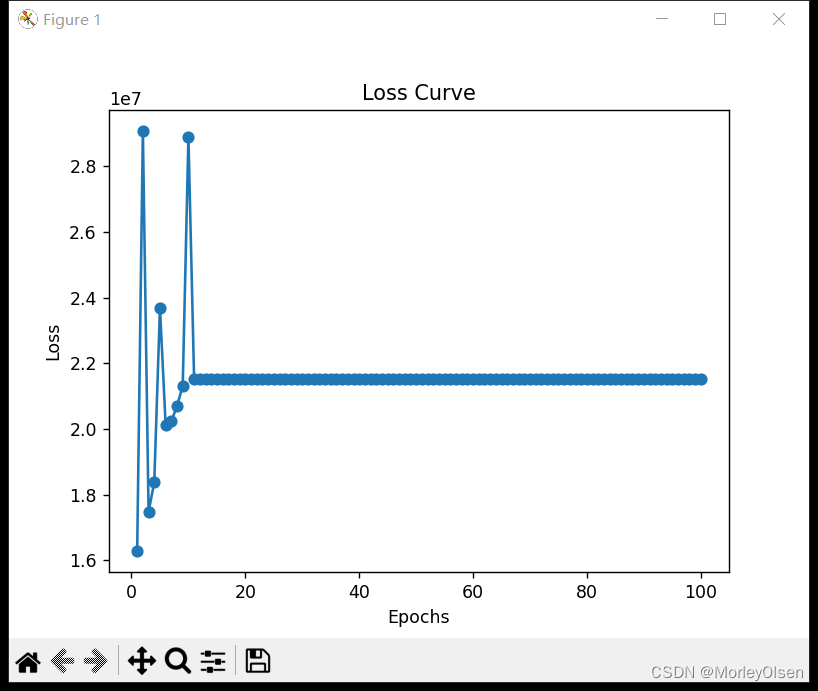
上述程序在训练过程中跟踪了模型的损失,并绘制出了损失曲线。在花卉数据集上的结果如下图所示。可以发现,随着迭代次数epoch的增加,模型的损失loss在逐渐稳定。
SVM模型训练完成后,计算了训练集准确率和测试集准确率,结果如下图所示。

综上所述,在花卉数据集上SVM的训练集准确率是1.00、测试集准确率是0.67。
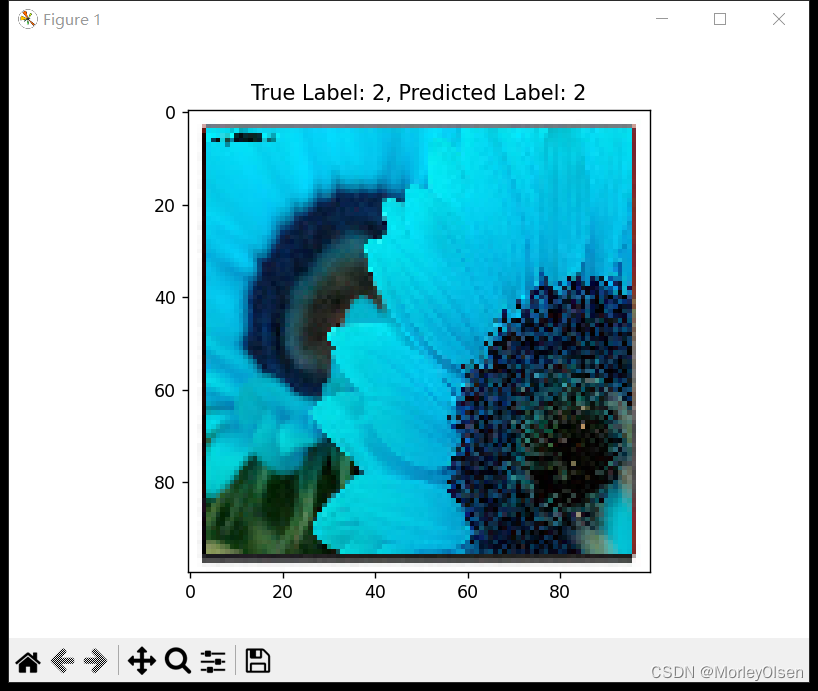
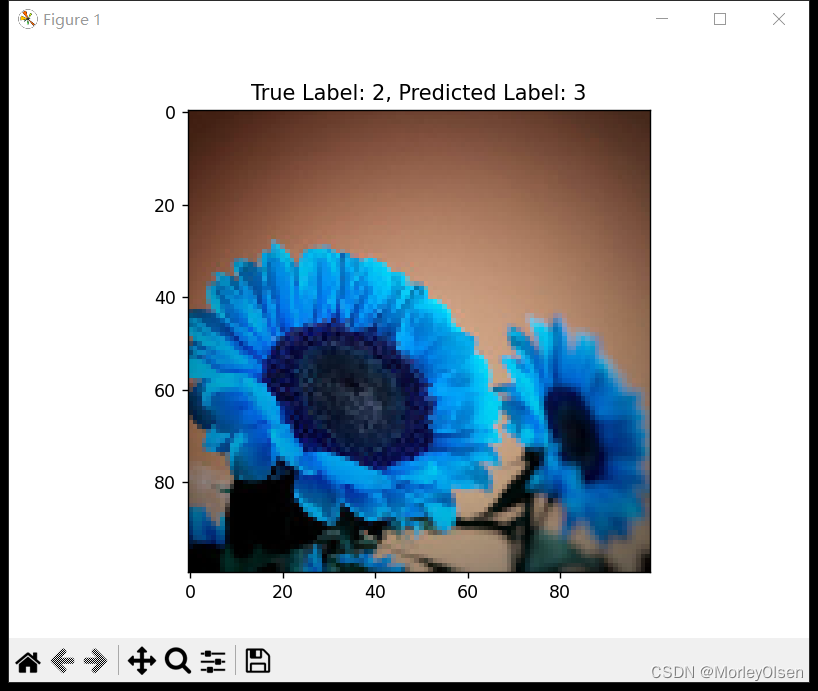
同时,上述程序中展示了模型的预测性能,对5张测试集图像进行了抽样展示,结果如下图所示。
【测试1:真实标签——2,预测标签——2,二者相同】

【测试2:真实标签——1,预测标签——1,二者相同】

【测试3:真实标签——3,预测标签——3,二者相同】

【测试4:真实标签——2,预测标签——2,二者相同】
【测试5:真实标签——2,预测标签——3,二者不相同】
3:进行人工神经网络和支持向量机的方法对比
在这两次的实验过程中,从训练过程来看,SVM的训练速度明显优于ANN的训练速度,且SVM的模型参数明显少于ANN的模型参数。从训练结果来看,SVM的准确率明显高于ANN的准确率。
同时,当ANN在训练集上表现出较高的准确率时,往往会在测试集上表现不佳,这表明模型存在过拟合问题。而支持向量机通常具有更好的泛化能力,对过拟合的抵抗能力较强。
综上所述,两种方法的对比总结如下。
(1)学习方式:
ANN:人工神经网络是一种基于神经元模型的机器学习方法,通过前向传播和反向传播来学习权重和参数,是一种端到端的学习方法,可以适应各种不同的任务。
SVM:支持向量机是一种监督学习方法,它寻找一个最优的超平面来分隔不同类别的数据点,是一种判别式学习方法,主要用于分类问题。
(2)可解释性:
ANN:神经网络通常被认为是黑盒模型,难以解释其内部的决策过程。
SVM:支持向量机的决策边界由支持向量确定,相对较容易解释和可视化。
(3)计算复杂度:
ANN:神经网络通常需要更多的计算资源和时间来训练。
SVM:支持向量机通常具有较低的计算复杂度,例如线性核情况。
(4)鲁棒性:
ANN:神经网络对于数据中的噪声和异常值较为敏感,需要大量数据来训练稳定的模型。
SVM:支持向量机对异常值有一定的鲁棒性,主要依赖于支持向量来确定决策边界。
(5)参数调整:
ANN:神经网络通常有很多参数需要调整,包括层数、神经元数量、学习率、激活函数等。
SVM:支持向量机的参数通常较少,主要是正则化参数C和核函数的选择。
4:基于实验指导的SVM模型实现(针对mnist手写数据集)
【1】代码任务解析:
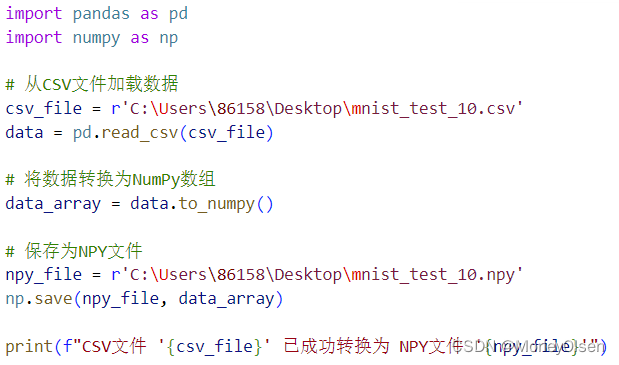
(1)将csv文件转化为npy文件

(2)修改SVM代码中main函数部
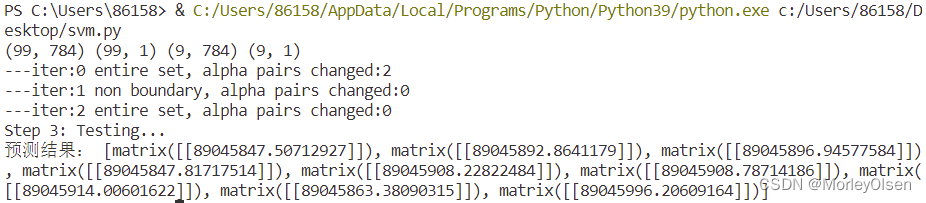
【2】程序输出结果:
四、遇到的问题和解决方法

问题1:在针对图像数据集运行实验指导中提供的代码时,实例化模型会出现以下的报错。
解决1:未解决,如果对矩阵进行flatten(平整化),则会继续出现dim不符合的报错。最后直接重新实现了新的代码,如SimpleSVM类所示。
五、实验总结
1:SVM是一种有监督学习算法,能够找到在高维空间中有效分隔不同类别的超平面,在高维问题中表现出色,适用于处理具有大量特征的数据集。SVM对异常值具有一定的鲁棒性,因为它主要依赖于支持向量来确定超平面,而不是全部数据点。同时,SVM有一些参数需要调整,如正则化参数C、核函数的类型和参数等。
2: SVM最初是用于二元分类问题的,但也可以通过一对多(One-vs-Rest,OvR)或一对一(One-vs-One,OvO)等策略扩展到多元分类问题。
3:最大间隔:能够最大化两个不同类别之间的间隔的超平面。最大间隔有助于提高模型的泛化性能。
4:SVM可以使用核函数来处理非线性可分的数据。常用的核函数包括线性核、多项式核和径向基函数(RBF)核。
六、程序源代码
1:基于sklearn依赖的SVM多分类代码
| import os import numpy as np import cv2 from sklearn.model_selection import train_test_split from sklearn.svm import SVC from sklearn.metrics import accuracy_score # 图像数据所在的根目录 data_root = r'C:\Users\86158\Desktop\dataset\data1' # 遍历每个类别的文件夹 class_labels = [] X = [] y = [] for class_label in os.listdir(data_root): class_path = os.path.join(data_root, class_label)
# 确保当前路径是一个目录 if os.path.isdir(class_path): class_labels.append(class_label)
# 分配一个唯一的整数标签给每个类别 label = len(class_labels) - 1
# 遍历当前类别的图像文件 for image_file in os.listdir(class_path): image_path = os.path.join(class_path, image_file)
# 使用OpenCV加载图像 image = cv2.imread(image_path)
# 调整图像大小为统一的尺寸 100x100 image = cv2.resize(image, (100, 100))
# 展平图像数据 image_flat = image.flatten()
# 将图像数据和标签添加到X和y中 X.append(image_flat) y.append(label) # 将图像数据转换为NumPy数组 X = np.array(X) y = np.array(y) # 数据集划分为训练集和测试集 X_train, X_test, y_train, y_test = train_test_split(X, y, test_size=0.2, random_state=42) # 创建SVM分类器 svm_classifier = SVC(kernel='linear', C=1.0, random_state=42) # 训练分类器 svm_classifier.fit(X_train, y_train) # 在训练集上进行预测 y_train_pred = svm_classifier.predict(X_train) # 计算训练集准确率 train_accuracy = accuracy_score(y_train, y_train_pred) print("训练集准确率:") print(f'Training Accuracy: {train_accuracy}') # 在测试集上进行预测 y_pred = svm_classifier.predict(X_test) # 计算准确率 accuracy = accuracy_score(y_test, y_pred) print("测试集准确率:") print(f'Accuracy: {accuracy}') |
2:基于数学原理手动实现的SVM多分类代码
| import numpy as np import os import cv2 from sklearn.model_selection import train_test_split from sklearn.metrics import accuracy_score import matplotlib.pyplot as plt import random class SimpleSVM: def __init__(self): self.weights = None self.classes = None def fit(self, X, y, epochs=100, lr=0.001, lambda_param=0.01): self.classes = np.unique(y) n_samples, n_features = X.shape n_classes = len(self.classes) # 初始化权重 self.weights = np.zeros((n_classes, n_features)) # 训练过程 for _ in range(epochs): for idx, x_i in enumerate(X): class_index = np.where(self.classes == y[idx])[0][0] for c in range(n_classes): if c == class_index: continue margin = 1 - np.dot(x_i, self.weights[class_index]) + np.dot(x_i, self.weights[c]) if margin > 0: self.weights[class_index] -= lr * (-2 * lambda_param * self.weights[class_index] - x_i) self.weights[c] -= lr * (-2 * lambda_param * self.weights[c] + x_i) def predict(self, X): predictions = np.dot(X, self.weights.T) return self.classes[np.argmax(predictions, axis=1)]
# 重新定义fit函数,在训练过程中输出loss def fit(self, X, y, epochs=100, lr=0.001, lambda_param=0.01): self.classes = np.unique(y) n_samples, n_features = X.shape n_classes = len(self.classes) # 初始化权重 self.weights = np.zeros((n_classes, n_features))
# 用于存储损失值 losses = [] # 训练过程 for epoch in range(epochs): for idx, x_i in enumerate(X): class_index = np.where(self.classes == y[idx])[0][0] for c in range(n_classes): if c == class_index: continue margin = 1 - np.dot(x_i, self.weights[class_index]) + np.dot(x_i, self.weights[c]) if margin > 0: self.weights[class_index] -= lr * (-2 * lambda_param * self.weights[class_index] - x_i) self.weights[c] -= lr * (-2 * lambda_param * self.weights[c] + x_i)
# 计算损失并添加到列表中 loss = 0.5 * np.sum(self.weights ** 2) + np.sum(np.maximum(0, 1 - np.dot(X, self.weights.T))) losses.append(loss)
# 绘制损失曲线 plt.plot(range(1, epochs + 1), losses, marker='o') plt.xlabel('Epochs') plt.ylabel('Loss') plt.title('Loss Curve') plt.show() data_root = r'C:\Users\86158\Desktop\dataset\data1' # 遍历每个类别的文件夹 class_labels = [] X = [] y = [] for class_label in os.listdir(data_root): class_path = os.path.join(data_root, class_label)
# 确保当前路径是一个目录 if os.path.isdir(class_path): class_labels.append(class_label) label = len(class_labels) - 1 # 分配一个唯一的整数标签给每个类别
# 遍历当前类别的图像文件 for image_file in os.listdir(class_path): image_path = os.path.join(class_path, image_file)
# 使用OpenCV加载图像 image = cv2.imread(image_path)
# 调整图像大小为统一的尺寸 100x100 image = cv2.resize(image, (100, 100))
# 展平图像数据 image_flat = image.flatten()
# 将图像数据和标签添加到X和y中 X.append(image_flat) y.append(label) # 将图像数据转换为NumPy数组 X = np.array(X) y = np.array(y) # 数据集划分为训练集和测试集 X_train, X_test, y_train, y_test = train_test_split(X, y, test_size=0.2, random_state=42) model = SimpleSVM() model.fit(X_train,y_train) y_pred = model.predict(X_test) # 计算准确率 accuracy = accuracy_score(y_test, y_pred) print(f'Accuracy: {accuracy}') # 从测试集中随机选择5个样本 sample_indices = random.sample(range(len(X_test)), 5) for idx in sample_indices: sample_image = X_test[idx].reshape(100, 100, 3) true_label = class_labels[y_test[idx]] predicted_label = class_labels[y_pred[idx]] plt.imshow(sample_image) plt.title(f'True Label: {true_label}, Predicted Label: {predicted_label}') plt.show() |
3:实验指导中的参考代码(修改后)
| from numpy import * import numpy as np import random # calulate kernel value def calcKernelValue(matrix_x, sample_x, kernelOption): kernelType = kernelOption[0] numSamples = matrix_x.shape[0] kernelValue = mat(zeros((numSamples, 1))) if kernelType == 'linear': kernelValue = matrix_x * sample_x.T elif kernelType == 'rbf': sigma = kernelOption[1] if sigma == 0: sigma = 1.0 for i in range(numSamples): diff = matrix_x[i, :] - sample_x kernelValue[i] = exp(diff * diff.T / (-2.0 * sigma ** 2)) else: raise NameError('Not support kernel type! You can use linear or rbf!') return kernelValue # calculate kernel matrix given train set and kernel type def calcKernelMatrix(train_x, kernelOption): numSamples = train_x.shape[0] kernelMatrix = mat(zeros((numSamples, numSamples))) for i in range(numSamples): kernelMatrix[:, i] = calcKernelValue(train_x, train_x[i, :], kernelOption) return kernelMatrix # define a struct just for storing variables and data class SVMStruct: def __init__(self, dataSet, labels, C, toler, kernelOption): self.train_x = dataSet self.train_y = labels self.C = C self.toler = toler self.numSamples = dataSet.shape[0] self.alphas = mat(zeros((self.numSamples, 1))) self.b = 0 self.errorCache = mat(zeros((self.numSamples, 2))) self.kernelOpt = kernelOption self.kernelMat = calcKernelMatrix(self.train_x, self.kernelOpt) # calculate the error for alpha k def calcError(svm, alpha_k): output_k = float(multiply(svm.alphas, svm.train_y).T * svm.kernelMat[:, alpha_k] + svm.b) error_k = output_k - float(svm.train_y[alpha_k]) return error_k # update the error cache for alpha k after optimize alpha k def updateError(svm, alpha_k): error = calcError(svm, alpha_k) svm.errorCache[alpha_k] = [1, error] # select alpha j which has the biggest step def selectAlpha_j(svm, alpha_i, error_i): svm.errorCache[alpha_i] = [1, error_i] candidateAlphaList = nonzero(svm.errorCache[:, 0].A)[0] maxStep = 0 alpha_j = 0 error_j = 0 if len(candidateAlphaList) > 1: for alpha_k in candidateAlphaList: if alpha_k == alpha_i: continue error_k = calcError(svm, alpha_k) if abs(error_k - error_i) > maxStep: maxStep = abs(error_k - error_i) alpha_j = alpha_k error_j = error_k else: alpha_j = alpha_i while alpha_j == alpha_i: alpha_j = int(random.uniform(0, svm.numSamples)) error_j = calcError(svm, alpha_j) return alpha_j, error_j # the inner loop for optimizing alpha i and alpha j def innerLoop(svm, alpha_i): error_i = calcError(svm, alpha_i) if (svm.train_y[alpha_i] * error_i < -svm.toler) and (svm.alphas[alpha_i] < svm.C) or ( svm.train_y[alpha_i] * error_i > svm.toler) and (svm.alphas[alpha_i] > 0): alpha_j, error_j = selectAlpha_j(svm, alpha_i, error_i) alpha_i_old = svm.alphas[alpha_i].copy() alpha_j_old = svm.alphas[alpha_j].copy() if svm.train_y[alpha_i] != svm.train_y[alpha_j]: L = max(0, svm.alphas[alpha_j] - svm.alphas[alpha_i]) H = min(svm.C, svm.C + svm.alphas[alpha_j] - svm.alphas[alpha_i]) else: L = max(0, svm.alphas[alpha_j] + svm.alphas[alpha_i] - svm.C) H = min(svm.C, svm.alphas[alpha_j] + svm.alphas[alpha_i]) if L == H: return 0 eta = 2.0 * svm.kernelMat[alpha_i, alpha_j] - svm.kernelMat[alpha_i, alpha_i] - svm.kernelMat[alpha_j, alpha_j] if eta >= 0: return 0 svm.alphas[alpha_j] -= svm.train_y[alpha_j] * (error_i - error_j) / eta if svm.alphas[alpha_j] > H: svm.alphas[alpha_j] = H if svm.alphas[alpha_j] < L: svm.alphas[alpha_j] = L if abs(alpha_j_old - svm.alphas[alpha_j]) < 0.00001: updateError(svm, alpha_j) return 0 svm.alphas[alpha_i] += svm.train_y[alpha_i] * svm.train_y[alpha_j] * (alpha_j_old - svm.alphas[alpha_j]) b1 = svm.b - error_i - svm.train_y[alpha_i] * (svm.alphas[alpha_i] - alpha_i_old) * svm.kernelMat[ alpha_i, alpha_i] * svm.train_y[alpha_j] * (svm.alphas[alpha_j] - alpha_j_old) * svm.kernelMat[ alpha_i, alpha_j] b2 = svm.b - error_j - svm.train_y[alpha_i] * (svm.alphas[alpha_i] - alpha_i_old) * svm.kernelMat[ alpha_i, alpha_j] - svm.train_y[alpha_j] * (svm.alphas[alpha_j] - alpha_j_old) * svm.kernelMat[ alpha_j, alpha_j] if (0 < svm.alphas[alpha_i]) and (svm.alphas[alpha_i] < svm.C): svm.b = b1 elif (0 < svm.alphas[alpha_j]) and (svm.alphas[alpha_j] < svm.C): svm.b = b2 else: svm.b = (b1 + b2) / 2.0 updateError(svm, alpha_j) updateError(svm, alpha_i) return 1 else: return 0 # the main training procedure def trainSVM(train_x, train_y, C, toler, maxIter, kernelOption=('rbf', 1.0)): svm = SVMStruct(mat(train_x), mat(train_y), C, toler, kernelOption) entireSet = True alphaPairsChanged = 0 iterCount = 0 while (iterCount < maxIter) and ((alphaPairsChanged > 0) or entireSet): alphaPairsChanged = 0 if entireSet: for i in range(svm.numSamples): alphaPairsChanged += innerLoop(svm, i) print('---iter:%d entire set, alpha pairs changed:%d' % (iterCount, alphaPairsChanged)) iterCount += 1 else: nonBoundAlphasList = nonzero((svm.alphas.A > 0) * (svm.alphas.A < svm.C))[0] for i in nonBoundAlphasList: alphaPairsChanged += innerLoop(svm, i) print('---iter:%d non boundary, alpha pairs changed:%d' % (iterCount, alphaPairsChanged)) iterCount += 1 if entireSet: entireSet = False elif alphaPairsChanged == 0: entireSet = True return svm # testing your trained svm model given test set def testSVM(svm, test_x): test_x = mat(test_x) numTestSamples = test_x.shape[0] supportVectorsIndex = nonzero(svm.alphas.A > 0)[0] supportVectors = svm.train_x[supportVectorsIndex] supportVectorLabels = svm.train_y[supportVectorsIndex] supportVectorAlphas = svm.alphas[supportVectorsIndex] re_predict = [] for i in range(numTestSamples): kernelValue = calcKernelValue(supportVectors, test_x[i, :], svm.kernelOpt) predict = kernelValue.T * multiply(supportVectorLabels, supportVectorAlphas) + svm.b re_predict.append(predict) return re_predict # Standardize the data def Standar(data): mean = np.mean(data, axis=1).reshape(-1, 1) std = np.std(data, axis=1).reshape(-1, 1) another_trans_data = data - mean return another_trans_data / std if __name__ == '__main__': C, toler, maxIter = 0.6, 0.001, 40 train_data = np.load(r'C:\Users\86158\Desktop\mnist_train_100.npy') test_data = np.load(r'C:\Users\86158\Desktop\mnist_test_10.npy') train_x, y_train, test_x, y_test = train_data[:, 1:], train_data[:, 0].reshape(-1, 1), test_data[:, 1:], test_data[:,0].reshape(-1, 1) X_large_train = Standar(train_x) X_large_test = Standar(test_x) print(train_x.shape, y_train.shape, test_x.shape, y_test.shape) svmClassifier = trainSVM(X_large_train, y_train, C, toler, maxIter, kernelOption=('linear', 0)) print("Step 3: Testing...") predict = testSVM(svmClassifier, X_large_test) print("预测结果:", predict) |
4:csv文件转换为npy文件的代码
| import pandas as pd import numpy as np # 从CSV文件加载数据 csv_file = r'C:\Users\86158\Desktop\mnist_test_10.csv' data = pd.read_csv(csv_file) # 将数据转换为NumPy数组 data_array = data.to_numpy() # 保存为NPY文件 npy_file = r'C:\Users\86158\Desktop\mnist_test_10.npy' np.save(npy_file, data_array) print(f"CSV文件 '{csv_file}' 已成功转换为 NPY文件 '{npy_file}'") |