基本信息
- 标题:Segment anything in medical images
- 中文标题:分割一切医学图像
- 发表年份: 2024年1月
- 期刊/会议: Nature Communications
- 分区: SCI 1区
- IF:16.6
- 作者: Jun Ma; Bo Wang(一作;通讯)
- 单位:加拿大多伦多大学 健康网络中心
- DOI:https://doi.org/10.1038/s41467-024-44824-z
- 开源代码:https://github.com/bowang-lab/MedSAM
摘要: 医学图像分割是临床实践中的一个关键组成部分,有助于准确诊断、治疗计划和疾病监测。然而,现有的方法通常针对特定的模式或疾病类型,在各种医学图像分割任务中缺乏可推广性。在这里,我们介绍了MedSAM,这是一个基础模型,旨在通过实现通用医学图像分割来弥合这一差距。该模型是在大型医学图像数据集上开发的,有1570263对图像-掩码对,涵盖10种成像模式和30多种癌症类型。我们对86个内部验证任务和60个外部验证任务进行了全面评估,证明了比模态专家模型更好的准确性和稳健性。通过在广泛的任务中提供准确高效的分段,MedSAM在加快诊断工具的发展和治疗计划的个性化方面具有巨大潜力。
章节速览
- Introduction
-
- Results
- 1.1 MedSAM:快速医学图像分割的基础模型
- 1.2 定量和定性分析
- 1.2 训练数据集大小的影响
- 1.4 MedSAM提升标注效率
-
- Discussion
-
- Methods
- 3.1 数据集管理和预处理
- 3.2 网络架构
- 3.3 训练方案及实验设置
- 3.4 损失函数
- 3.5 人类注释
- 3.6 评估指标
- 3.7 统计分析
- 3.8 使用软件
- 3.9 报告总结
Introduction
医学图像分割领域对通用模型的需求日益增长:即一次训练后能够应用于广泛分割任务的模型。这样的模型不仅在模型容量方面表现出更高的多功能性,还有可能在不同任务中产生更加一致的结果。
然而,由于自然图像与医学图像之间存在显著差异,因此分割基础模型(例如 SAM)在医学图像分割领域的适用性仍然有限。SAM本质上是一种可提示的分割方法,需要使用点或边界框来指定分割目标。
许多研究已经将开箱即用的SAM模型应用于典型的医学图像分割任务和其他具有挑战性的场景。我们进一步介绍了MedSAM,这是一种改进的基础模型,可显着增强 SAM 在医学图像上的分割性能。MedSAM 通过在包含超过一百万对医学图像-掩模对的前所未有的数据集上微调 SAM 来实现这一目标。
1. Results
1.1 MedSAM:快速医学图像分割的基础模型
MedSAM 旨在发挥通用医学图像分割基础模型的作用。构建此类模型的一个关键方面是能够适应成像条件、解剖结构和病理条件的各种变化。为了应对这一挑战,我们策划了一个多样化的大规模医学图像分割数据集,其中包含 1,570,263 个医学图像掩模对,涵盖 10 种成像模式、30 多种癌症类型和多种成像协议
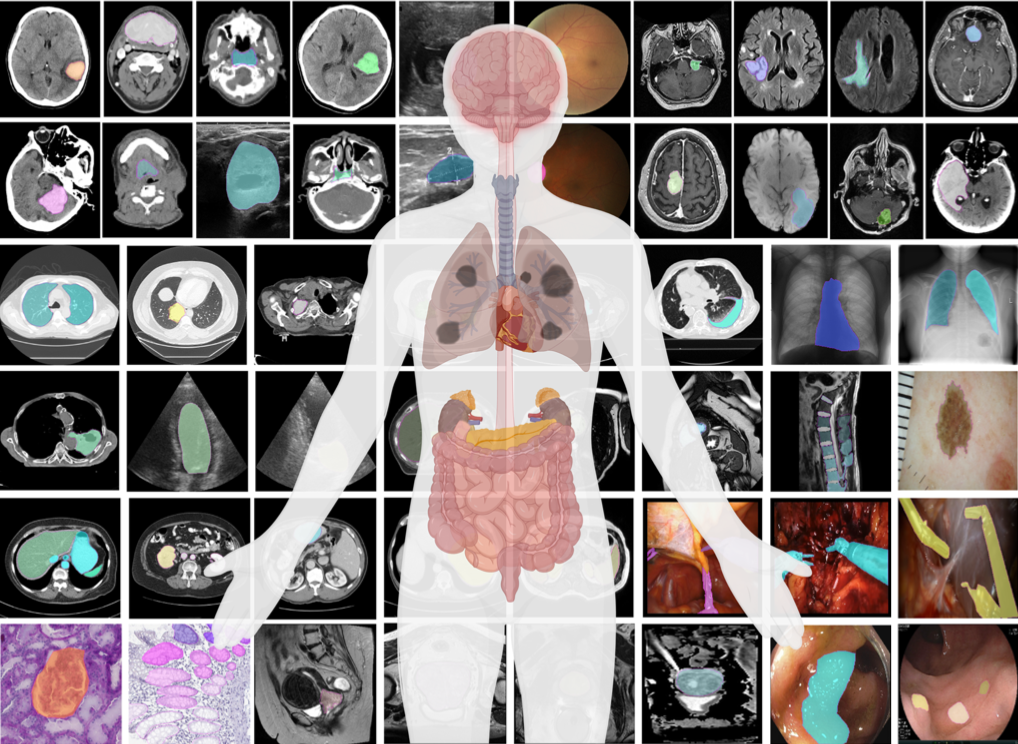
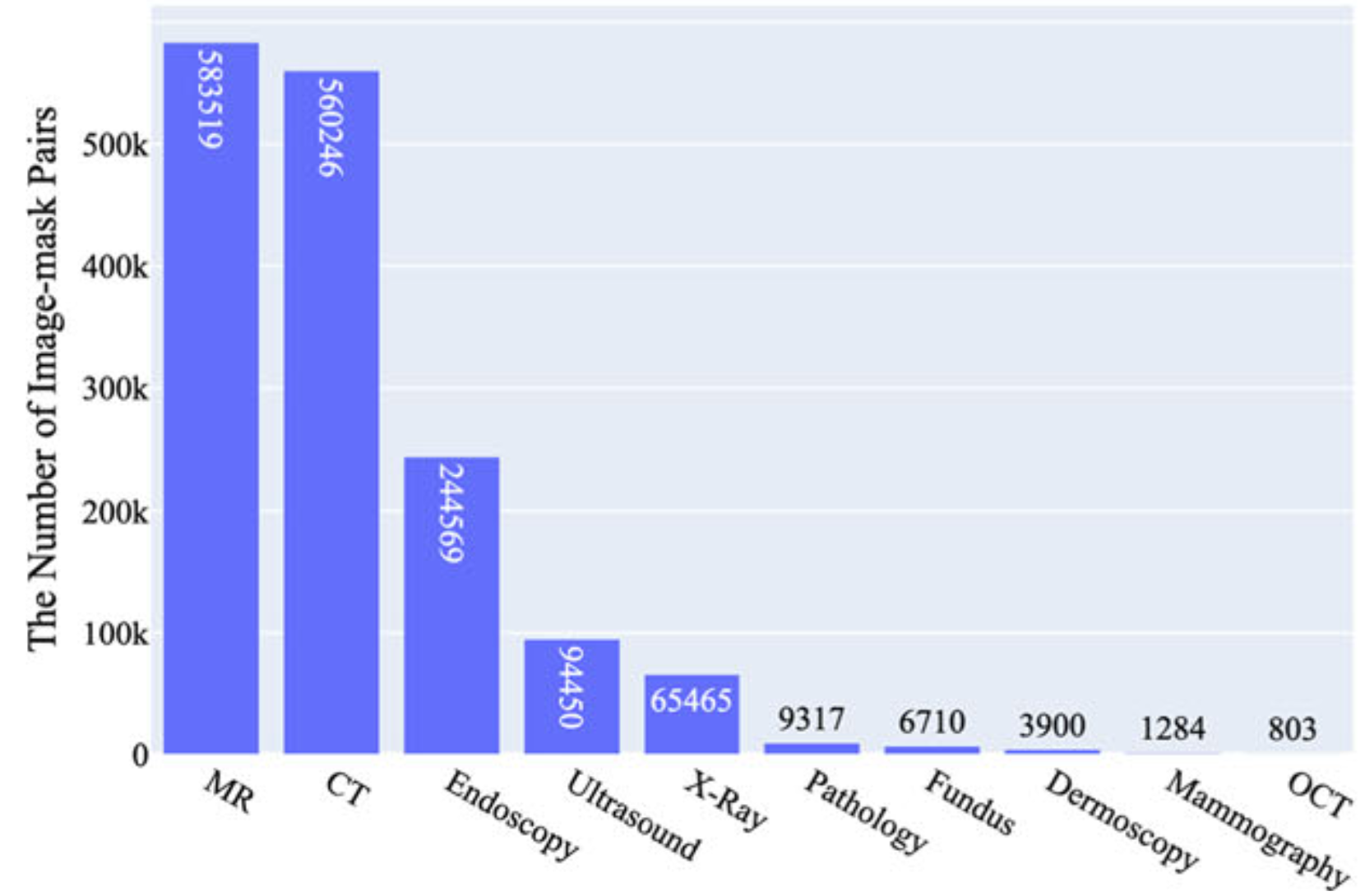
图 2a概述了数据集中不同医学成像模式的图像分布,按总数排序。很明显,计算机断层扫描(CT)、磁共振成像(MRI)和内窥镜检查是主要的检查方式,反映出它们在临床实践中的普遍性。
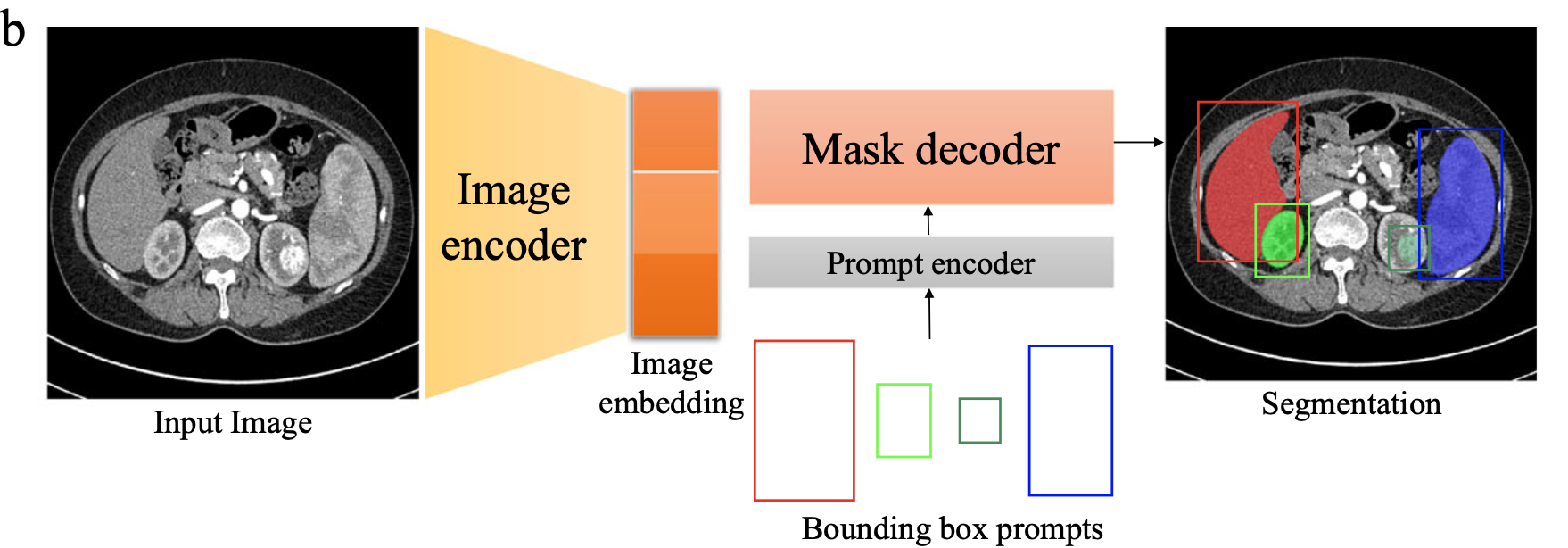
我们采用了SAM中的网络架构,包括图像编码器、提示编码器和掩模解码器(图2b)。图像编码器将输入图像映射到高维图像嵌入空间。提示编码器通过位置编码将用户绘制的边界框转换为特征表示。最后,掩模解码器使用交叉注意力(方法)将图像嵌入和提示特征融合在一起。
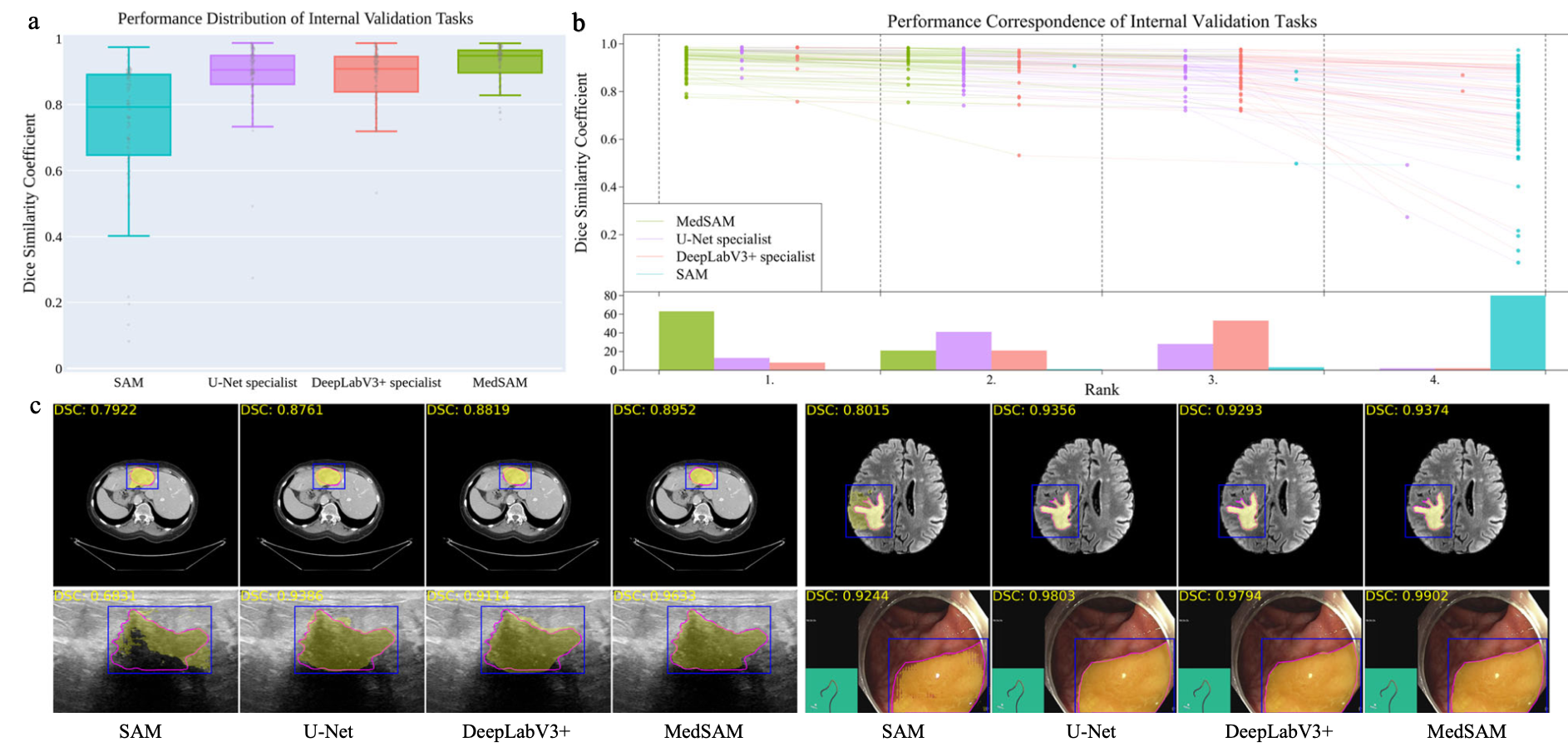
1.2 定量和定性分析
我们通过内部验证和外部验证评估了 MedSAM。内部验证包含 86 个分割任务。外部验证包括 60 个分割任务,所有这些任务要么来自新数据集,要么涉及看不见的分割目标
1.3 训练数据集大小的影响
我们还研究了不同数据集大小对 MedSAM 性能的影响,因为训练数据集大小已被证明对模型性能至关重要。我们还在两种不同的数据集大小上训练了 MedSAM:10K 和 100K 图像,并将它们的性能与默认的 MedSAM 模型进行了比较
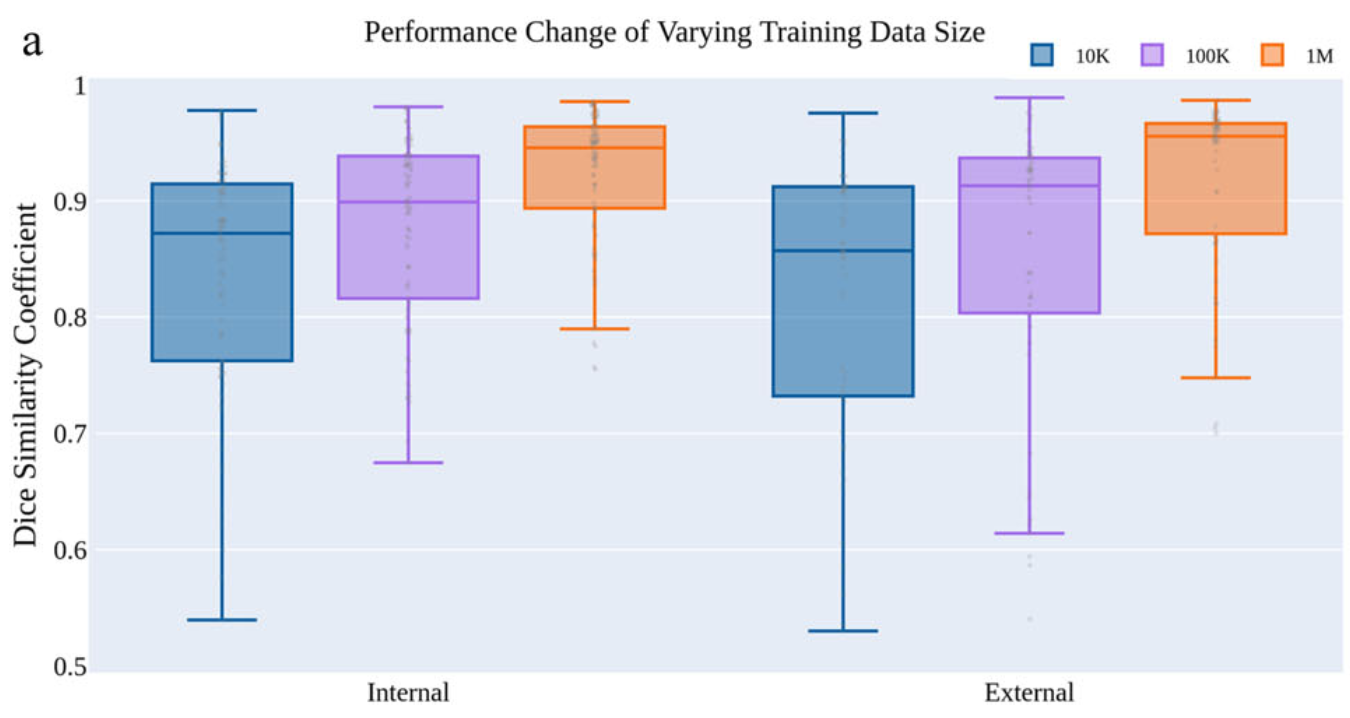
增加训练图像的数量显着提高了内部和外部验证集的性能
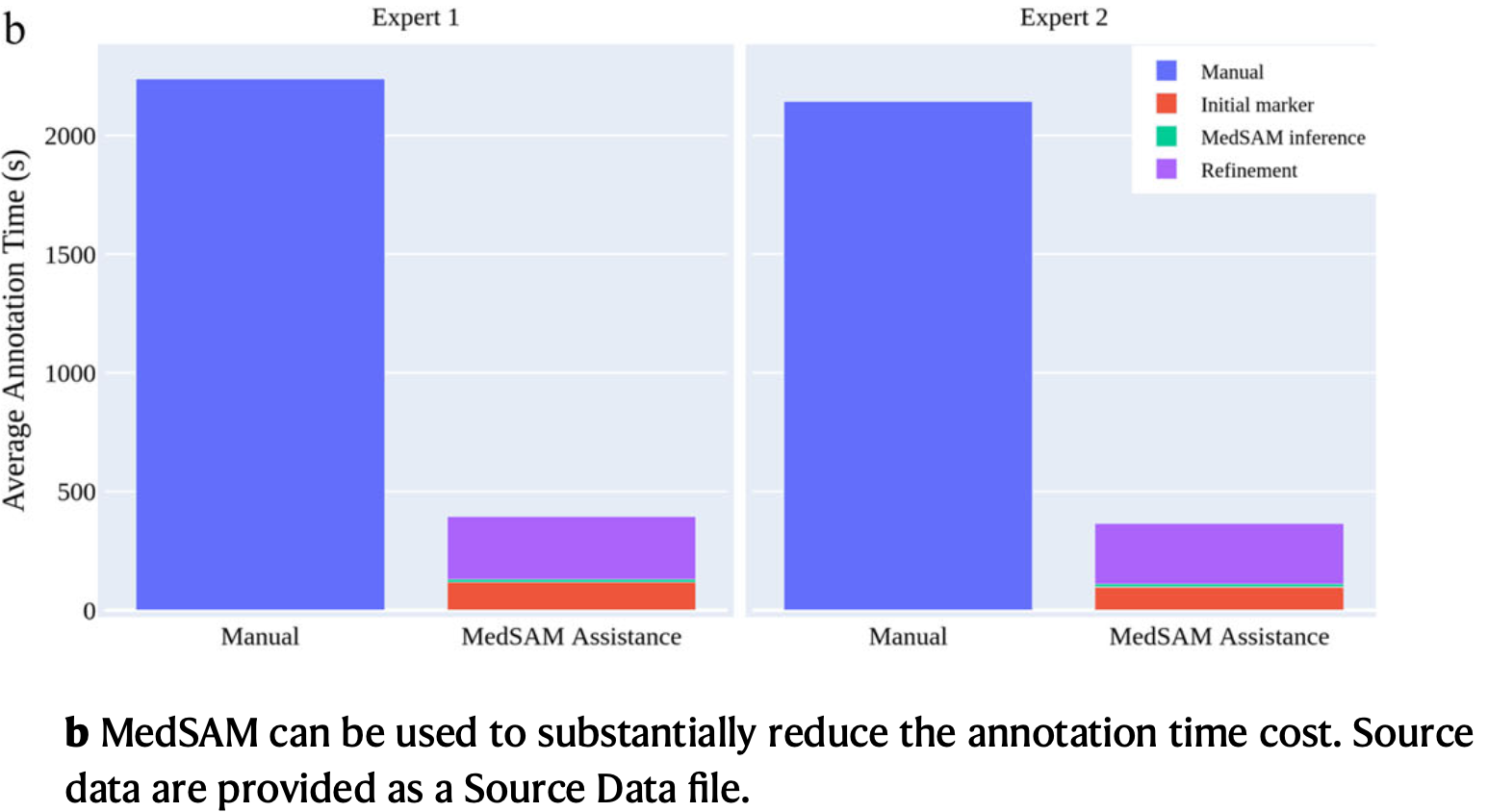
1.4 MedSAM提升标注效率
此外,我们进行了人工注释研究来评估两个管道的时间成本(方法)。对于第一个管道,两名人类专家以逐片方式手动注释 3D 肾上腺肿瘤。对于第二条流程,专家们首先每隔3-10片用线性标记(初始标记)绘制肿瘤长轴和短轴,这是肿瘤反应评估的常见做法。然后,使用 MedSAM 根据这些稀疏线性注释对肿瘤进行分割。结果表明,在 MedSAM 的帮助下,两位专家的注释时间分别大幅减少了 82.37% 和 82.95%
2. Discussion
尽管 MedSAM 拥有强大的功能,但它也存在一定的局限性。
- 训练集中的模态不平衡,其中 CT、MRI 和内窥镜图像在数据集中占主导地位。这可能会影响模型在较少代表性的模式(例如乳房X光检查)上的性能。
- 它在分割血管状分支结构方面存在困难,因为在此设置中边界框提示可能不明确。例如,在眼底图像中,动脉和静脉共享相同的边界框。
然而,这些限制并没有削弱 MedSAM 的实用性。由于 MedSAM 从大规模训练集中学习了丰富且有代表性的医学图像特征,因此可以对其进行微调,以有效地将新任务从代表性较低的模式或复杂的结构(如血管)中分割出来。
3. Methods
3.1 数据集管理和预处理
数据来源:这些数据集是从互联网上的各种来源获得的,包括TCIA、Kaggle、Grand-Challenge、Scientific Data、CodaLab 和MICCAI。
数据处理:所有3D 数据集(DICOM、nrrd 或 mhd 格式)都转换为广泛使用的 NifTI 格式。此外,灰度图像(例如 X 射线和超声)以及 RGB 图像(包括内窥镜检查、皮肤镜检查、眼底和病理图像)也被转换为 png 格式。
归一化方案
- CT图像归一化:对 Hounsfield 单位进行归一化。软组织、肺和脑所采用的窗口宽度和水平值分别为(W:400,L:40)、(W:1500,L:-160)和(W:80,L:40)。随后,强度值被重新调整到 [0, 255] 的范围。
- MR、X 射线、超声波、乳房 X 光检查和光学相干断层扫描 (OCT) 图像,我们将强度值剪裁到第 0.5 个百分位数和第 99.5 个百分位数之间的范围,然后将其重新缩放到 [0, 255] 范围。
- RGB图像(例如内窥镜、皮肤镜、眼底和病理图像),如果它们已经在[0, 255]的预期强度范围内,则它们的强度保持不变。但是,如果它们超出此范围,我们会利用最大-最小归一化将强度值重新调整为 [0, 255]。
图像尺寸
最后,为了满足模型的输入要求,将所有图像调整为统一大小1024×1024×3。对于全幻灯片病理图像,使用滑动窗口方法提取无重叠的斑块。位于边界上的斑块用 0 填充到该大小。对于 3D CT 和 MR 图像,每个 2D 切片的大小调整为 1024 × 1024,并且通道重复 3 次以保持一致性。其余的 2D 图像直接调整为 1024 × 1024 × 3。
3.2 网络架构
本研究中使用的网络是基于Transformer架构,该架构在自然语言处理和图像识别等各个领域展现出了显著的有效性。具体而言,该网络包括一个基于Vision Transformer(ViT)的图像编码器,负责提取图像特征;一个提示编码器,用于整合用户的交互(边界框);以及一个掩模解码器,利用图像嵌入、提示嵌入和输出令牌生成分割结果和置信度分数。
3.3 训练方案及实验设置
模型使用预先训练的 SAM 模型和 ViT-Base 模型进行初始化。损失函数是Dice损失和交叉熵损失之间的未加权总和。AdamW 优化器(β 1 = 0.9,β 2 = 0.999)进行优化,初始学习率为 1e-4,权重衰减为 0.01。全局批量大小为 160,未使用数据增强。该模型在 20 个 A100 (80G) GPU 上进行了 150 个 epoch 的训练,并选择最后一个检查点作为最终模型。
看到最后,20 个 A100,我欣慰了,并不是我脑子不够用才发不了这么好的文章



![[ai笔记3] ai春晚观后感-谈谈ai与艺术](https://img-blog.csdnimg.cn/img_convert/3f79d9ca4a9b800a1dfa88c5a5190ac6.png)

![[缓存] - Redis](https://img-blog.csdnimg.cn/direct/8319a8221102466c9a4afe72a7cb5882.png)




![[BeginCTF]真龙之力](https://img-blog.csdnimg.cn/direct/e591f36df6794dddb7122a61014c010e.png)