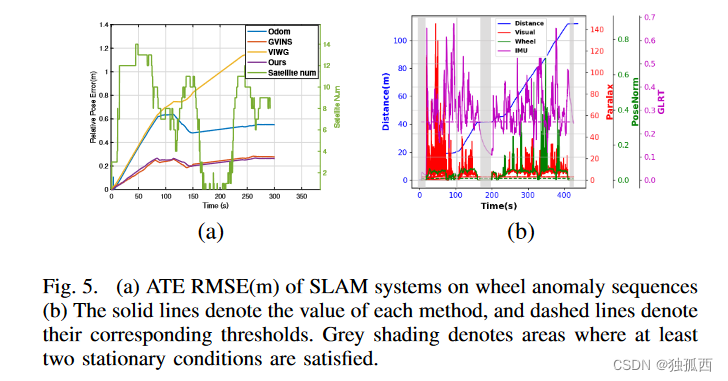
IOBR查看其收录的相关基因集(自备)_肿瘤 tme特征 iobr-CSDN博客
IOBR2:多维度解析肿瘤微环境 - 知乎 (zhihu.com)
学习手册:https://iobr.github.io/book/ (里面有详细教程)
系统综合的分析工具(Immuno-Oncology Biological Research 2,IOBR2)。IOBR2 提供六个基于多组学数据的 TME 分析模块。这些模块涵盖数据预处理、TME 估计、TME 渗透模式、细胞相互作用、基因组和 TME 相互作用、TME 相关特征的可视化以及基于关键特征的建模。IOBR2集成了多种重要的微环境分析算法和特征估计方法,简化了TME的分析和下游可视化。除了提供一种快速、简单的方法来从单细胞数据构建基因特征之外,IOBR2 还提供了一种从单细胞 RNAseq 构建 TME 反卷积参考矩阵的方法。分析流程和特征可视化是用户友好的,并提供复杂 TME 的全面描述,提供对肿瘤免疫相互作用的见解。提供了全面的 gitbook (https://iobr.github.io/book/),其中包含用户友好的手册和每个模块的完整分析工作流程。
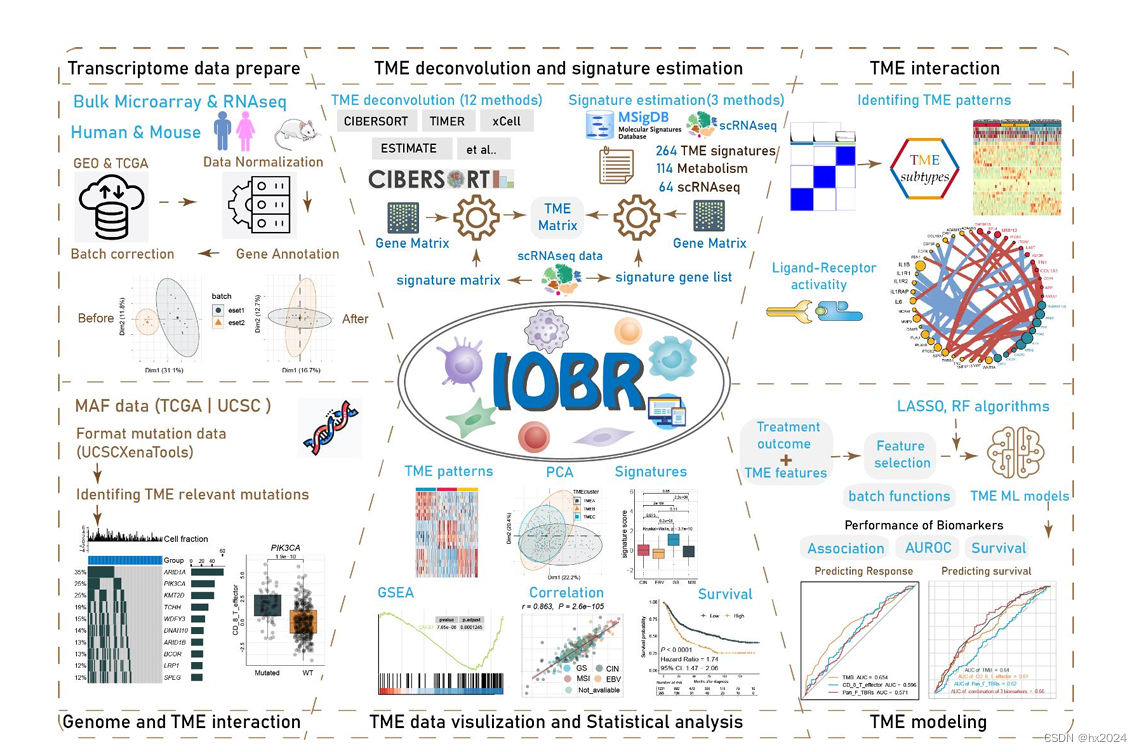
更新版好像比1增加了数据集分析和单细胞分析

安装:
依赖包:
if (!requireNamespace("BiocManager", quietly = TRUE)) install.packages("BiocManager")
depens<-c('tibble', 'survival', 'survminer', 'limma', "DESeq2","devtools", 'limSolve', 'GSVA', 'e1071', 'preprocessCore', "devtools", "tidyHeatmap", "caret", "glmnet", "ppcor", "timeROC", "pracma", "factoextra", "FactoMineR", "WGCNA", "patchwork", 'ggplot2', "biomaRt", 'ggpubr', "PMCMRplus")
for(i in 1:length(depens)){depen<-depens[i]if (!requireNamespace(depen, quietly = TRUE)) BiocManager::install(depen,update = FALSE)
}#安装
if (!requireNamespace("IOBR", quietly = TRUE)) devtools::install_github("IOBR/IOBR")library(IOBR)参考文献:
1:https://www.biorxiv.org/content/biorxiv/early/2024/01/15/2024.01.13.575484.full.pdf