| 欢迎浏览我的CSND博客! Blockbuater_drug …点击进入 |
|---|
文章目录
前言
本文是UCSF DOCK的使用案例分享,包括:
(1)配体、受体输入文件处理;
(2)分子对接;
(3)配体-受体相互作用中范德华与静电能量分解的指纹footprint 。
一、操作环境
操作环境:Ubuntu 22.04
软件版本:UCSF DOCK 6.11,安装可以参考这篇博文;UCSF Chimera 1.17.3,UCSF ChimeraX 1.7.1,安装可以参考这篇博文。
二、研究背景
NX-2127是一款具有双重活性的口服小分子靶向蛋白降解剂,可以布鲁顿氏酪氨酸激酶(BTK)蛋白。在与BTK结合的同时,NX-2127还可以招募E3泛素连接酶,使BTK蛋白“泛素化”,从而BTK被蛋白酶体降解。
BTK抑制剂作为抗肿瘤药物和自身免疫疾病靶点的有效性已在临床上得到了验证,目前已有多款BTK抑制剂获批上市。
通过分子对接可以研究小分子药物作为配体与药物靶点的作用模式;以分子对接为基础的虚拟筛选可以帮助从大型化合物库中筛选出与靶点蛋白有结合潜力的小分子。
本文通过BTK蛋白靶点与其固有配体NX-2127的对接研究展示DOCK对接程序的可行新,通过虚拟筛选ZINC库,找到有潜在结合力的新分子,可作为药物研究的起点。
三、受体-配体结构文件准备
本次介绍的处理好的结构文件,可以从这里下载:UCSF DOCK 分子对接详细案例(01)输入文件.
3.1准备文件夹DOCK_workdir, 下载晶体结构
mkdir DOCK_workdir && cd DOCK_workdir
mkdir 000_files 001_structure 002_surface_spheres 003_gridbox 004_min_rigid_dock 0041_fixed_dock 0042_flex_dock 0043_footprint
cd mkdir 000_files
3.1.1 来自湿实验的受体配体共晶结构:
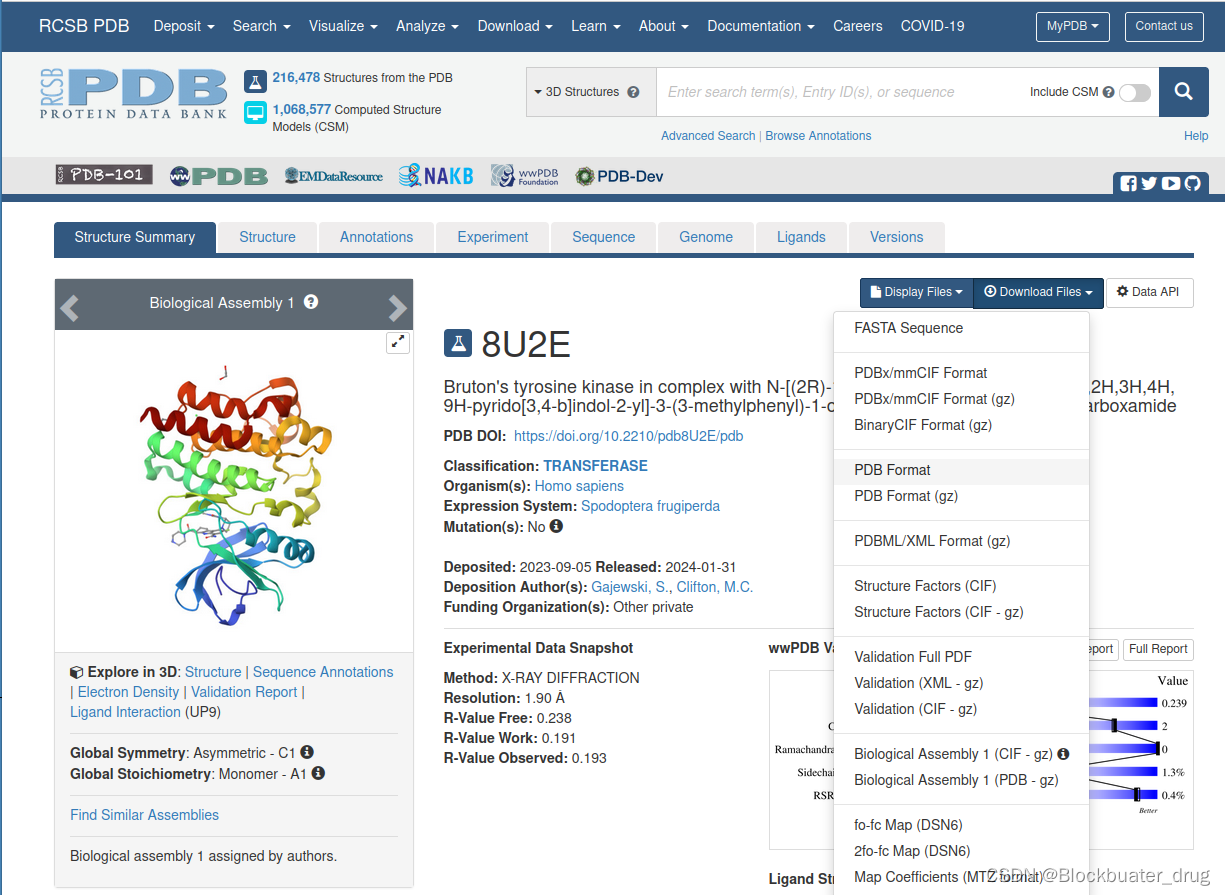
pdbid 8U2E,NX-2127与BTK的X-ray共晶结构此前得到解析,配体NX-2127在PDB结构中的编号为up9。
RCSB下载页面在这里。
选择下载PDB Format和 FASTA sequence。共晶结构PDB文件:8u2e.pdb; 序列FASTA文件:rcsb_pdb_8U2E.fasta
3.1.2 来自深度学习和语言模型推理预测的蛋白结构:
ChimeraX提供AlphaFold与ESMFold在线预测链接,在ChimeraX的界面选择:Tools -> Structure Prediction -> Alphafold和Tools -> Structure Prediction -> ESMFold,可提供输入序列的窗口,打开加载rcsb_pdb_8U2E.fasta,选择predict,结束后选择fetch,即可下载到ChimeraX窗口展示,使用方便。
很不幸目前ESMFold已无法提供服务,AlphaFold往往出现网络不畅而罢工。
关于蛋白结构预测,笔者此前介绍了AlphaFold与ESMFold本地便捷安装与使用体验,感兴趣可参考:
AlphaFold2.3 conda版本详细安装与使用
ESMFold conda安装、使用及与AlphaFold的简单比较
以及预测结果质量如何判别的介绍:蛋白结构预测模型评价指标
预测的结构是一个不带配体的apo结构,需要进一步优化,确定结合位点,通过分子对接以及分子动力学方法获得受体-配体复合物,然后可以使用DOCK在结合配体的位置做分子对接。
3.2 配体、受体文件准备
Chimera和ChimeraX均可以处理受体文件,去溶剂、补全可能缺失的loop、质子化、加电荷。可以参考相关博文。
以8u2e.pdb处理好的配体、受体文件存储在001_structure文件夹, up9是pdb结构中配体的编号;
DOCK中固有配体、受体结构均采用Mol2文件,分别需要加氢和不加氢的两种状态结构,包括dms文件一共5个:

四、sphere文件准备
DOCK采用sphere代表受体上可对接配体的区域,使用命令sphgen传入设置好的参数,即可得到所有可能的sphere。
cd ../002_surface_spherescp ../001_structure/8u2e_rec.dms ./
新建文件INSPH,输入以下参数: (4.0和1.4分别代表sphere球的最大半径和最小半径,8u2e_binding_spheres.sph 为输出文件的名字。
8u2e_rec.dms
R
X
0
4.0
1.4
8u2e_binding_spheres.sph
输入命令:
sphgen -i INSPH -o OUTSPH
用Chimera查看结果,显示配体结合区域(洋红色)很好地被识别。
chimera ../001_structure/8u2e_rec_withH.mol2 ../001_structure/up9_rec_withH.mol2 ./8u2e_binding_spheres.sph
 通过sphere_selector命令,选择性的找到配体区域的sphere,用法:
通过sphere_selector命令,选择性的找到配体区域的sphere,用法:
USAGE: sphere_selector [sphere输出文件] [ligand mol2 文件] [配体周围范围,A]
输入以下:
sphere_selector 8u2e_binding_spheres.sph ../001_structure/up9_rec_withH.mol2 10.0
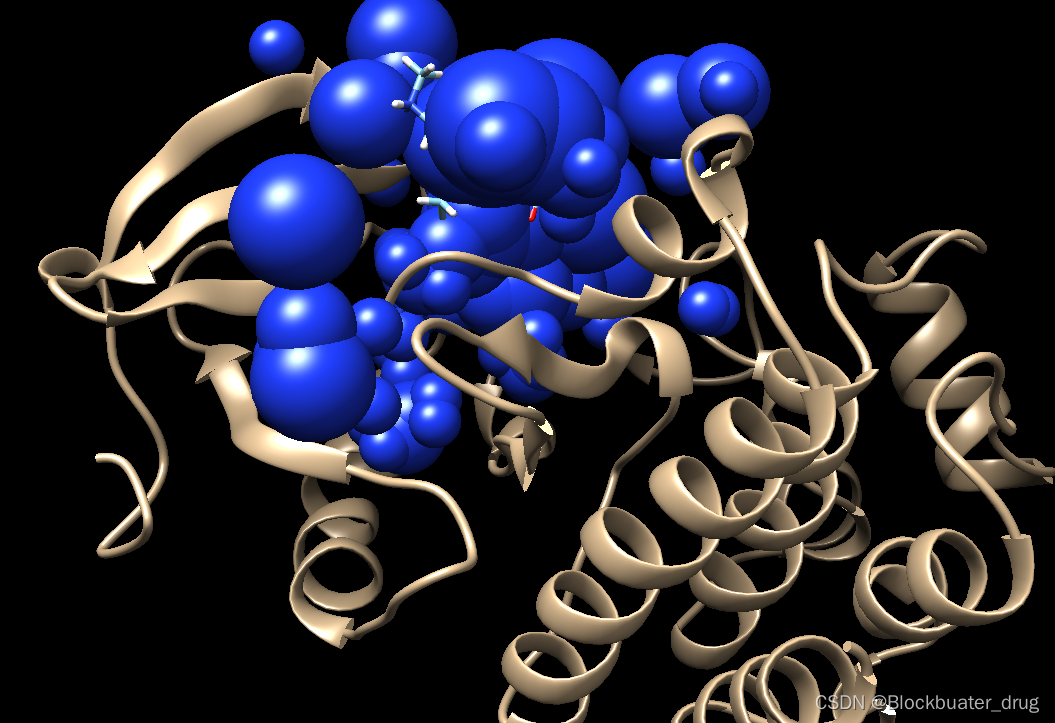
将受体8u2e_rec_withH.mol2文件,配体文件up9_rec_withH.mol2和selected_spheres.sph加载进Chimera,Terminal中输入以下:
chimera ../001_structure/8u2e_rec_withH.mol2 ../001_structure/up9_rec_withH.mol2 selected_spheres.sph
展示如下, 可以看到spheres充分占据了和代表了配体结合的位置,后续对接位点就在这里。
10.0的数值根据需要可以调整,对接范围直接影响虚拟筛选的速度和结果,应该根据研究需要,配合sphere半径最大值,最小值合理设置,锁定预期的对接位点。
五、 对接盒子与格点确定 Box and Grid
5.1 产生box
DOCK采用格点划分空间,以此来搜索和计算。对接前需要生成格点文件和设定box范围。
切换到003_gridbox目录,新建盒子确定命令参数的文件showbox.in。
cd ../003_gridbox
showbox.in内容:
Y
8.0
../002_surface_spheres/selected_spheres.sph
1
8u2e.box.pdb
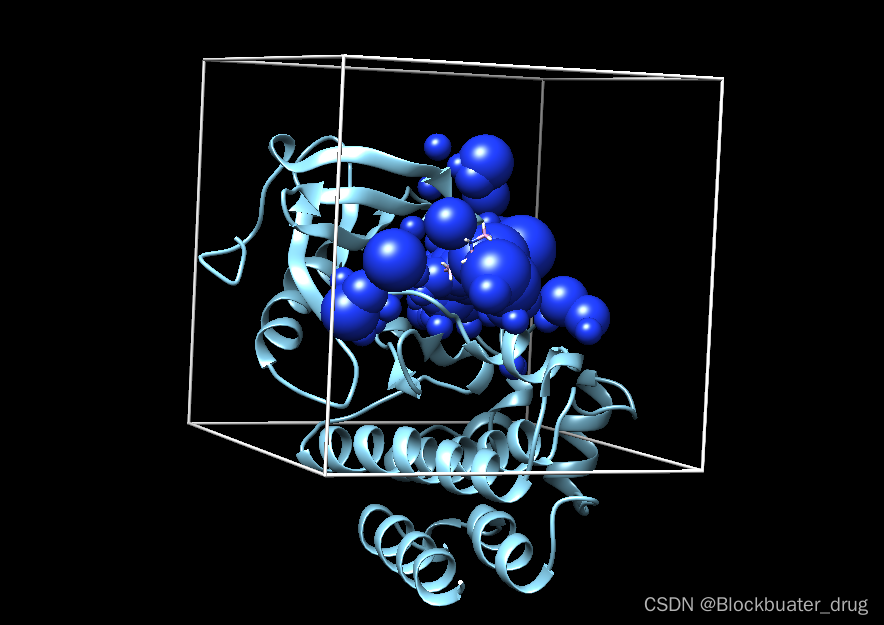
运行命令,生成8u2e.box.pdb,文件。
showbox < showbox.in
可以查看一下;如下图,是符合预期的。
chimera 8u2e.box.pdb ../001_structure/8u2e_rec_withH.mol2 ../001_structure/up9_rec_withH.mol2 ../002_surface_spheres/selected_spheres.sph
5.2 产生格点文件
vim grid.in
输入以下内容:
allow_non_integral_charges nocompute_grids yesgrid_spacing 0.4output_molecule nocontact_score noenergy_score yesenergy_cutoff_distance 9999atom_model aattractive_exponent 6repulsive_exponent 9distance_dielectric yesdielectric_factor 4bump_filter yesbump_overlap 0.75receptor_file ../001_structure/8u2e_rec_withH.mol2box_file 8u2e.box.pdbvdw_definition_file $DOCKHOME/parameters/vdw_AMBER_parm99.defnscore_grid_prefix grid
grid命令并不识别变量,需要将$DOCKHOME替换为自己安装的绝对路径,下同。
grid -i grid.in -o gridinfo.out
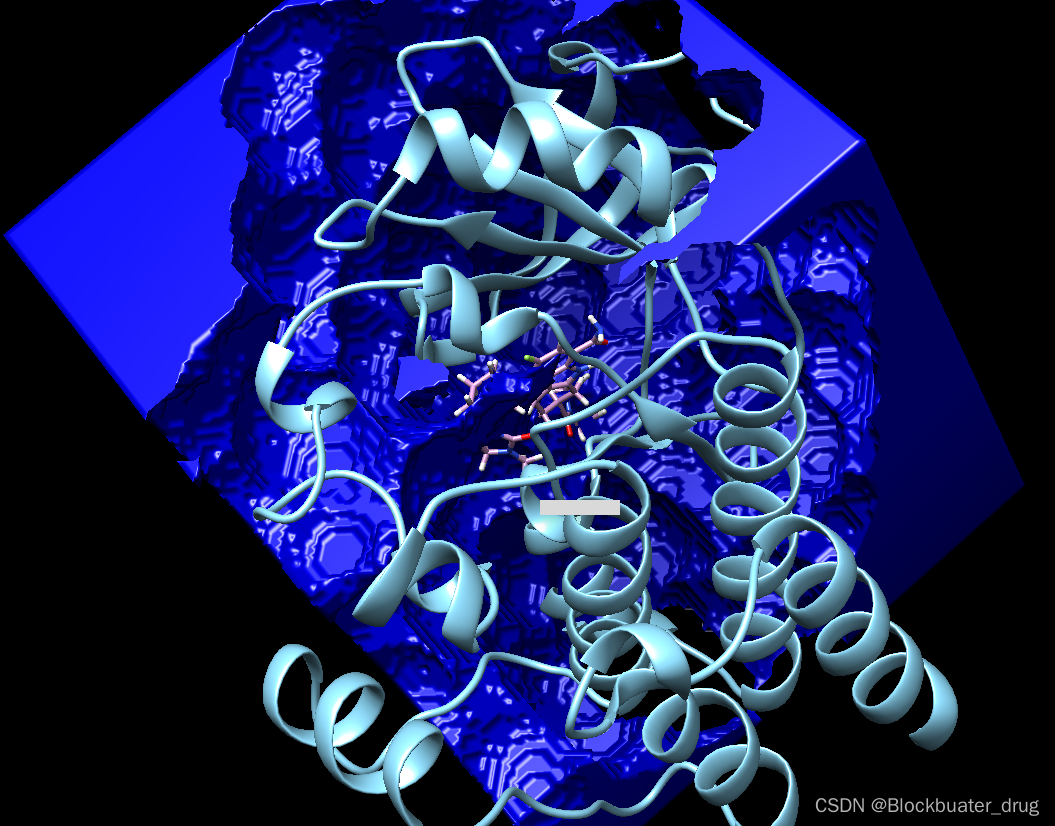
结果可视化,此处参数不再调整,用户可以根据需要优化,将一定程度影响到计算速度。
chimera grid.bmp ../001_structure/8u2e_rec_withH.mol2 ../001_structure/up9_rec_withH.mol2
5.3 能量最小化
新建并生成参数文件:
vim min.in
conformer_search_type rigiduse_internal_energy yesinternal_energy_rep_exp 12internal_energy_cutoff 100.0ligand_atom_file ../000_files/up9_lig_wH.mol2limit_max_ligands noskip_molecule noread_mol_solvation nocalculate_rmsd yesuse_rmsd_reference_mol yes rmsd_reference_filename ../000_files/up9_lig_wH.mol2use_database_filter noorient_ligand nobump_filter noscore_molecules yescontact_score_primary nogrid_score_primary yesgrid_score_rep_rad_scale 1grid_score_vdw_scale 1grid_score_es_scale 1grid_score_grid_prefix grid minimize_ligand yessimplex_max_iterations 1000simplex_tors_premin_iterations 0simplex_max_cycles 1simplex_score_converge 0.1simplex_cycle_converge 1.0simplex_trans_step 1.0simplex_rot_step 0.1simplex_tors_step 10.0simplex_random_seed 0simplex_restraint_min yessimplex_coefficient_restraint 10.0atom_model allvdw_defn_file $DOCKHOME/parameters/vdw_AMBER_parm99.defnflex_defn_file $DOCKHOME/parameters/flex.defnflex_drive_file $DOCKHOME/parameters/flex_drive.tblligand_outfile_prefix up9.lig.minwrite_orientations nonum_scored_conformers 1rank_ligands no
运行min,生成up9.lig.min_scored.mol2文件:
dock6 -i min.in -o min.out
比较一下优化后的:
chimera up9.lig.min_scored.mol2 ../001_structure/up9_rec_withH.mol2
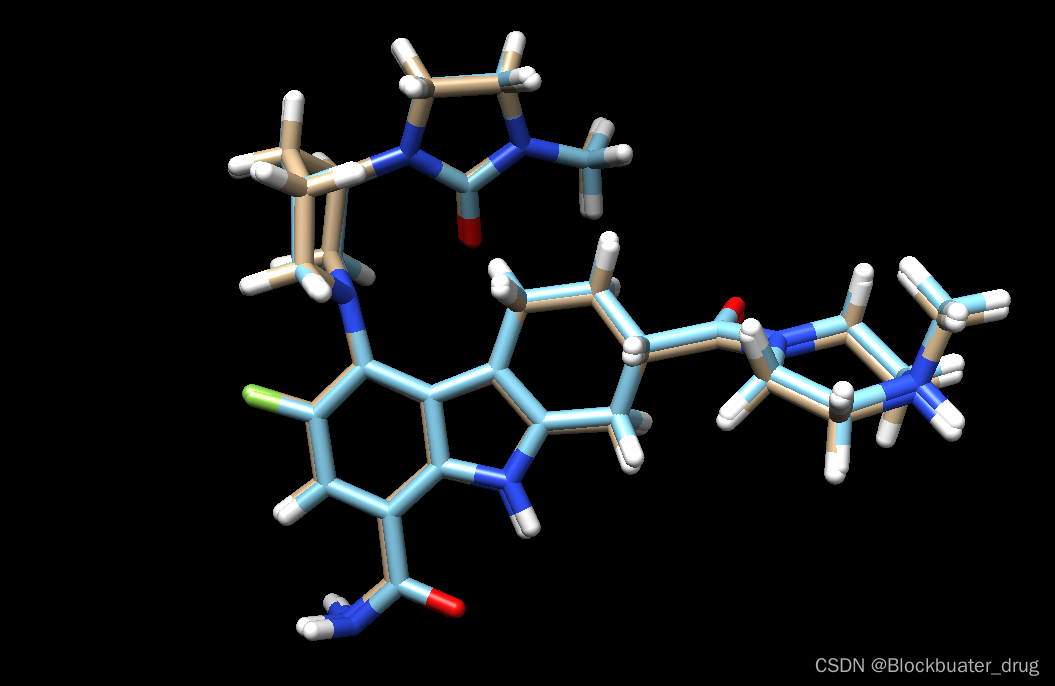 以上工作完成后就可以进入分子对接步骤。
以上工作完成后就可以进入分子对接步骤。
六、分子对接dock
接下来将在能量最小化基础上进行rigid dock,fixed anchor dock和flex dock。
切换文件目录:
cd ../0041_rigid_dock
6.1 rigid dock
vim rigid.in
新建参数输入文件rigid.in,输入以下内容:
conformer_search_type rigid
use_internal_energy yes
internal_energy_rep_exp 12
internal_energy_cutoff 100.0
ligand_atom_file ../003_gridbox/up9.lig.min_scored.mol2
limit_max_ligands no
skip_molecule no
read_mol_solvation no
calculate_rmsd yes
use_rmsd_reference_mol yes
rmsd_reference_filename ../003_gridbox/up9.lig.min_scored.mol2
use_database_filter no
orient_ligand yes
automated_matching yes
receptor_site_file ../002_surface_spheres/selected_spheres.sph
max_orientations 1000
critical_points no
chemical_matching no
use_ligand_spheres no
bump_filter no
score_molecules yes
contact_score_primary no
grid_score_primary yes
grid_score_rep_rad_scale 1
grid_score_vdw_scale 1
grid_score_es_scale 1
grid_score_grid_prefix ../003_gridbox/grid
minimize_ligand yes
simplex_max_iterations 1000
simplex_tors_premin_iterations 0
simplex_max_cycles 1
simplex_score_converge 0.1
simplex_cycle_converge 1.0
simplex_trans_step 1.0
simplex_rot_step 0.1
simplex_tors_step 10.0
simplex_random_seed 0
simplex_restraint_min no
atom_model all
vdw_defn_file $DOCKHOME/parameters/vdw_AMBER_parm99.defn
flex_defn_file $DOCKHOME/parameters/flex.defn
flex_drive_file $DOCKHOME/parameters/flex_drive.tbl
ligand_outfile_prefix rigid.out
write_orientations no
num_scored_conformers 1
rank_ligands no no
运行命令:
dock6 -i rigid.in -o rigid.out
查看结果:
chimera rigid.out_scored.mol2 ../003_gridbox/up9.lig.min_scored.mol2
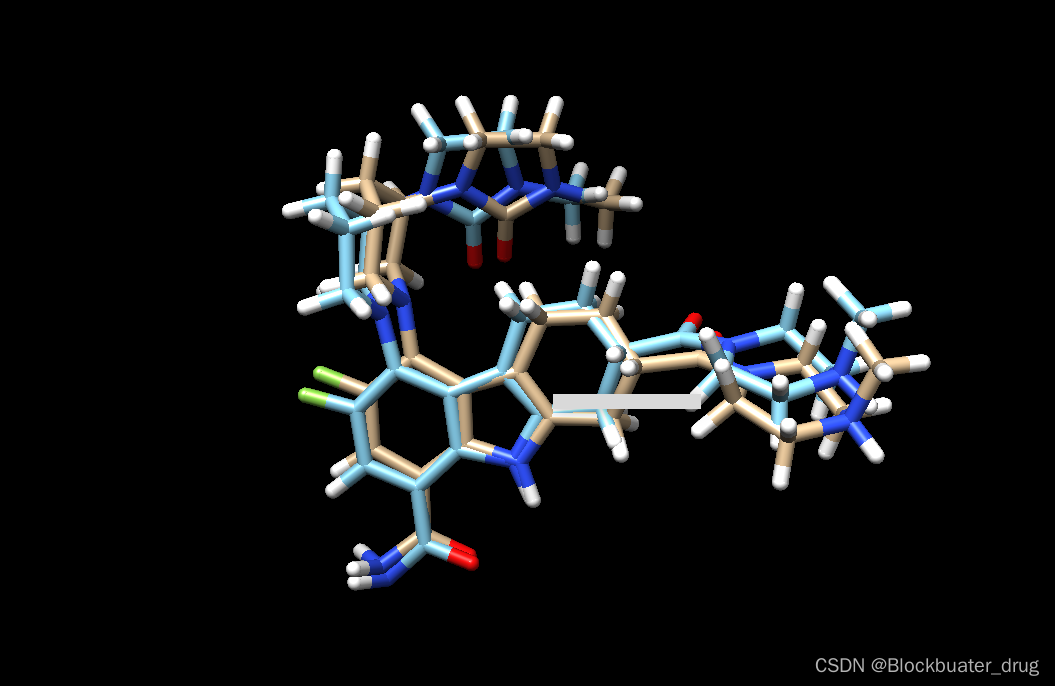
6.2 fixed anchor dock
cd ../0042_fixed_dock
新建参数输入文件fixed.in,输入以下内容:
conformer_search_type flex
write_fragment_libraries no
user_specified_anchor no
limit_max_anchors no
min_anchor_size 5
pruning_use_clustering yes
pruning_max_orients 1000
pruning_clustering_cutoff 100
pruning_conformer_score_cutoff 100.0
pruning_conformer_score_scaling_factor 1.0
use_clash_overlap no
write_growth_tree no
use_internal_energy yes
internal_energy_rep_exp 12
internal_energy_cutoff 100.0
ligand_atom_file ../001_structure/up9_rec_withH.mol2
limit_max_ligands no
skip_molecule no
read_mol_solvation no
calculate_rmsd yes
use_rmsd_reference_mol yes
rmsd_reference_filename ../001_structure/up9_rec_withH.mol2
use_database_filter no
orient_ligand no
bump_filter no
score_molecules yes
contact_score_primary no
grid_score_primary yes
grid_score_rep_rad_scale 1
grid_score_vdw_scale 1
grid_score_es_scale 1
grid_score_grid_prefix ../003_gridbox/grid
minimize_ligand yes
minimize_anchor yes
minimize_flexible_growth yes
use_advanced_simplex_parameters no
minimize_flexible_growth_ramp no
simplex_max_cycles 1
simplex_score_converge 0.1
simplex_cycle_converge 1.0
simplex_trans_step 1.0
simplex_rot_step 0.1
simplex_tors_step 10.0
simplex_anchor_max_iterations 500
simplex_grow_max_iterations 500
simplex_grow_tors_premin_iterations 0
simplex_random_seed 0
simplex_restraint_min no
atom_model all
vdw_defn_file $DOCKHOME/parameters/vdw_AMBER_parm99.defn
flex_defn_file $DOCKHOME/parameters/flex.defn
flex_drive_file $DOCKHOME/parameters/flex_drive.tbl
ligand_outfile_prefix fixed.out
write_orientations no
num_scored_conformers 100
write_conformations no
cluster_conformations yes
cluster_rmsd_threshold 2.0
rank_ligands no
运行:
dock6 -i fixed.in -o fixed.out
查看结果fixed.out_scored.mol2,是一系列pose,与共晶pose和min后作比较:
chimera fixed.out_scored.mol2 ../001_structure/up9_rec_withH.mol2 ../003_gridbox/up9.lig.min_scored.mol2
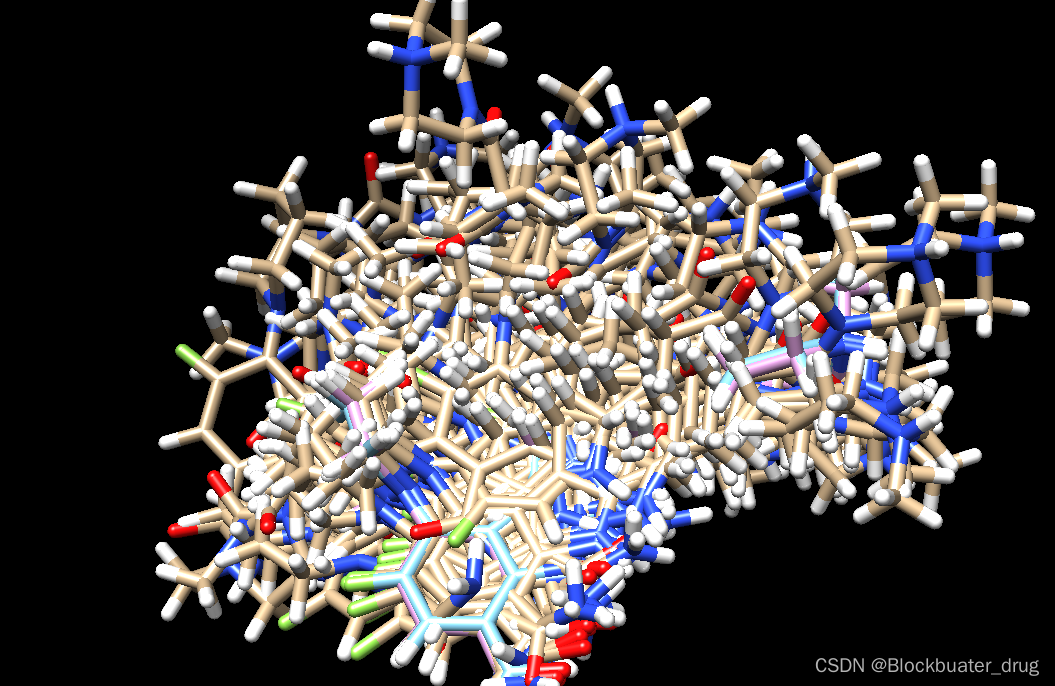 fixed.out_scored.mol2文件内容,记录了28个pose的结果信息。
fixed.out_scored.mol2文件内容,记录了28个pose的结果信息。

6.3 flex dock
切换文件夹:
cd ../0043_flex
新建参数输入文件flex.in,输入以下内容:
vim flex.in
conformer_search_type flex
write_fragment_libraries no
user_specified_anchor no
limit_max_anchors no
min_anchor_size 5
pruning_use_clustering yes
pruning_max_orients 1000
pruning_clustering_cutoff 100
pruning_conformer_score_cutoff 100.0
pruning_conformer_score_scaling_factor 1.0
use_clash_overlap no
write_growth_tree no
use_internal_energy yes
internal_energy_rep_exp 12
internal_energy_cutoff 100.0
ligand_atom_file ../003_gridbox/up9.lig.min_scored.mol2
limit_max_ligands no
skip_molecule no
read_mol_solvation no
calculate_rmsd yes
use_rmsd_reference_mol yes
rmsd_reference_filename ../003_gridbox/up9.lig.min_scored.mol2
use_database_filter no
orient_ligand yes
automated_matching yes
receptor_site_file ../002_surface_spheres/selected_spheres.sph
max_orientations 1000
critical_points no
chemical_matching no
use_ligand_spheres no
bump_filter no
score_molecules yes
contact_score_primary no
grid_score_primary yes
grid_score_rep_rad_scale 1
grid_score_vdw_scale 1
grid_score_es_scale 1
grid_score_grid_prefix ../003_gridbox/grid
minimize_ligand yes
minimize_anchor yes
minimize_flexible_growth yes
use_advanced_simplex_parameters no
minimize_flexible_growth_ramp no
simplex_max_cycles 1
simplex_score_converge 0.1
simplex_cycle_converge 1.0
simplex_trans_step 1.0
simplex_rot_step 0.1
simplex_tors_step 10.0
simplex_anchor_max_iterations 500
simplex_grow_max_iterations 500
simplex_grow_tors_premin_iterations 0
simplex_random_seed 0
simplex_restraint_min no
atom_model all
vdw_defn_file $DOCKHOME/parameters/vdw_AMBER_parm99.defn
flex_defn_file $DOCKHOME/parameters/flex.defn
flex_drive_file $DOCKHOME/parameters/flex_drive.tbl
ligand_outfile_prefix flex.out
write_orientations no
num_scored_conformers 1
rank_ligands no
运行:
dock6 -i flex.in -o flex.out
查看结果:
chimera flex.out_scored.mol2 ../001_structure/up9_rec_withH.mol2 ../003_gridbox/up9.lig.min_scored.mol2
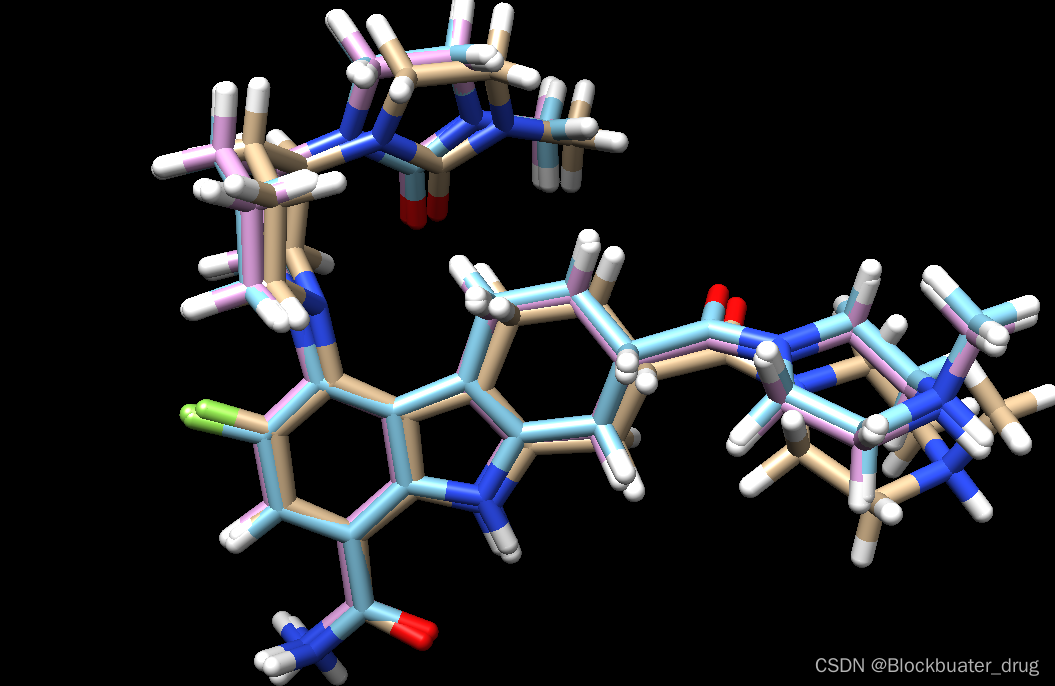 可以看出,对接结果与共晶结构重叠良好,其中侧链部分有较小的差异。
可以看出,对接结果与共晶结构重叠良好,其中侧链部分有较小的差异。
6.4 配体-受体footprint指纹
切换文件夹:
cd ../0044_footprint
新建参数输入文件footprint.in.in,输入以下内容:
vim footprint.in
conformer_search_type rigid
use_internal_energy no
ligand_atom_file ../003_gridbox/up9.lig.min_scored.mol2
limit_max_ligands no
skip_molecule no
read_mol_solvation no
calculate_rmsd no
use_database_filter no
orient_ligand no
bump_filter no
score_molecules yes
contact_score_primary no
grid_score_primary no
multigrid_score_primary no
dock3.5_score_primary no
continuous_score_primary no
footprint_similarity_score_primary yes
fps_score_use_footprint_reference_mol2 yes
fps_score_footprint_reference_mol2_filename ../001_structure/up9_rec_withH.mol2
fps_score_foot_compare_type Euclidean
fps_score_normalize_foot no
fps_score_foot_comp_all_residue yes
fps_score_receptor_filename ../001_structure/8u2e_rec_withH.mol2
fps_score_vdw_att_exp 6
fps_score_vdw_rep_exp 9
fps_score_vdw_rep_rad_scale 1
fps_score_use_distance_dependent_dielectric yes
fps_score_dielectric 4.0
fps_score_vdw_fp_scale 1
fps_score_es_fp_scale 1
fps_score_hb_fp_scale 0
minimize_ligand no
atom_model all
vdw_defn_file $DOCKHOME/parameters/vdw_AMBER_parm99.defn
flex_defn_file $DOCKHOME/parameters/flex.defn
flex_drive_file $DOCKHOME/parameters/flex_drive.tbl
ligand_outfile_prefix footprint.out
write_footprints yes
write_hbonds yes
write_orientations no
num_scored_conformers 1
rank_ligands no
运行:
dock6 -i footprint.in -o footprint.out
查看结果:
python plot_footprint_single_magnitude.py footprint.out_footprint_scored.txt 50

footprint的结果查看有官方推荐的Python脚本,如果网站打不开,可以从这里下载:footprint 查看脚本 plot_footprint_single_magnitude.py
从footprint可以直观的看到对范德华作用和静电作用贡献较大的残基,用于结合模式进一步分析对比。
总结
本文是UCSF DOCK的使用案例分享,包括:(1)配体、受体输入文件处理;(2)分子对接;(3)配体-受体相互作用中范德华与静电能量分解的指纹footprint 。
参考资料
- https://download.csdn.net/download/weixin_40192882/88895809
- https://ringo.ams.stonybrook.edu/index.php/Rizzo_Lab_Information_and_Tutorials
- https://bbdrug.blog.csdn.net/article/details/136135888
- https://bbdrug.blog.csdn.net/article/details/136312633
| 欢迎浏览我的CSND博客! Blockbuater_drug …点击进入 |
|---|






![[Android View] 可绘制形状 (Shape Xml)](https://img-blog.csdnimg.cn/direct/18beaa5600c14933ac0269b946d8701c.png)
![[linux][xdp] xdp 入门](https://img-blog.csdnimg.cn/direct/defd49fdfb1e473fa7d37798ec1bd8a1.png)