- 对接完整步骤

- 具体操作
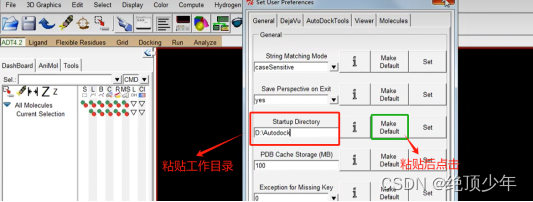
- 设置工作目录

- 设置工作目录
保证工作目录下必须要有这五个文件:
-
- 对蛋白质的操作
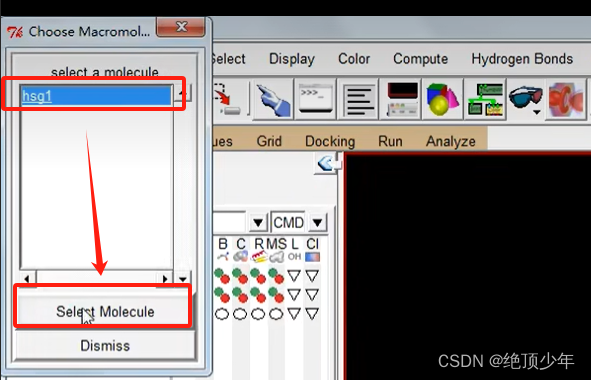
- 打开蛋白质

- 打开蛋白质
- 对蛋白质的操作
-
-
- 去水,结构周围的小红点。

- 去水,结构周围的小红点。
-
-
-
- 加氢

- 加氢
-

-
-
- 将蛋白质设为受体
-

-
-
- 点击确定进行保存
-


进行下一步小分子
-
- 小分子具体操作
- 打开小分子
- 小分子具体操作

-
-
- 对小分子进行加氢
-


-
-
- 将小分子设定为配体
-



-
-
- 检测扭转键以及其中心
-



-
-
- 导出为pdbqt
-

-
-
- 选中删除
-

-
- 进行autogride
 然后在打开的文件框双击蛋白质文件,然后出现下方弹窗点击yes
然后在打开的文件框双击蛋白质文件,然后出现下方弹窗点击yes
- 进行autogride

-
-
- 导入小分子
-

-
-
- 方便演示可以通过对应框换一个模型
-
-
-
- 设定运行box的大小,调整大小使其包裹住整个蛋白质,但如果知道蛋白质的活性位点的话也可以只包裹住对应的活性位点位置。(鼠标左键旋转,右键拖动,滑轮放大与缩小)图二下面未框部分为左右上下移动。此步骤完成后不要叉掉。
-


-
-
- 将小分子从box中取出来,按下图操作后,记得再勾选上第四步那个框框然后叉掉就好了
-

-
-
- 保存盒子的大小
-

-
-
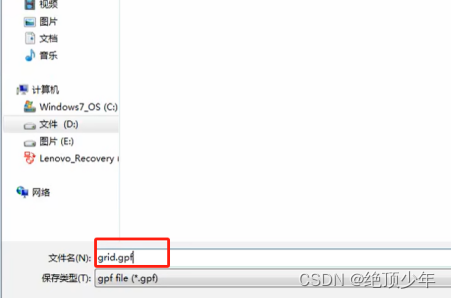
- 导出为gpf
-

-
-
- 保存时候注意命名的后缀
-
-
-
- 然后运行autogride
-



出现该弹窗后切记要耐心等待他自己消失,不要叉掉
-
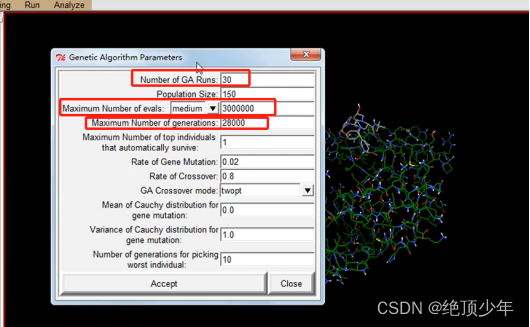
- 准备autodock的运行参数以及计算方法
- 按下图操作后选中蛋白质的pdbgt文件
- 准备autodock的运行参数以及计算方法












-
-
- 运行完成后删除分子
-


-
-
- 查看对接结果
-







此操作显示生成的结合位点的结合能信息。


去水方式:

再设定为配体时候可能会报错,进行g电荷计算: