小罗碎碎念
本期文献主题:病理AI领域的最新基础模型
今天的推文是一期生日特辑,定时在下午六点二十一分发表(今天农历六月二十一,哈哈),算是自己给自己的24岁生日礼物,希望24岁这一年,能离自己的目标越来越近!!
我在这期推文汇总了三篇顶刊发表的病理AI领域的基础模型,并按照时间顺序依次介绍给大家,希望你们能从这篇推文中获取自己想要的信息。
我是罗小罗同学,我们顶峰见!!
| 发表时间 | 发表期刊 | |
|---|---|---|
| Towards a general-purpose foundation model for computational pathology | 24-03-19 | Nature Medicine |
| A whole-slide foundation model for digital pathology from real-world data | 24-05-22 | Nature |
| A foundation model for clinical-grade computational pathology and rare cancers detection | 24-07-22 | Nature Medicine |
一、UNI:开启计算病理学新篇章的自监督基础模型

一作&通讯
| 角色 | 姓名 | 单位(中文) |
|---|---|---|
| 第一作者 | Richard J. Chen | 哈佛医学院布里格姆和妇女医院病理科 |
| 第一作者 | Tong Ding | 哈佛医学院工程与应用科学学院 |
| 第一作者 | Ming Y. Lu | 哈佛医学院和麻省理工学院癌症项目 |
| 通讯作者 | Faisal Mahmood | 哈佛医学院布里格姆和妇女医院病理科 |
文献概述
这篇文章介绍了一个名为UNI的新型通用自监督模型,它在计算病理学领域通过大规模预训练显著提高了组织图像分析的性能,并在多种临床任务上展现了卓越的泛化能力。
-
研究背景:计算病理学在评估组织图像方面至关重要,需要从全切片图像(Whole-Slide Images, WSIs)中客观地表征组织病理学实体。WSIs的高分辨率和形态特征的变异性带来了显著挑战,使得大规模注释数据变得复杂,限制了高性能应用的发展。
-
研究目的:为了解决这些挑战,研究者们提出了使用预训练图像编码器的方法,通过从自然图像数据集的迁移学习或在公开可用的组织病理学数据集上的自监督学习来实现。然而,这些方法在不同组织类型和规模上的开发和评估还不够广泛。
-
研究方法:文章介绍了UNI,这是一个用于病理学的通用自监督模型,使用超过1亿张图像(超过77 TB数据)从超过10万个诊断H&E染色的WSIs中预训练,涵盖了20种主要组织类型。模型在34个代表性的CPath任务上进行了评估,这些任务在诊断难度上有所不同。
-
研究结果:UNI在多个任务中表现出色,除了超越了之前的最佳模型,还展示了CPath中的新建模能力,例如分辨率无关的组织分类、使用少量样本类原型的切片分类,以及在OncoTree分类系统中对多达108种癌症类型的疾病亚型泛化分类。
-
模型评估:研究者们还评估了UNI在不同数据规模下的预训练效果,以及不同ViT(Vision Transformer)架构大小对模型性能的影响。通过与MoCoV3等其他自监督学习算法的比较,研究者们发现DINOv2算法在大规模数据集上的表现更好。
-
临床应用:文章还讨论了UNI在临床病理学中的潜在应用,包括肿瘤检测、亚型分类、分级和分期,以及分子改变预测等。研究者们强调了UNI在处理罕见疾病和诊断复杂性较高的任务中的优势。
-
数据集和实验设置:研究者们使用了大规模、分层的癌症分类任务来评估UNI的泛化能力,这些任务遵循OncoTree癌症分类系统。他们还评估了不同数据多样性和预训练长度对模型性能的影响。
-
结论:UNI作为一个通用的自监督视觉编码器,展示了在多种机器学习设置中的多功能性,包括ROI级别的分类、分割和图像检索,以及切片级别的弱监督学习。研究者们认为UNI有潜力成为进一步发展解剖病理学中人工智能模型的基础模型。
文章还详细讨论了数据集的构建、模型的架构和预训练协议、评估设置、比较和基线、弱监督切片分类、线性和K-最近邻探针评估、ROI检索、ROI级别的细胞类型分割、少样本ROI分类和原型学习等方法和技术细节。
重点关注
Fig. 1 提供了对 UNI 模型的全面概述,它是一个用于解剖病理学的通用自监督视觉编码器,基于视觉变换器(vision transformer)架构,并且在34个临床病理学任务中实现了最先进的性能。
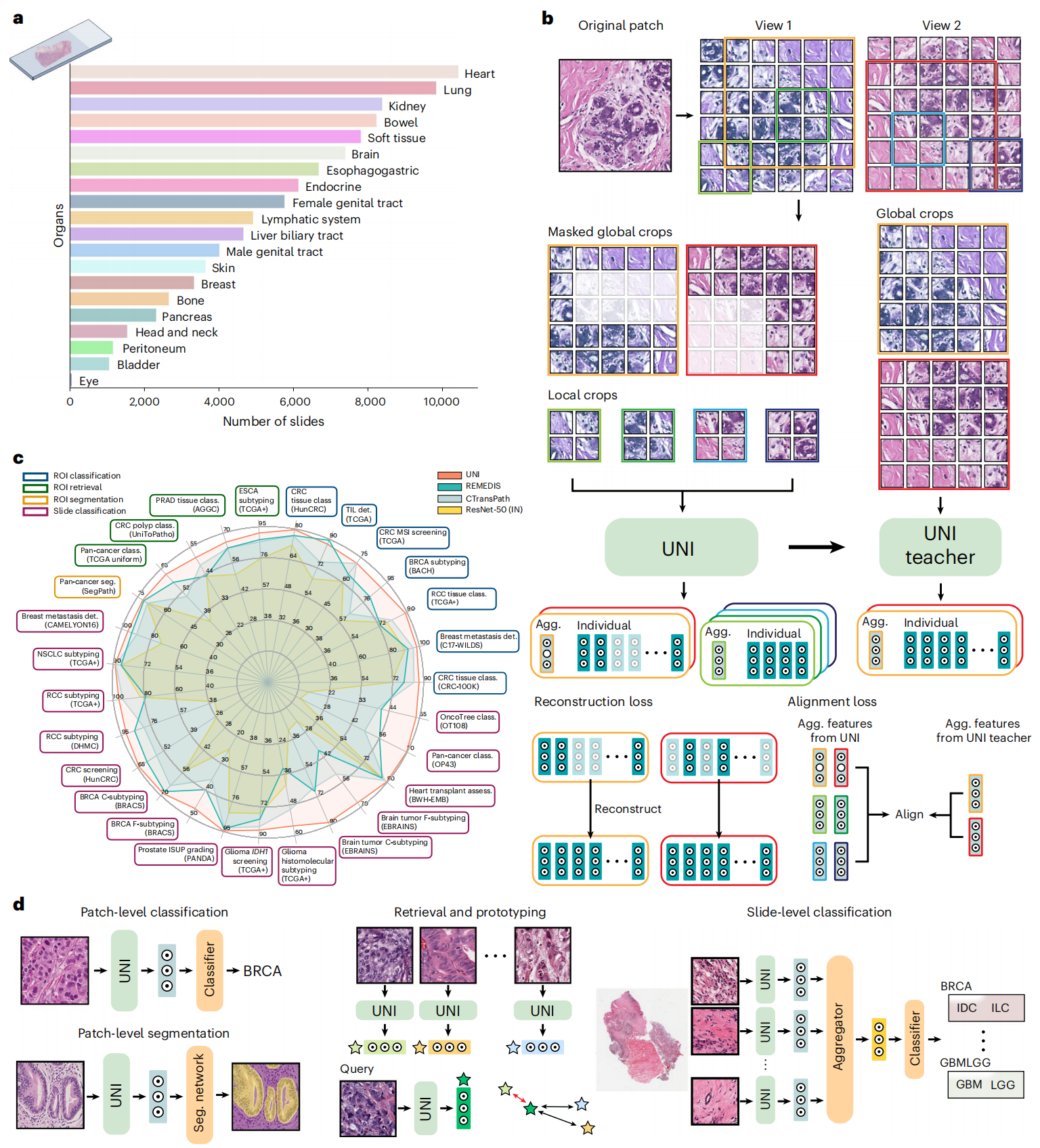
a. 大规模预训练数据集 Mass-100K 的切片分布:这个数据集包含了从超过10万个诊断性的全切片图像(WSIs)中抽取的1亿个组织样本,覆盖了20种主要的器官类型。这表明 UNI 模型在预训练阶段就拥有了一个庞大且多样化的数据基础。
b. 使用 DINOv2 自我监督训练算法的 UNI 预训练:UNI 在 Mass-100K 数据集上使用 DINOv2 算法进行预训练,该算法包括一个掩码图像建模目标和一个自我蒸馏目标。这意味着 UNI 通过自监督学习,能够无需人工标注即可学习到图像的特征表示。
c. UNI 在临床病理学任务中的性能:在34个不同的临床病理学任务中,UNI 普遍优于其他预训练编码器,报告的是8个SegPath任务的平均性能。这显示了 UNI 在多种病理学应用中的泛化能力和高效性。
d. 评估任务的多样性:评估任务包括了区域感兴趣(ROI)级别的分类、分割、检索和原型制作,以及切片级别的分类任务。这表明 UNI 不仅能够在图像的局部区域进行精确分析,还能够在整个切片的尺度上进行综合评估。
整体来看,Fig. 1 展示了 UNI 作为一个基础模型在病理学图像分析中的广泛应用和强大性能,其预训练策略和在多样化任务上的表现证明了其作为病理学图像分析领域的一个重要进展。
二、Prov-GigaPath:能够处理包含数十亿像素的全切片图像的全新数字病理基础模型

一作&通讯
| 作者角色 | 作者姓名 | 单位名称(中文) |
|---|---|---|
| 第一作者 | Hanwen Xu | 微软研究院,华盛顿州雷德蒙德,美国 |
| 第一作者 | Naoto Usuyama | 华盛顿大学保罗·G·艾伦计算机科学与工程学院,华盛顿州西雅图,美国 |
| 通讯作者 | Carlo Bifulco | Providence基因组学,俄勒冈州波特兰,美国 |
| 通讯作者 | Sheng Wang | 华盛顿大学保罗·G·艾伦计算机科学与工程学院,华盛顿州西雅图,美国 |
| 通讯作者 | Hoifung Poon | 微软研究院,华盛顿州雷德蒙德,美国 |
文献概述
这篇文章是发表在《Nature》的一篇关于数字病理学领域的研究,介绍了一个名为Prov-GigaPath的全新数字病理基础模型,该模型在大规模真实世界数据上进行了预训练,能够处理包含数十亿像素的全切片图像。
-
背景与挑战:数字病理学面临的计算挑战包括处理标准千兆像素级别的切片,这些切片可能包含数万个图像瓦片。以往的模型常常通过抽样切片中的一小部分瓦片来处理,这会丢失重要的切片级上下文信息。
-
Prov-GigaPath模型:作者提出了Prov-GigaPath,这是一个在1.3十亿个256×256病理图像瓦片上预训练的全切片病理学基础模型。这些图像瓦片来自美国大型医疗网络Providence的171,189个全切片,涵盖了超过30,000名患者的31种主要组织类型。
-
GigaPath架构:为了预训练Prov-GigaPath,作者提出了GigaPath,这是一种新颖的视觉变换器架构,用于预训练千兆像素级别的病理切片。GigaPath采用了新开发的LongNet方法来适应数字病理学中的数万个图像瓦片。
-
性能评估:作者构建了一个包含9个癌症亚型任务和17个病理组学任务的数字病理学基准,使用Providence和TCGA的数据进行评估。Prov-GigaPath在26个任务中的25个上达到了最先进的性能,并且在18个任务上显著优于第二好的方法。
-
多模态预训练:文章还探讨了Prov-GigaPath在病理报告的视觉-语言预训练方面的潜力,通过结合病理报告,展示了Prov-GigaPath在病理图像分析方面的多模态集成数据分析能力。
-
临床应用:Prov-GigaPath作为一个开放权重的基础模型,展示了在各种数字病理学任务上的卓越性能,强调了真实世界数据和全切片建模的重要性。
-
数据与代码可用性:用于预训练的病理影像数据和相关临床数据不能公开,但作者提供了一些去标识化的测试子集,并且Prov-GigaPath的模型权重和相关源代码可以在GitHub上访问。
文章强调了Prov-GigaPath在提高癌症诊断、预后预测和个性化医疗方面的潜力,并展望了其在未来可能的应用。
重点关注
图1提供了Prov-GigaPath模型架构的概览,具体内容可以分为以下几个部分:
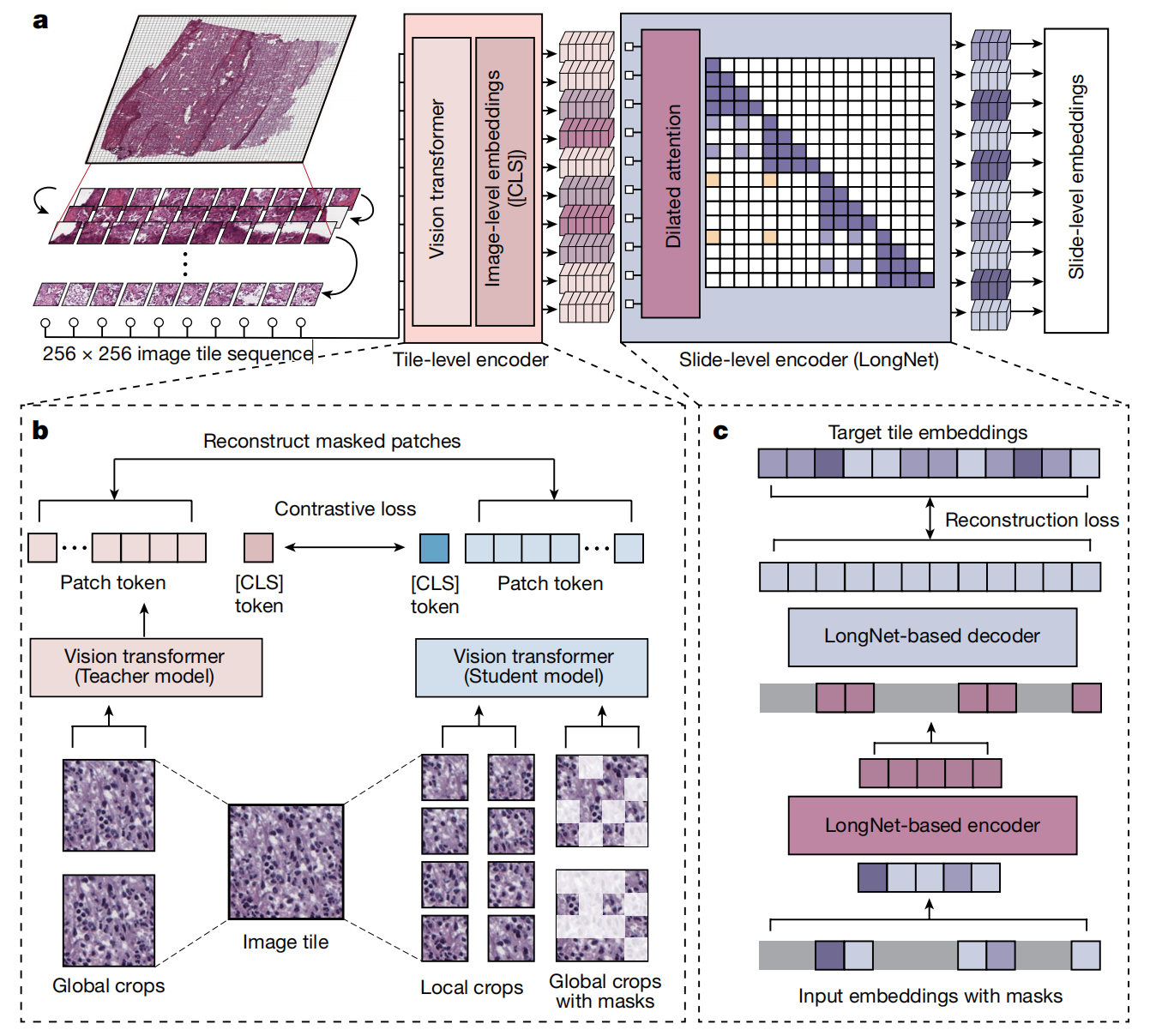
a. 模型架构流程图:
- Prov-GigaPath首先将每个输入的全切片图像(Whole Slide Image, WSI)序列化为按行主序排列的256×256像素图像瓦片序列。
- 使用图像瓦片级编码器(tile encoder)将每个图像瓦片转换成视觉嵌入(visual embedding)。
- 接着,Prov-GigaPath应用基于LongNet架构的切片级编码器(slide encoder),生成上下文化的嵌入(contextualized embeddings),这些嵌入可以作为各种下游应用的基础。
b. 图像瓦片级预训练:
- 使用DINOv2框架进行图像瓦片级的自监督预训练。DINOv2是一种先进的图像自监督学习框架,能够学习图像的特征表示。
c. 切片级预训练:
- 使用带有掩码的自编码器(masked autoencoder)和LongNet进行切片级的自监督预训练。LongNet是一种新开发的方法,适用于超长序列建模,能够适应数字病理学的需求。
[CLS]分类标记:
- 在模型中,[CLS]标记是分类(classification)的标记,通常用于Transformer模型中的序列分类任务。在训练过程中,这个标记会聚合整个序列的信息,以预测整个切片的标签。
总结:
Prov-GigaPath模型通过两阶段的预训练过程来学习图像瓦片和切片级别的特征。首先,它将全切片图像分割成小的图像瓦片,并学习每个瓦片的视觉特征。然后,它使用这些瓦片的特征来生成整个切片的上下文嵌入,这些嵌入可以捕捉到切片的全局模式和局部细节,为后续的病理分析任务提供支持。这种设计使得Prov-GigaPath能够有效地处理和分析大规模的病理图像数据,并在多种病理学任务中表现出色。
三、泛癌种检测与生物标志物预测:Virchow基础模型的性能与潜力

一作&通讯
| 作者类型 | 作者姓名 | 单位名称(英文) | 单位名称(中文) |
|---|---|---|---|
| 第一作者 | Eugene Vorontsov | Paige, New York, NY, US | 佩奇公司,纽约,美国 |
| 第一作者 | Alican Bozkurt | Microsoft Research, Cambridge, MA, US | 微软研究院,剑桥,马萨诸塞州,美国 |
| 通讯作者 | Siqi Liu | Paige, New York, NY, US | 佩奇公司,纽约,美国 |
| 通讯作者 | Thomas J. Fuchs | Memorial Sloan Kettering Cancer Center, New York, NY, US | 纪念斯隆凯特琳癌症中心,纽约,美国 |
文献概述
这篇文章介绍了Virchow,一个用于临床级计算病理学和罕见癌症检测的新型大型基础模型,它通过自监督学习在大量病理图像数据上训练,在多种癌症检测和生物标志物预测任务中展现出高性能。
Virchow是目前最大的计算病理学基础模型,它不仅能够预测生物标志物和识别细胞,还能实现泛癌种检测,包括九种常见和七种罕见的癌症类型。研究显示,即使在训练数据较少的情况下,基于Virchow构建的泛癌种检测器也能够达到与特定组织临床级模型相似的性能,并且在某些罕见癌症变体上表现更优。
Virchow模型通过自监督学习算法训练,能够生成数据表示(嵌入),这些嵌入能够很好地泛化到多种预测任务。与传统的计算病理学方法相比,Virchow能够捕捉到更广泛的组织形态学变化和实验室准备的变化,这对于临床实践中的泛化至关重要。此外,Virchow模型在处理罕见肿瘤类型或不常见的诊断任务(如预测特定的基因组变化、临床结果和治疗反应)时,显示出了其独特的价值。
研究还展示了Virchow在生物标志物预测方面的应用,这可以减少对额外测试的需要,加快为患者提供关键数据的速度。Virchow模型在多个层面上展示了其在计算病理学新领域的强大潜力,包括在罕见癌症检测和生物标志物预测方面的性能。
文章还讨论了Virchow模型的潜在临床影响,包括在临床实践中减少诊断周转时间、为不常见癌症开发临床级产品、以及使用常规H&E染色的WSI进行生物标志物预测等。
此外,文章也指出了Virchow模型的局限性,包括训练数据集来源单一、模型和数据规模的饱和点尚未明确等,并对未来的研究方向提出了展望。
重点关注
图1提供了Virchow模型研究的全面概述,包括训练数据集、训练算法和应用。
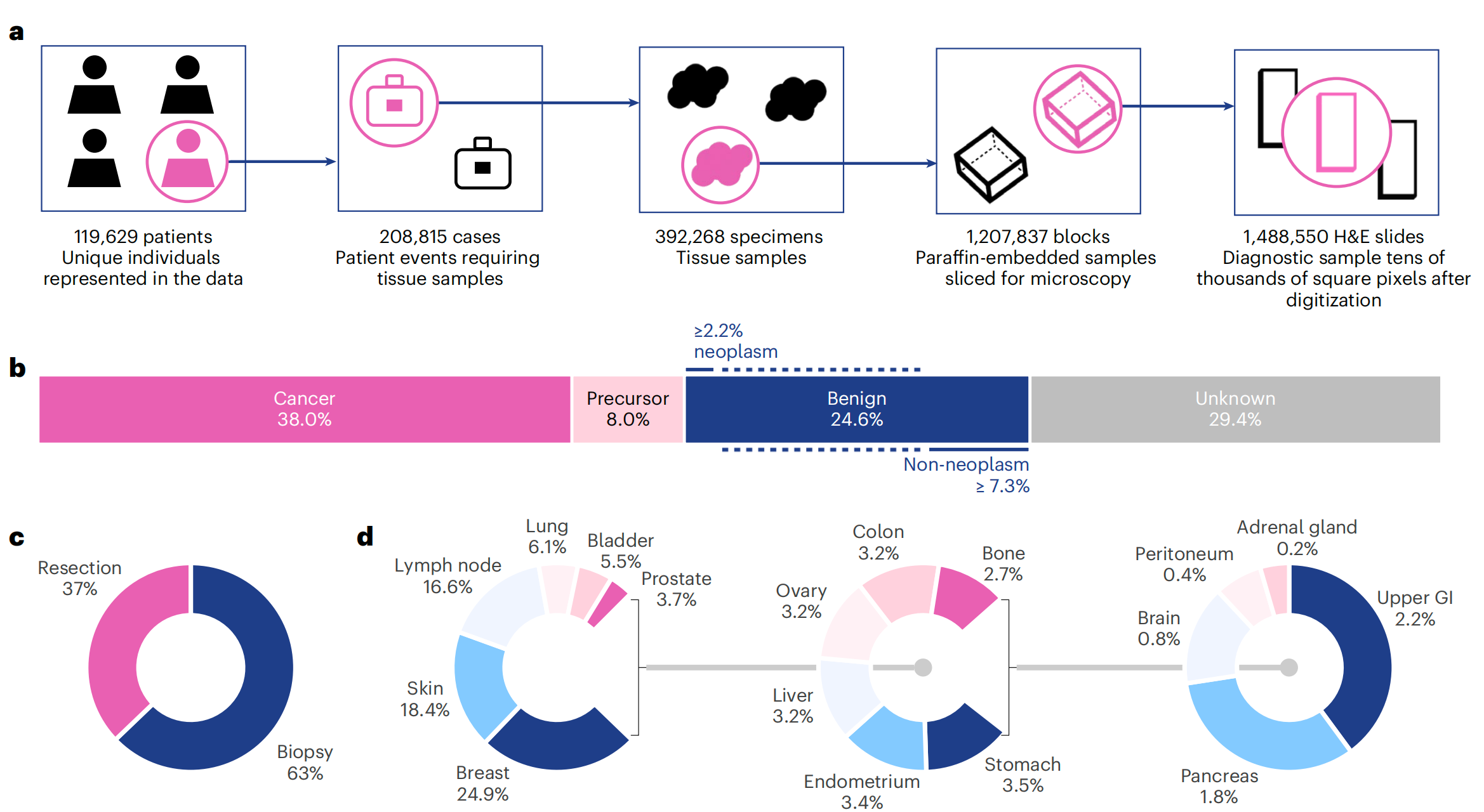
a. 训练数据描述:数据集可以从多个维度描述,包括患者(patients)、病例(cases)、样本(specimens)、组织块(blocks)或切片(slides)。这些维度展示了数据集的规模和多样性。
b. 癌症状态的切片分布:这部分展示了不同癌症状态下的切片分布,可能包括癌症、前体病变(如原位癌)、良性病变和未知状态。这有助于理解模型在不同癌症类型上的表现。
c. 手术类型的切片分布:这部分展示了根据手术类型(如活检、切除等)的切片分布。这有助于评估模型在不同手术背景下的适用性。
d. 组织类型的切片分布:这部分展示了不同组织类型的切片分布,如乳腺、皮肤、淋巴结等。这有助于了解模型在不同组织类型上的泛化能力。
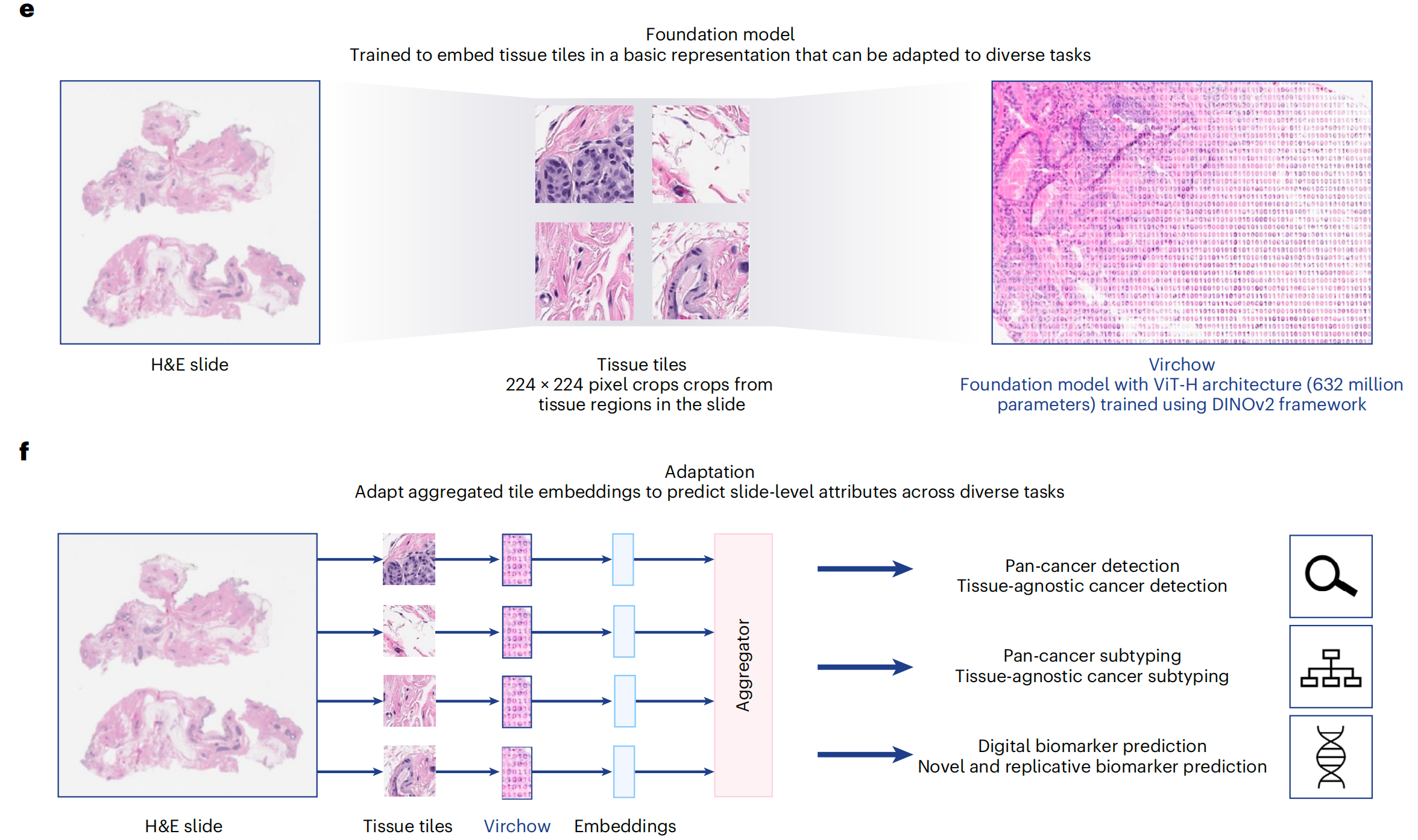
e. 训练中的数据流:在训练过程中,切片被处理成小块(tiles),然后这些小块被裁剪成全局视图和局部视图。这种处理方式有助于模型学习到更丰富的局部和全局特征。
f. 基础模型的应用:展示了如何使用聚合器模型(aggregator model)来预测切片级别的属性。聚合器模型将小块嵌入(tile embeddings)聚合起来,以预测整个切片的属性,如癌症存在与否、癌症类型等。GI(胃肠道)在这里可能是指模型在胃肠道相关病理图像上的应用。
总结来说,图1展示了Virchow模型从训练数据的准备到模型训练,再到最终应用的整个流程。通过这种设计,Virchow能够处理和分析大规模的病理图像数据,为临床病理学提供支持。









![[数据集][目标检测]金属罐缺陷检测数据集VOC+YOLO格式8095张4类别](https://i-blog.csdnimg.cn/direct/9bbf7c52fa9e4e5cb05cf4d76b7ffea1.png)