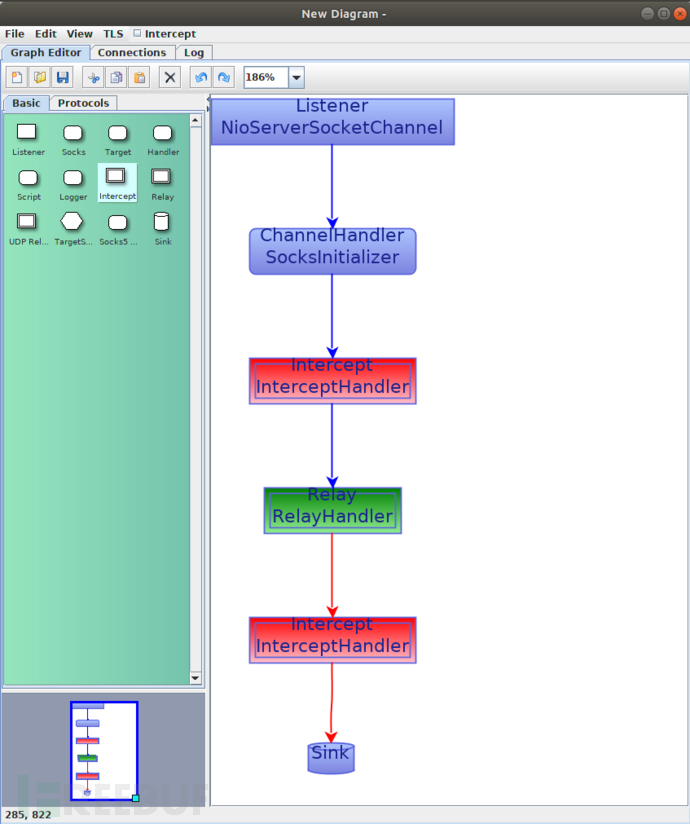
零基础入门转录组数据分析——机器学习算法之boruta(筛选特征基因)
目录
- 零基础入门转录组数据分析——机器学习算法之boruta(筛选特征基因)
- 1. boruta基础知识
- 2. boruta(Rstudio)——代码实操
- 2. 1 数据处理
- 2. 2 构建boruta模型
- 2. 3 boruta结果简单可视化
- 注:配套资源只要改个路径就能运行,本人已检测过可以跑通,请放心食用,食用过程遇到问题,可先自行百度,实在解决不了可以私信
1. boruta基础知识
1.1 boruta是什么?
Boruta算法是一种基于随机森林的特征选择方法,旨在从给定的特征集合中找到真正重要的特征,并区分出无关的特征
1.2 boruta的算法原理是什么?
Boruta算法的核心思想是通过比较原始特征与其对应的“影子特征”(shadow features)在随机森林中的重要性,来确定哪些特征是真正重要的。影子特征是通过将原始特征的顺序打乱、加入噪声和随机化来生成的,它们模拟了随机选择的特征,用于与原始特征进行比较
1.3 boruta的优势?
- 自动化特征选择: Boruta算法可以自动进行特征选择,无需手动调整参数或选择特定的特征子集,这有助于减少人工干预。
- 全相关特征选择: Boruta算法能够识别与目标变量全相关的特征,这些特征可能对预测模型具有显著贡献,有助于更全面地了解特征集合中的重要信息。
- 避免过拟合: 与传统的特征选择方法相比,Boruta算法通过使用随机森林和自助重采样技术可以有效地处理大规模数据集并保持泛化能力,从而避免过拟合问题。
- 提高模型可解释能力: Boruta算法提供了每个特征的重要性分数,这有助于理解特征之间的相对重要性并解释模型预测结果。
1.4 boruta的本质是什么?
筛选出一些关键特征,这些关键特征相对于其他特征来说,区分样本的能力更加精确。
举个栗子: 输入了8个基因的表达矩阵,此时先对数据中的这8个基因随机排列,构建随机组合的影子特征(shadow features),然后基于训练集中基因表达量对于样本的区分程度,对每个基因的重要性(importance)进行打分,看每个基因在原数据中的评分是否比在随机排列中的评分更高,有的话就被记录下来。
注:作者关于这个算法底层原理了解不深入,关于这个算法的底层概念大家可以自行检索学习,这个教程是带领大家实战——即用代码实现boruta筛选关键基因,重点不在于原理讲解
综上所述: boruta就是一种用来筛选 关键特征 的方法(其底层算法依据是随机森林),这个关键特征可以是临床指标,也可以是重要基因等,并且在关键特征选择的时候避免了人为的选择,输出基因重要性提高了可解释性。
2. boruta(Rstudio)——代码实操
本项目以TCGA——肺腺癌为例展开分析
物种:人类(Homo sapiens)
R版本:4.2.2
R包:tidyverse,Boruta,caret
废话不多说,代码如下:
2. 1 数据处理
设置工作空间:
rm(list = ls()) # 删除工作空间中所有的对象
setwd('/XX/XX/XX') # 设置工作路径
if(!dir.exists('./10_boruta')){dir.create('./10_boruta')
}
setwd('./10_boruta/')
加载包:
library(tidyverse)
library(Boruta)
library(caret)
导入要分析的表达矩阵train_data ,并对train_data 的列名进行处理(这是因为在读入的时候系统会默认把样本id中的“-”替换成“.”,所以要给替换回去)
train_data <- read.csv("./data_fpkm.csv", row.names = 1, check.names = F) # 行名为全部基因名,每列为样本名
colnames(train_data) <- gsub('.', '-', colnames(train_data), fixed = T)
train_data 如下图所示,行为基因名(symbol),列为样本名

导入分组信息表group
group <- read.csv("./data_group.csv", row.names = 1) # 为每个样本的分组信息(tumor和normal)
colnames(group) <- c('sample', 'group')
group 如下图所示,第一列sample为样本名,第二列为样本对应的分组 (分组为二分类变量:disease和control)

导入要筛选的基因hub_gene (8个基因)
hub_gene <- data.frame(symbol = gene <- c('ADAMTS2', 'ADAMTS4', 'AGRN', 'COL5A1', 'CTSB', 'FMOD', 'LAMB3', 'LAMB4'))
colnames(hub_gene) <- "symbol"
hub_gene 如下图所示,只有一列:8个基因的基因名

从全部的基因表达矩阵中取出这8个基因对应的表达矩阵,并且与之前准备的分组信息表进行合并
dat <- train_data[rownames(train_data) %in% hub_gene$symbol, ] %>%t() %>%as.data.frame() # 整理后行为样本名,列为基因名
dat$sample <- rownames(dat)
dat <- merge(dat, group, var = "sample")
dat <- column_to_rownames(dat, var = "sample") %>% as.data.frame()table(dat$group)
dat$group <- factor(dat$group, levels = c('disease', 'control'))
dat 如下图所示,行为基因名,前8列为基因对应的表达矩阵,第9列为合并的分组信息表

2. 2 构建boruta模型
设置随机种子并运行Boruta特征选择
- data = dat——使用Boruta算法在数据集dat上运行
- group~.——表示预测的是dat中的group列
- doTrace = 2——表示在控制台中显示详细的运行信息
- maxRuns = 500——表示指定了算法运行的最大迭代次数为500。
set.seed(123)
boruta.train <- Boruta(group~., data = dat, doTrace = 2, maxRuns = 500)
boruta.train结果如下图所示,运行了499次迭代,耗时20.38秒,其中有7个特征是确定的,有一个特征—LAMB4是不确定的。

修正Boruta结果(这一步是可选的,主要用于修正Boruta算法可能产生的某些不稳定的特征选择结果)
final.boruta <- TentativeRoughFix(boruta.train)
修正后的final.boruta结果如下图所示,其余没什么变化,唯独那个不确定的LAMB4变成了不重要的

接下来从构建好的boruta模型中提取出关键基因并保存
## 结果
boruta_gene <- data.frame(symbol = getSelectedAttributes(final.boruta, withTentative = T))
write.csv(boruta_gene, file = 'boruta_gene.csv')
2. 3 boruta结果简单可视化
接下来一步就是要对boruta结果进行简单可视化,毕竟文章里是要放图的,并且图片展现的效果会更好!!!
# boruta结果简单可视化(内置函数)
{plot(final.boruta, xlab = "", xaxt = "n")lz<-lapply(1:ncol(final.boruta$ImpHistory),function(i)final.boruta$ImpHistory[is.finite(final.boruta$ImpHistory[,i]),i])names(lz) <- colnames(final.boruta$ImpHistory)Labels <- sort(sapply(lz,median))axis(side = 1,las=2,labels = names(Labels),at = 1:ncol(final.boruta$ImpHistory), cex.axis = 0.8)
}
结果如下图所示横坐标为特征(基因名),及随机的影子特征,纵坐标为重要性,绿色的箱子表示通过验证筛选的特征,而红色的箱子表示未通过筛选的特征,蓝色箱子为影子特征,有时候结果还会出现黄色的箱子,那些就代表不确定的特征(可以选择性考虑是否纳入)。

结语:
以上就是boruta算法筛选关键基因的所有过程,如果有什么需要补充或不懂的地方,大家可以私聊我或者在下方评论。
如果觉得本教程对你有所帮助,点赞关注不迷路!!!
与教程配套的原始数据+代码+处理好的数据见配套资源
注:配套资源只要改个路径就能运行,本人已检测过可以跑通,请放心食用,食用过程遇到问题,可先自行百度,实在解决不了可以私信
- 目录部分跳转链接:零基础入门生信数据分析——导读