1写在前面
医院天天叫我们填问卷,我真是不能理解。🫠
动不动就问我们对医院的福利满意吗,对自己的收入满意吗,觉不觉得工作负荷太重了???🙂
我们满不满意,觉不觉得累,医院心里没点数吗!?~~~🤒
不能再说了,再说我的号就要被河蟹掉了。🥸
今天的教程是相对比较基础的了,分享一下我处理Expression matrix时经常遇到的一个小问题,就是重复基因名或者探针名的问题。🤪
这个问题的处理起来也简单也复杂,你可以随机取一个,可以都去掉,可以取最大值,可以取均值,仁者见仁,智者见智吧。🧐
接着是今天的正文,盘点一下我个人常用的几种处理重复基因的方法!~🥳
2用到的包
rm(list = ls())
library(tidyverse)
library(limma)
library(IOBR)
3示例数据
今天是随机生成的数据,20个样本,30个基因。🙃
set.seed(123456)
exprSet <- matrix(runif(600, 1, 10), ncol= 20)
colnames(exprSet) <- paste0("Sample",1:20)
genes <- paste0("Gene", sample(LETTERS, 30, replace=T))
exprSet <- data.frame(genes,exprSet)
DT::datatable(exprSet)
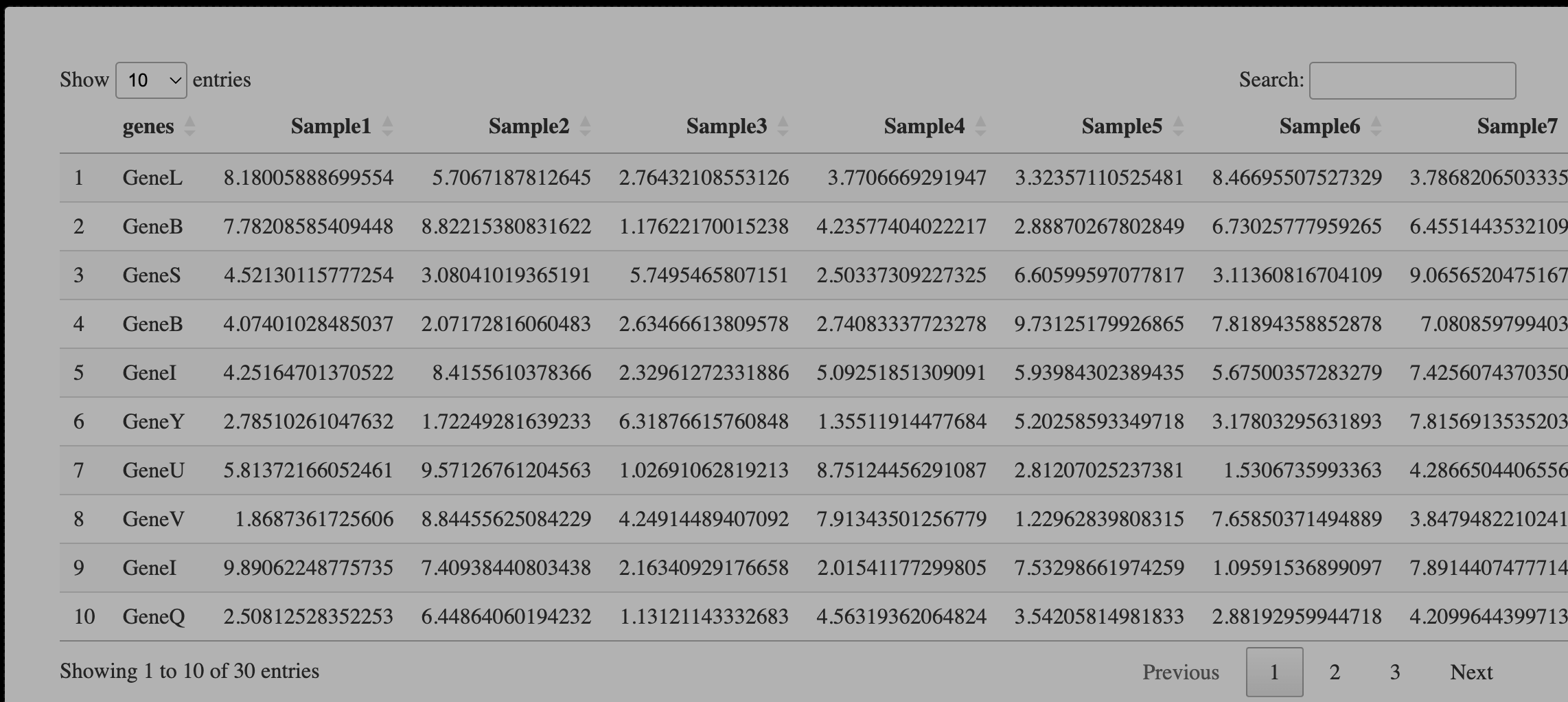
看一下有几个重复的基因吧,10个。😘
table(duplicated(exprSet$genes))

4方法一(取高值)
这里需要注意一下哦,对于相同的基因,应该挑选行平均值大的一整行,而不应该打乱。🥳
index <- order(rowMeans(exprSet[,-1]),decreasing = T)
exprSet_ordered <- exprSet[index,]
keep <- !duplicated(exprSet_ordered$genes)
exprSet_max <- exprSet_ordered[keep,]
DT::datatable(exprSet_max)
再看一下重复的基因去掉了没有,好的,任务完成。😙
table(duplicated(exprSet_max$genes))

5方法二(取均值)
5.1 aggregate函数法
exprSet_mean <- aggregate(.~genes, mean, data = exprSet)
DT::datatable(exprSet_mean)
table(duplicated(exprSet_mean$genes))

5.2 avereps函数法
exprSet_avereps <- avereps(exprSet,ID = exprSet$genes) %>%
as.data.frame()
DT::datatable(exprSet_avereps)
table(duplicated(exprSet_avereps$genes))

6方法三(IOBR包)
最后介绍一下IOBR包的remove_duplicate_genes()函数,非常好用!~🤓
虽然这里method写的是mean,但实际上是挑选了平均值最大的一行,我个人也是比较喜欢这种方法。🤨
exprSet_IOBR_mean <- remove_duplicate_genes(exprSet, column_of_symbol = "genes", method = "mean")
DT::datatable(exprSet_IOBR_mean)
table(duplicated(rownames(exprSet_IOBR_mean)))


点个在看吧各位~ ✐.ɴɪᴄᴇ ᴅᴀʏ 〰
📍 🤣 chatPDF | 别再自己读文献了!让chatGPT来帮你读吧!~
📍 🤩 WGCNA | 值得你深入学习的生信分析方法!~
📍 🤩 ComplexHeatmap | 颜狗写的高颜值热图代码!
📍 🤥 ComplexHeatmap | 你的热图注释还挤在一起看不清吗!?
📍 🤨 Google | 谷歌翻译崩了我们怎么办!?(附完美解决方案)
📍 🤩 scRNA-seq | 吐血整理的单细胞入门教程
📍 🤣 NetworkD3 | 让我们一起画个动态的桑基图吧~
📍 🤩 RColorBrewer | 再多的配色也能轻松搞定!~
📍 🧐 rms | 批量完成你的线性回归
📍 🤩 CMplot | 完美复刻Nature上的曼哈顿图
📍 🤠 Network | 高颜值动态网络可视化工具
📍 🤗 boxjitter | 完美复刻Nature上的高颜值统计图
📍 🤫 linkET | 完美解决ggcor安装失败方案(附教程)
📍 ......
本文由 mdnice 多平台发布