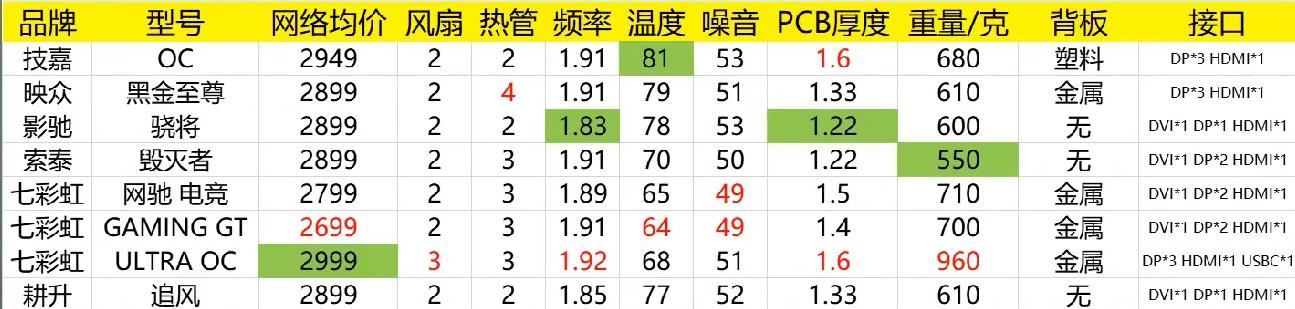
最近看文献的时候看到一张曼哈顿图,是对绵羊进行种间fst的比较找受选择的位点,当时看到这张图就感觉与之前看过的曼哈顿与众不同,图中用线段来表示的具体数值而不是常规的点,看多了点图,感觉线图还挺好看,所以就想复现一下,顺便当记个笔记了。
不知道这种图有没有具体的包,之前见过棒棒糖图,还是很相似的。不过不用考虑那么多,万图皆可ggplot,基本上看到的图都能用ggplot的绘制,所以我就用ggplot复现吧。

主要用到的是ggplot中的geom_segment(),这个函数是用来画线段的,之前我我一般用annotate中的geom='segment’来画线段,因为至此是变量批量的画,所以当然要换成geom_segment(),geom_segment()与常规绘图函数不同,它需要四个坐标,即x,xend,y,yend,我们只需要固定某一方向就可以绘制出想要的线段。
rm(list=ls())
options(stringsAsFactors = F)library(ggplot2)
ggplot()+geom_segment(aes(x=1:5,xend=1:5,y=0,yend=seq(0,0.8,0.2)))+ylim(0,1) #随便找几个数
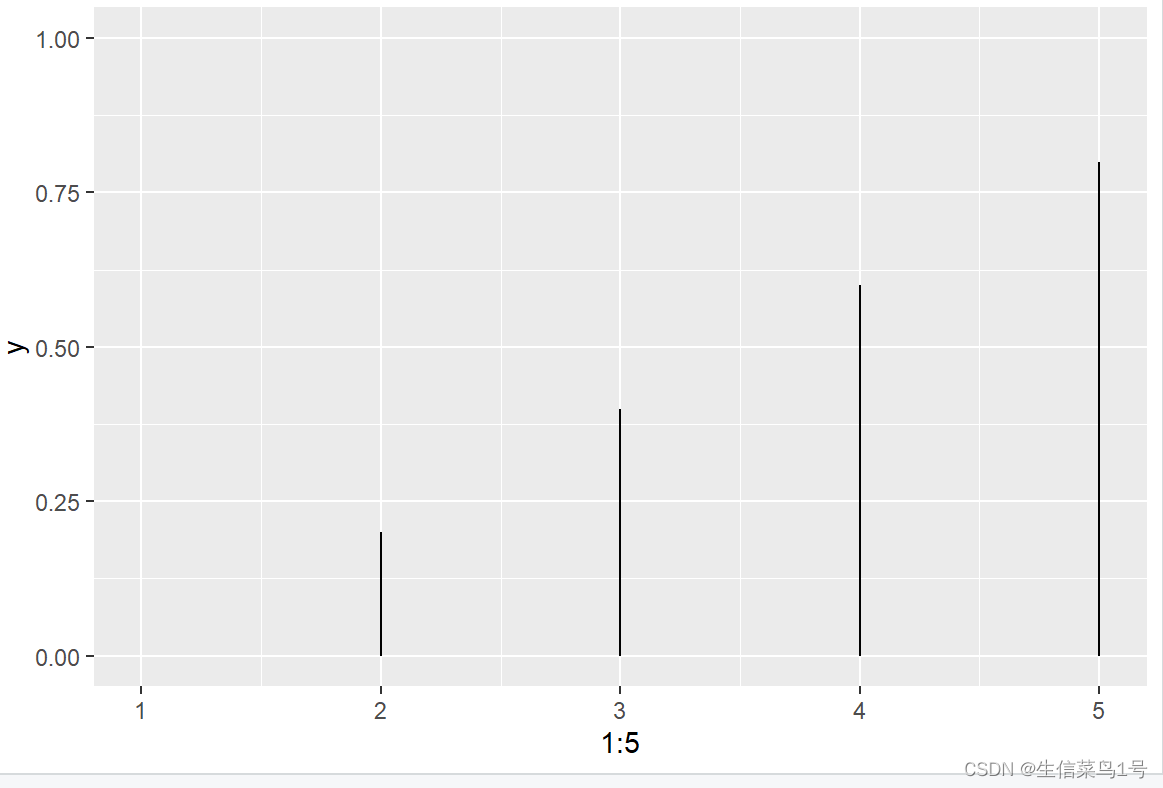
可以看到上图,把x和xend设置为相同,y设置为0,具体大小赋予yend就能产生竖线,这样进一步绘图。
首先就是准备数据,随机产生1万个数据,然后分为500个一组,共20组,并且交替赋予A和B字符,用于颜色分组。
data=data.frame(s1=1:10000,s2=runif(10000,0,0.3)+runif(10000,0,0.5),s3=rep(rep(c('A','B'),10),each=500))
data$s1=as.character(data$s1) #把数值型转换为字符型,用于排列顺序
#runif(min,max,n) 随机生成min到max之间的n个小数
#s1是x轴位置
#s2为y轴大小
#s3为颜色分组

注意s1为x位置,我一般要把它转换为字符型,这样在ggplot中可以进行离散型数据的设置,因为数值型数据是连续的数据,在x轴设置时如果数据中间有缺失部分会导致x轴无法紧密排列,而离散型数据则始终紧密排列。
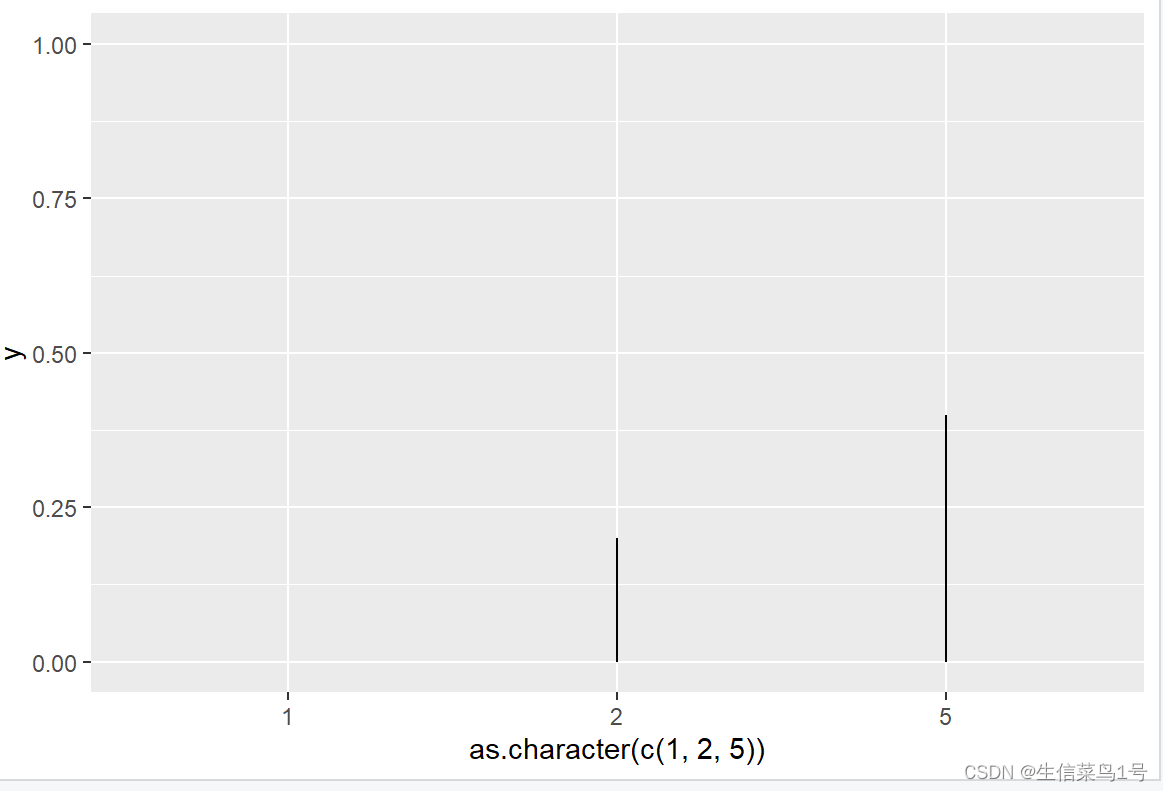
数值型连续数据如下,可以看到根据根据数值大小进行排列,如果中间数据没有连续(2,5),则会产生空缺。

字符型离散数据如下,可以看到自始至终的排列紧密的,虽然只有2和5,但是两者排列紧密。
之后就是绘图
ggplot(data = data)+geom_segment(aes(x =s1, y =0,xend =s1, yend =s2,color=s3),linetype=1,size=0.1)
初步的图就是这样,很粗糙,需要进一步修改。
ggplot(data = data)+geom_segment(aes(x =s1, y =0,xend =s1, yend =s2,color=s3),linetype=1,size=0.1)+scale_color_manual(values = c('#fa450f','#242b66'))+ #颜色设置scale_y_continuous(expand = c(0,0),limits =c(0,1),breaks=seq(0,1,0.2))+scale_x_discrete(expand = c(0,0),breaks=seq(250,10000,500),labels=paste0('chr',1:20),limits=as.character(c(1:10000)))+ #设置x轴breaks和labelstheme_classic()+ #经典主题labs(x='chromosome',y='score')+ #x轴y轴labtheme(legend.position = 'none')+ #去除图例annotate(geom = 'segment',x=0,xend=nrow(data),y=quantile(data$s2,0.95), #添加阈值线yend=quantile(data$s2,0.95),lty=4,color='black')
因为是随机的数据,没有染色体之分,所以我把每500个数据设置为一条染色体,共设置20条染色体。然后添加个阈值吧,用quantile函数找到在数据中前95%的数据大小,用annotate去添加,然后设置一下阈值线样式(lty)和颜色,最后把生成的结果给它拉长压扁一下,用ggsave保存一下就ok了,我感觉这基本还原科,就是数据不够离散,看不出什么峰值,不过也算可以了。

在实际应用的时候需要加入染色体号,这时候就需要先把每条染色体的数据中的位点从小到大排列一下,然后赋予每个位点一个label,然后在scale_x_discrete中设置limits定义一下顺序就好,在每条染色体数据中间再用breaks加上label就ok了。
感觉绘图还是比较简单的,之前讲过CMplot绘制曼哈顿,感觉自己也可以找个时间研究研究,也设计个绘图包,把各种图形的绘制代码集合起来,这可比设置一种新的算法什么的简单太多了,可以尝试。