将几个数据集之间的交集进行可视化,除了可以利用Venn图外,还可以使用upset这种图形。这种方法是发表在2014年nature methods上面。Sets and intersections | Nature Methods
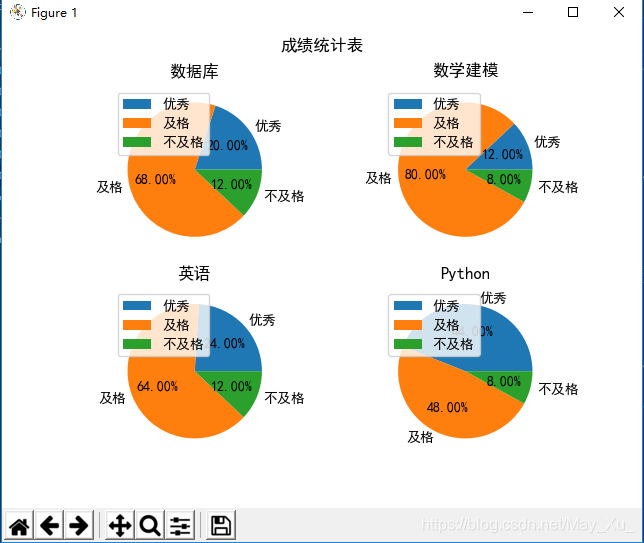
upset图形大致是这个样子
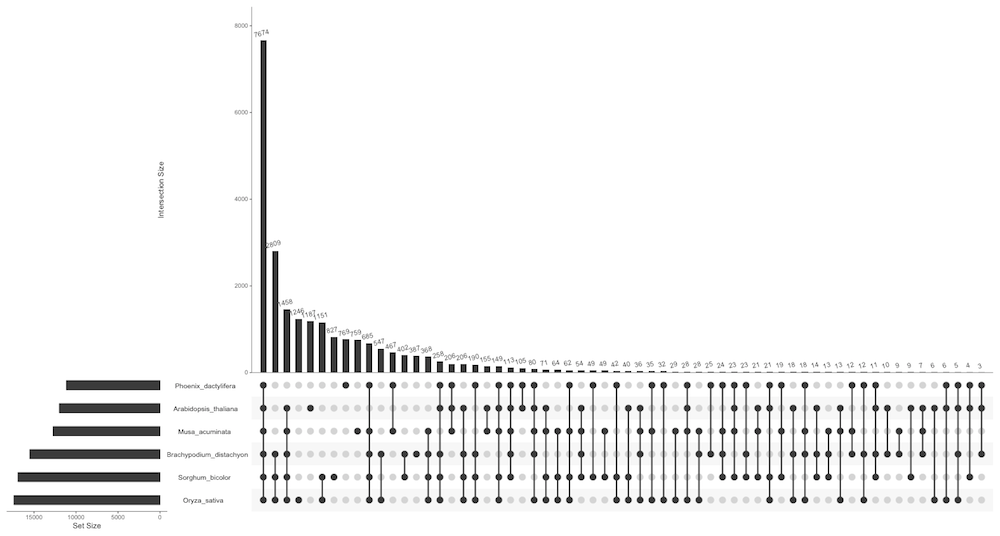
图形基本含义
那怎么观察这种图形,或者图形的意义是什么?
例如下图对这一部分进行了展示

这是UpSet图和Venn图之间的联系
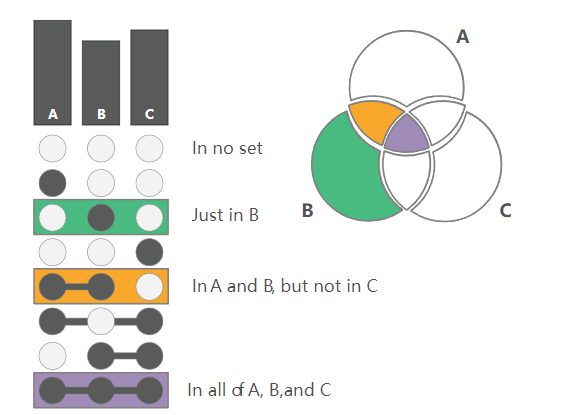
这种图的好处在于,我们可以在矩阵旁边以柱状图的形式绘制交叉点的大小,正如下面的例子中看到

这样可以对交叉点的数量进行直观比较,也可以存储更多的信息。
并且,也可以通过交叉点的数量,来对柱状图进行排序
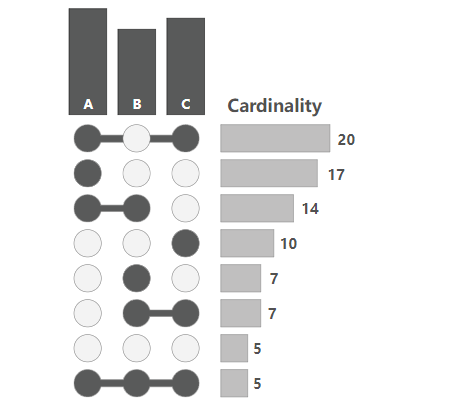
然后将图形按照水平或者竖直方式进行排列即可。大部分文章当中都是用的水平排列

怎么画一个简单的UpSet图形
UpSet图形可以使用R、python、Javascript都可以绘制,这里使用R绘制
载入数据
加载R包
install.packages("UpSetR")
library(UpSetR)
使用的是UpSetR中的数据,
mutations <- read.csv( system.file("extdata", "mutations.csv", package = "UpSetR"), header=T, sep = ",")
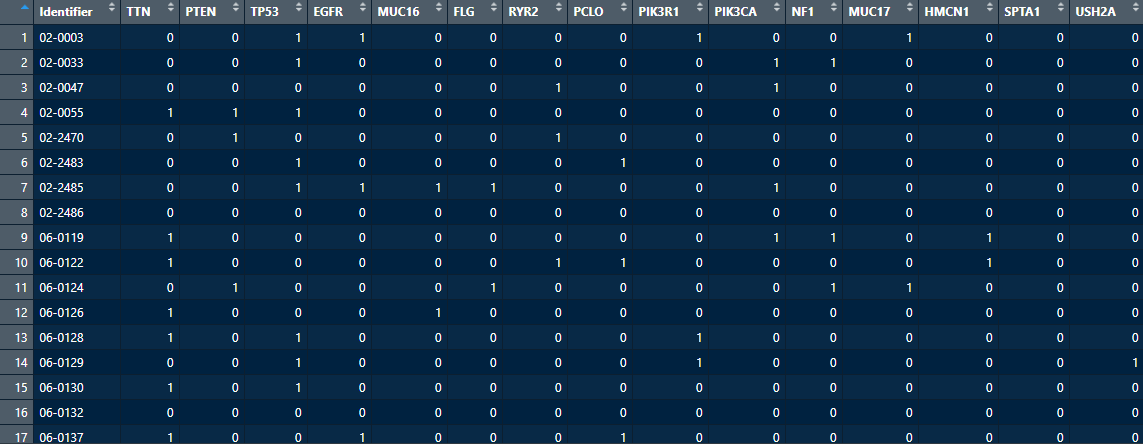
该突变数据集是来自TCGA,代表了多形性胶质母细胞瘤队列中100个突变最多的基因的突变。
行是样本名称,从第2列开始,是基因突变情况
现在就要表现选定的几个基因,有多少突变样本,并且之间的交集如何(也就是某个样本中,几个基因共同突变的情况)。
绘图
upset(mutations, sets = c("PTEN", "TP53", "EGFR", "PIK3R1", "RB1"), sets.bar.color = "#56B4E9",order.by = "freq", empty.intersections = "on")
绘制过程还是很简单

其中的解释,就是第一部分的内容。
当然,可以自行修改,让颜色变得艳丽起来
upset(mutations, sets = c("PTEN", "TP53", "EGFR", "PIK3R1", "RB1"),keep.order = F,sets.bar.color = "red",matrix.color = "blue", shade.color = "gray88", main.bar.color = "brown",color.pal = 3,scale.intersections = "identity")


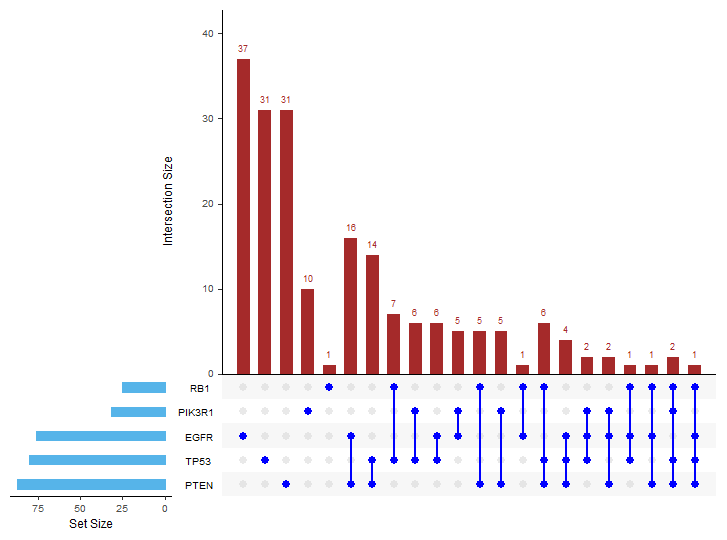
其实没想到,就这样在原版UpSet的基础上,加入TCGA分析,然后也发了文章,真是只要有想法,总能搞些文章。
参考文章
UpSet Home
UpSetR package - RDocumentation
GitHub - hms-dbmi/UpSetR: An R implementation of the UpSet set visualization technique published by Lex, Gehlenborg, et al…
Upsetr | Gehlenborg Lab
Sets and intersections | Nature Methods
Basic Usage (r-project.org)
R 数据可视化 —— 集合可视化 UpSetR - 知乎