前言
本专栏针对的目标物体为物体裂缝量化,提取裂缝的骨架有助于裂缝长度的求解,故这一篇也是本专栏的开篇。
细化算法选择与分析
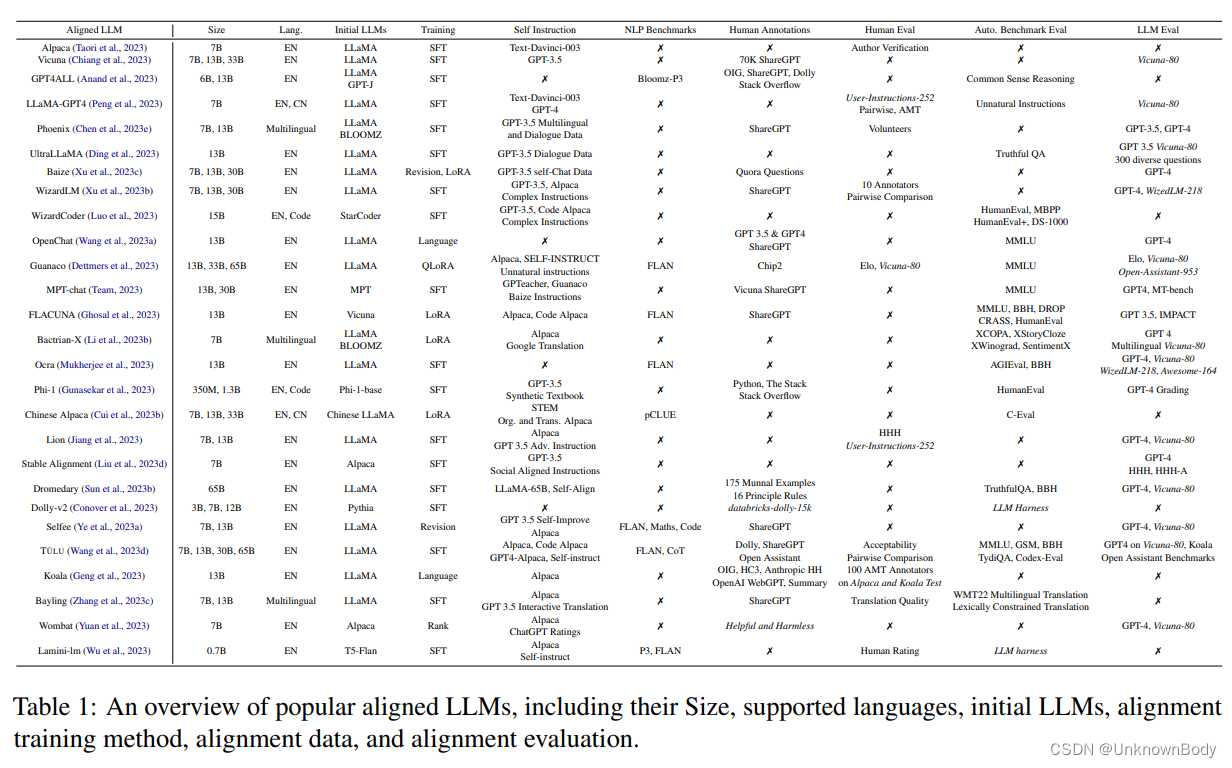
裂缝骨架的提取是十分有必要,如果我们能够得到裂缝的骨架图那么就很容易获得整条裂缝的长度。在当前经典的细化算法,比如Zhang并行细化算法、Hilditch细化算法,Rosenfeld细化算法等都有被广泛应用在骨架提取算法之中。
其中Zhang并行细化算法细化过程简单,也是应用最广的一种,且细化后骨架位于图像中心线上,但是其结果图像往往有较多突起点和毛刺产生。Hilditch是基于二值化图像的基础上进行的,利用串行与并行相结合的方式提取骨架,虽然其细化效果较好,但是该算法判定条件繁复而冗余,导致处理时间较长,考虑到这些因素,故不采用Hilditch。Rosenfeld是一种基于边界追踪的细化算法,与 Zhang-Suen 和 Hilditch 等算法相比,Rosenfeld算法也被广泛应用于骨架提取。它通常产生相对平滑的骨架,较少出现突起点和毛刺。
从效果上来说应当选择Rosenfeld算法,但手写实现与skimage集成的Zhang-Suen算法比较,Zhang-Suen更胜一筹。
环境搭建
从上到下依次安装即可,如果报错没有这个包,就进行安装即可。
安装skimage
pip install scikit-image -i https://pypi.tuna.tsinghua.edu.cn/simple
安装opencv
pip install opencv-python -i https://pypi.tuna.tsinghua.edu.cn/simple
pip install opencv-contrib-python -i https://pypi.tuna.tsinghua.edu.cn/simple
安装matplotlib
pip install -i https://pypi.tuna.tsinghua.edu.cn/simple matplotlib==3.5.2
安装pyzjr
pip install pyzjr -i https://pypi.tuna.tsinghua.edu.cn/simple
裂缝数据集
我们针对的是适用于voc数据集的8位png彩图,从网上可以找到开源数据集进行测试,我这里采用的是裂缝森林的数据集,你可以从Kaggle上找到crackforest | Kaggle。如果你下载的数据集是mat后缀的,可以使用下面这个脚本进行转换。
# Mat2png.pyfrom os.path import isdir
from scipy import io
import os, sys
import numpy as np
from PIL import Imageif __name__ == '__main__':file_path = './groundTruth/'png_img_dir = './groundTruthPngImg/'if not isdir(png_img_dir):os.makedirs(png_img_dir)image_path_lists = os.listdir(file_path)images_path = []for index in range(len(image_path_lists)):image_file = os.path.join(file_path, image_path_lists[index])# print(image_file)#./CrackForest-dataset-master/groundTruth/001.matimages_path.append(image_file)image_mat = io.loadmat(image_file)segmentation_image = image_mat['groundTruth']['Segmentation'][0]segmentation_image_array = np.array(segmentation_image[0])image = Image.fromarray((segmentation_image_array - 1) * 255)png_image_path = os.path.join(png_img_dir, "%s.png" % image_path_lists[index][0:3])image.save(png_image_path)如果你下载的时候就是这样的标签图最好不过了:

但请注意,如果不是自己标注的数据集,就最好要检查它的像素是否正确。

远看没有任何的问题,如果你使用图片查看器或ps放大查看就会看到下面的情况:

这种图片是有问题的,所以必须要进行二值化操作。
裂缝骨架提取
Zhang-Suen我曾经使用过手写版本的,不是超时就是运行报错,而在skimage里面的skeletionize函数就能轻易的实现骨架化,这里调用multifile模式,即可进行文件夹的遍历,将骨架化后的图像保存到另一个文件夹当中。
import numpy as np
from skimage.filters import threshold_otsu,median
from skimage.morphology import skeletonize,dilation,disk
import os
import cv2
from skimage import io, morphologydef sketion(mode='multifile', input_folder='num', output_folder='output', single_pic='num/001.png'):""":param mode: 检测模式——single_pic检测单张图片并保存,multifile检测多张图片并保存:param input_folder: 目标文件夹:param output_folder: 输出文件夹:param single_pic: 用于检测单张图片的路径:return: 返回输出文件夹的路径的骨架图"""if mode == 'single':image = io.imread(single_pic, as_gray=True)# 使用Otsu阈值方法进行二值化处理thresh = threshold_otsu(image)binary = image > threshskeleton = skeletonize(binary)io.imshow(skeleton)io.imsave('output.png', skeleton)io.show()elif mode == 'multifile':if not os.path.exists(output_folder):os.makedirs(output_folder) # 如果输出文件夹不存在,就创建它for filename in os.listdir(input_folder):if filename.endswith('.jpg') or filename.endswith('.png'):image = io.imread(os.path.join(input_folder, filename), as_gray=True)thresh = threshold_otsu(image)binary = image > threshbinary = dilation(binary, disk(3))binary = median(binary, selem=morphology.disk(5))# 效果不错binary = dilation(binary, disk(2))binary = median(binary, selem=morphology.disk(5))# 添加闭运算selem = morphology.disk(3)binary = morphology.closing(binary, selem)skeleton = skeletonize(binary)output_filename = os.path.join(output_folder, filename)io.imsave(output_filename, skeleton)return output_folder这里先进行一个简单的测试。读取对应文件夹下的图片,看效果如下:
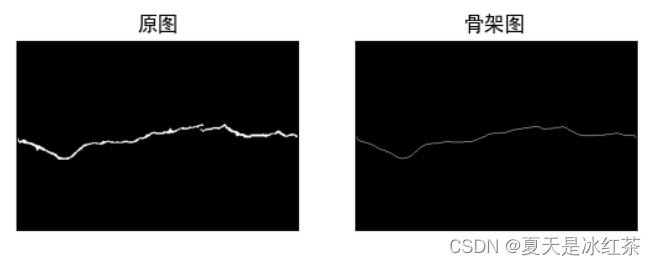
import cv2
import pyzjr as pz
from matplotlib import pyplot as plt
img1=cv2.imread("./num/001.png")
img2=cv2.imread("./output/001.png")
stackedimg=pz.Stackedtorch([img1,img2],1,2,["原图","骨架图"])
plt.show()注意
这里的中值滤波median是我经过多种滤波测试后获得的较好的结果,你应当在自己的数据集上进行测试,包括膨胀与闭运算的操作,找到适用于自己的数据集上组合。
![政府大数据资源中心建设总体方案[56页PPT]](https://img-blog.csdnimg.cn/img_convert/04510f953ab933d682ce720b2d4c84b9.jpeg)