步骤1:点击资源,点击Java下载
https://www.oracle.com/

步骤2:点击java下载、JDK23下载,下载第一行第一个

步骤3:解压到一个空文件夹下,复制lib地址

步骤4:在设置里面搜索“高级系统设置”;点击环境变量
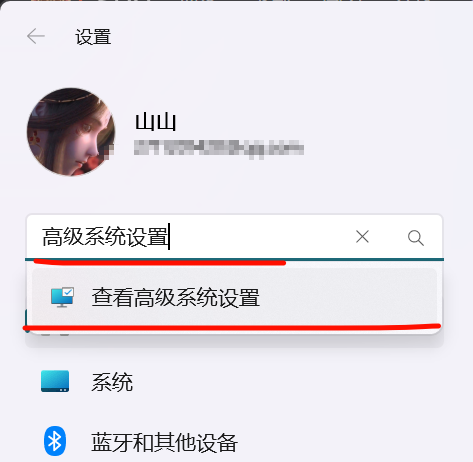

步骤5:点击新建ANDROID_HOME,位置是Android Studio SDK的位置
点击新建JAVA_HOME,位置是

步骤6:找到用户变量里Path点击编辑,新建%JAVA HOME%lbin


步骤7:检查是否配置成功
查找cmd以管理员身份运行,出现类似java verslon“1.8.0 291”,就是配置成功了








![[SAP ABAP] SE11 / SE16N 修改标准表(慎用)](https://i-blog.csdnimg.cn/direct/2b2782e32e734b7d9c11969e9bdfd30b.png)







![[ESP32:Vscode+PlatformIO]新建工程 常用配置与设置](https://i-blog.csdnimg.cn/direct/27f56a93effa4b05a4774be6190a93ae.png#pic_center)



