论文标题:Improving the generalizability of protein-ligand binding predictions with AI-Bind
论文地址:Improving the generalizability of protein-ligand binding predictions with AI-Bind | Nature Communications
代码:
Barabasi-Lab/AI-Bind: Improving the generalizability of protein-ligand binding predictions with AI-Bind | Zenodo
一、问题
虽然分子动力学和对接模拟经常用于识别潜在的蛋白质-配体结合,但模拟的计算复杂性(即运行时间)和缺乏3D蛋白质结构极大地限制了大规模测试的覆盖范围和可行性。因此,人们提出了基于机器学习(ML)和人工智能(AI)的模型来规避现有方法的计算限制。
二元分类的成功训练需要正样本,即已知相互结合的蛋白质和配体对,通常从蛋白质-配体结合数据库中提取,如DrugBank, BindingDB, Tox21, ChEMBL或药物靶标共用(Drug Target Commons, DTC)。训练还需要负样本,即不相互作用或仅弱相互作用的成对。然而,与不同蛋白质和配体相关的正负样本分布不均,一些蛋白质和配体的正样本多于负样本。
ML模型从蛋白质-配体相互作用网络中节点的程度来学习结合模式,而忽略了相关的节点元数据,如配体的化学结构或蛋白质的氨基酸序列。当预测新的(即从未见过的)蛋白质靶点和配体之间的结合时,ML模型的性能会下降。
二部网络将结合信息表示为具有两种不同类型节点的图:一种对应于蛋白质(也称为靶标,例如代表人类或病毒蛋白质),另一种对应于配体(代表潜在的药物或天然化合物)。蛋白质-配体注释(protein-ligand annotation),在二部网络中被表示为蛋白质和配体之间的联系。
连接到蛋白质或配体的注释的数量遵循 fat-tailed分布,这表明绝大多数蛋白质和配体只有少量注释。
此外,通过对解离常数Kd等动力学常数施加阈值来确定正负注释。如果与蛋白质配体对相关的动力学常数小于设定的阈值,将该对视为正样品或结合样品;否则,这对将被标记为负的或非绑定的。Kd在记录中不是随机分布的,这种不平衡促使ML模型在进行结合预测时利用程度信息(正负注释),而不是从分子结构中学习结合模式。
为了研究拓扑捷径的出现,对于具有注释数量ki的每个节点i,通过度的量化可用训练信息的平衡:

由于大多数蛋白质和配体缺乏结合或非结合注释(表1),因此得到的{ρi}接近1或0,这些ρ值代表预测问题中的注释不平衡。
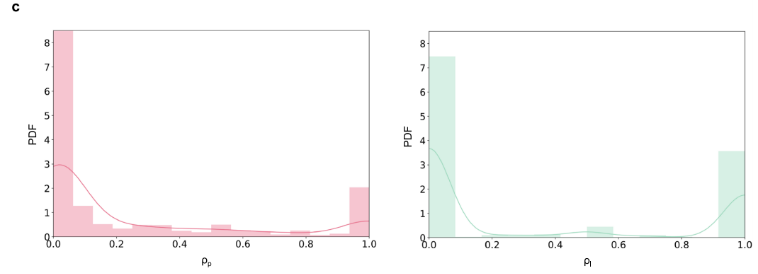
由于许多最先进的深度学习模型,如deepurpose,统一采样可用的正注释和负注释,它们将更高的结合概率分配给具有更高ρ的蛋白质和配体:
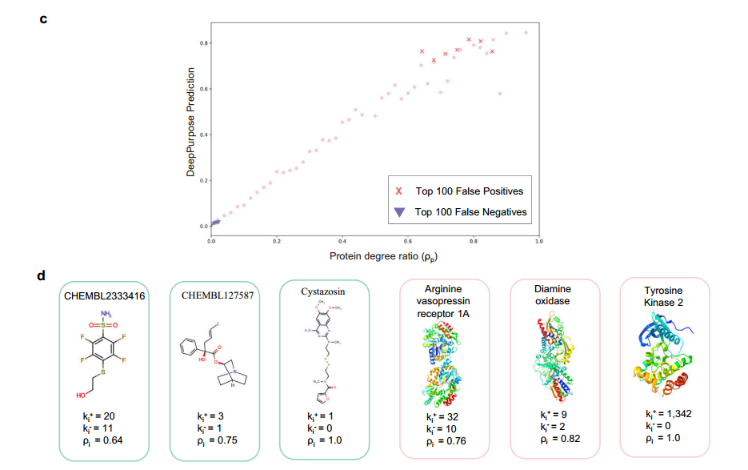
因此,interaction预测是由蛋白质-配体网络中的拓扑捷径驱动的,这与训练数据中存在的正注释和负注释相关,而不是表征蛋白质和配体的结构特征。
通过仅利用来自网络度序列的拓扑约束来预测结合的可能性。在配置模型(图3a,方法)中,观察到链路的概率仅由其末端节点的度决定。

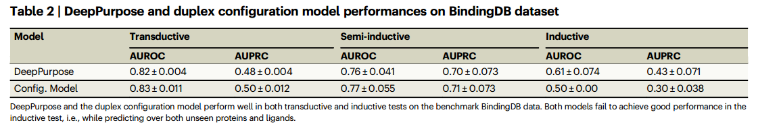
在BindingDB的5倍交叉验证中,表现最好的DeepPurpose架构Transformer-CNN5达到AUROC of 0.86 (±0.005) and AUPRC of 0.64 (±0.009),仅依赖于Configuration Model的性能与深度学习模型一样好,证实了蛋白质-配体相互作用网络的拓扑结构驱动预测。
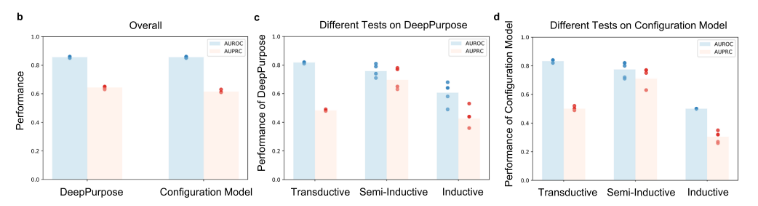
测试了三种不同的场景:(i)看不见的边缘(转导测试),当测试数据集中的蛋白质和配体都存在于训练数据中;(ii)看不见的目标(半感应测试),当只有来自测试数据集的配体存在于训练数据中时;(iii)看不见的节点(归纳测试),当测试数据集中的蛋白质和配体在训练数据中都不存在时
发现deepurpose和配置模型在场景(i)和(ii)中都表现良好:
对于诱导测试场景(iii),当面对新的蛋白质和配体时,两者的性能都显着下降:
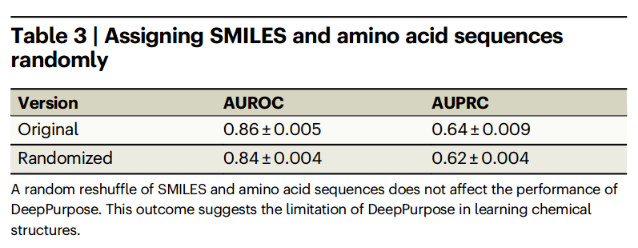
同时,随机洗牌了训练集中的化学SMILES和氨基酸序列,同时保持每个节点相同的正负注释,这一操作不会改变测试性能:
这些测试证实了deepurpose利用网络拓扑作为学习捷径,无法将预测推广到训练数据之外的蛋白质和配体。
二、模型方法(别人提出的基础模型)
1、Data preparation
基于动力学常数Ki、Kd、IC50和EC50,认为来自BindingDB和DTC的样品是结合的或非结合的。分别使用≤103 nM和≥106 nM的阈值来获得阳性和(绝对)阴性注释,
Positive samples.
认为DrugBank的binding信息是阳性样本。为了获得更多的药物阳性样本,使用InChIKeys在BindingDB中进行了搜索。从BindingDB中获得了4330个与DrugBank中的药物相关的结合注释。总体而言,共收集了28188份药物阳性样本。
Network-derived negative samples.
随机选择了间隔7 hops的蛋白质配体对的子集作为阴性样本,以在训练数据中的阳性和阴性样本之间建立总体类别平衡。对≥11跳的远距离配对进行了测试和验证。此外,通过对动力学常数(Ki、Kd、IC50和EC50)取阈值,在测试和验证中包括了从BindingDB导出的绝对非结合对。
2、Novel deep learning architectures
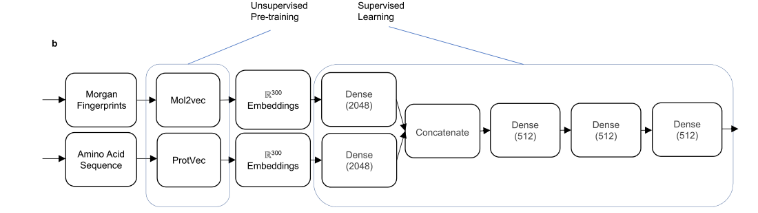
pipeline与各种神经架构兼容,这里提出三种: VecNet, Siamese和VAENet
VecNet.
VAENet.
将配体嵌入潜在空间。Morgan指纹被直接馈送到卷积层,通过最小化信息损失来创建潜在空间嵌入,同时从潜在表示重建分子。对ZINC数据库中的950万种化学物质以及结合数据集中的所有药物和天然化合物进行了可变自动编码器的训练。使用ProtVec进行target嵌入。
Siamese model.
使用一次性学习方法将配体和蛋白质嵌入同一空间。构建了蛋白质靶标、非结合配体、结合配体形式的三元组,并训练模型以找到一个嵌入空间,该嵌入空间最大化非结合对之间的Euclidean距离,同时最小化结合对的Euclidean距离。
三、AI-Bind and statistics across models
1、输入
配体:将同分异构体SMILES作为输入。考虑了一个由DrugBank中可用的所有药物分子和Natural compounds in Food Database (NCFD)中天然存在的化合物组成的搜索空间,并且可以通过利用更大的化学文库(如PubChem)进行扩展。
蛋白质:使用从蛋白质数据库protein Data Bank (PDB)、Universal protein knowledgebase (UniProt)和GeneCards中检索到的氨基酸序列作为输入。
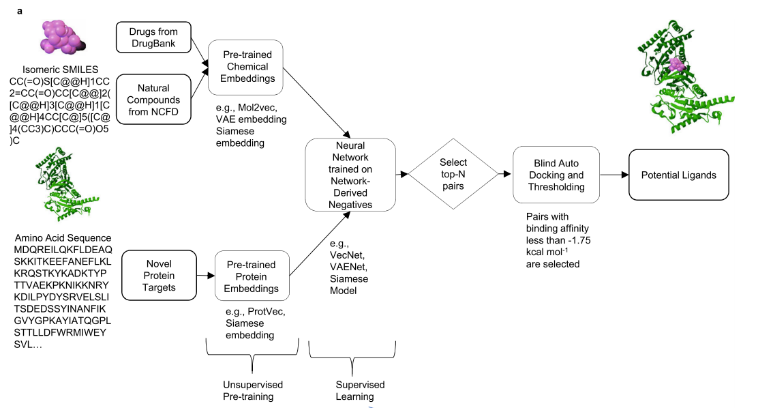
2、模型获益点
(1)依赖于网络衍生的阴性来平衡每种蛋白质和配体的阳性和阴性样品的数量。具体而言,使用最短路径距离≥7的蛋白质-配体对作为负样本,确保神经网络同时观察到每种蛋白质和配体的结合和非结合示例。

(2)在无监督预训练期间,使用在更大的化学和蛋白质结构集合上训练的节点embedding,而不是已知结合注释的集合。允许AI-Bind学习更广泛的结构模式。事实上,像deepurpose这样的模型是在BindingDB中提供的862337个配体和7504种蛋白质上训练的,或者在DrugBank中提供的7307个配体和4762种蛋白质上训练的,而AI-Bind的VecNet中的无监督表示是在ZINC和ChEMBL数据库中的1990万种化合物上训练的,在Swiss-Prot中的546790种蛋白质上训练的。
3、系统性比较
比较AI-Bind与DeepPurpose的性能以及配置模型在5倍交叉验证上的性能:
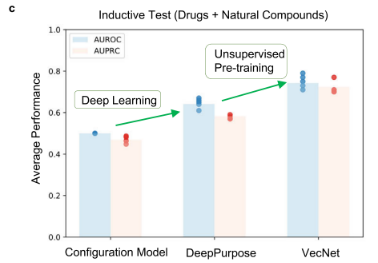
配置模型在感应测试中表现不佳(AUROC为0.5,AUPRC为0.464±0.017)。由于网络衍生的负值消除了注释不平衡,DeepPurpose对新蛋白质和配体的识别性能有所提高(AUROC为0.646±0.023,AUPRC为0.576±0.009)。AI-Bind的VecNet在未见节点上表现最佳,AUROC为0.75±0.032,AUPRC为0.718±0.029。
数据集推广到天然存在的化合物,与药物相比具有复杂的化学结构和更少的训练注释:
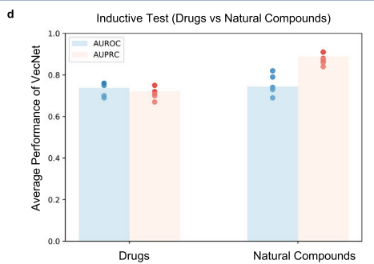
除了deepurure, AI-Bind的VecNet与MolTrans (AUROC 0.612±0.028,AUPRC 0.478±0.034)相比,始终具有更好的性能(AUROC 0.75±0.032,AUPRC 0.718±0.029)
4、Validation of AI-Bind predictions on COVID-19 proteins
将预测与分子对接模拟、蛋白质-配体结合的体外和临床结果进行了比较。SARS-CoV-2病毒蛋白和332种SARS-CoV-2病毒蛋白靶向的人类蛋白作为验证集。这些蛋白质在AI-Bind的训练数据中缺失,因此代表新的靶标,并允许依靠最近了解的COVID-19知识来验证AI-Bind的预测。从UniProt中检索了16个SARS-CoV-2病毒蛋白和330个人蛋白的FASTA格式氨基酸序列,并将其作为AI-Bind的VecNet输入。
目标是预测药物库中的药物或天然存在的化合物,这些化合物可以结合与COVID-19相关的16种SARS-CoV-2或330种人类蛋白质中的任何一种,从而可能破坏病毒感染。
在根据AI-Bind的VecNet (pVecNet ij)预测的结合概率对所有蛋白质配体对进行分类后,我们使用AutoDock Vina进行盲对接模拟,测试预测的前100个和后100个结合相互作用,该模拟通过考虑3D蛋白质结构上所有可能的结合位置来估计结合亲和力。
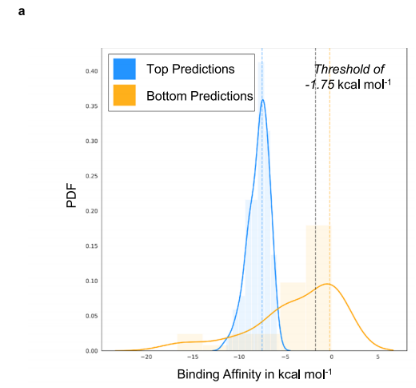
在前100对和后100对预测对中存在的54个蛋白质中,23个在PDB和UniProt中具有3D结构,59个相关配体结构中有51个在PubChem中可用,允许我们执行128个对接模拟(84个涉及顶部预测,44个涉及底部预测)。发现AI-Bind的84个预测中有74个确实是经过验证的绑定对。此外,发现VecNet预测的中位数结合亲和力为- 7.65 kcal mol-1,而底部的预测为- 3.0 kcal mol-1
第二个测试,使用- 1.75 kcal mol−1的结合亲和值和p上对应于归纳测试集上最高F1-Score的最佳阈值,从对接和AI-Bind预测中获得二元标签(结合或非结合)。在推导的混淆矩阵中,观察到sensitivity= 0.76,代表AI-Bind做出的结合预测中真正结合的部分,即true positive /(true positive + False Negatives)的比率,F1-Score = 0.82。这证实,与随机选择相比,AI-Bind预测提供的rank表与binding affinities获得的rank表具有显著的相似性:
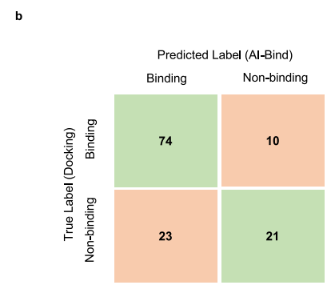
进一步通过在5折设置中随机选择20对蛋白质配体对来检验这些性能指标的稳定性,观察F1-Score = 0.90±0.02。此外,发现AI-Bind的VecNet 预测和对接得到的蛋白-配体结合自由能(ΔG)与rspearman呈反相关,ΔG = -0.51。由于较低的绑定亲和值对应于较强的绑定,这些结果证明了AI-Bind预测和对接模拟之间的一致性。在50个平均结合概率最高的配体中,发现了两种fda批准的药物Anidulafungin (NDA#021948)和Cyclosporine (ANDA#065017)
AI-Bind还提供了几种具有潜在治疗意义的新预测。例如,它预测天然存在的化合物Spironolactone, Oleanolic acid, and Echinocystic acid是COVID-19蛋白的潜在配体,这三种配体都与含有三方基元的蛋白(Trim)结合,后者是SARS-CoV-2病毒蛋白开放阅读框3a (Orf3a)和非结构蛋白9 (Nsp9)结合的人类蛋白。AutoDock Vina支持这些预测。
5、Identifying active binding sites
除了预测结合概率,AI-Bind还可以用于识别氨基酸序列上可能的活性结合位点,即使没有三维蛋白质结构。具体来说,可以用来确定氨基酸序列中哪些氨基酸三元组在结合预测中发挥最重要的作用,指示潜在的蛋白质-配体结合位置。通过扰动了序列中的每个氨基酸三元组,并观察了AI-Bind预测的变化,获得的结合概率曲线中的Valleys 值代表了氨基酸序列上最能预测结合位置的三元组。为了验证AI-Bind预测的结合位点,重点研究了人类蛋白质Trim59,已经从多次对接模拟中获得了这种蛋白质的结果。使用PyMOL50可视化了Trim59上的结合口袋,并鉴定了与配体分子结合的氨基酸残基:
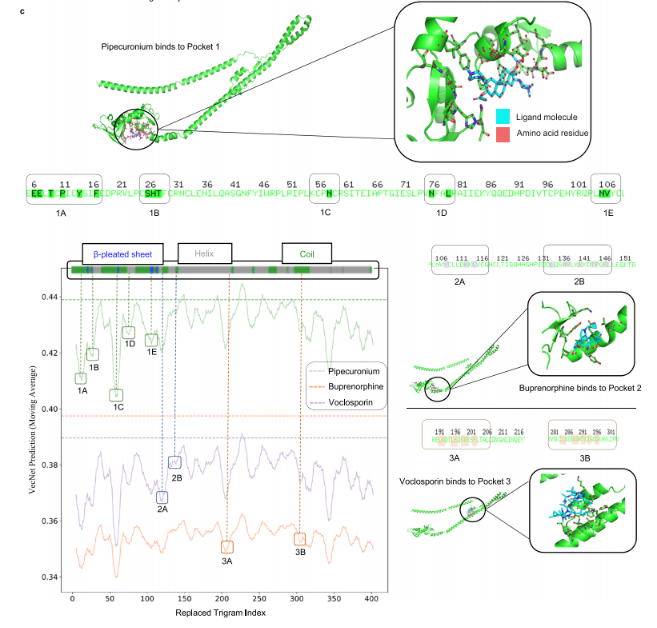
发现负责结合的氨基酸残基直接映射到AI-Bind识别的结合概率分布中的Valleys。通过查看Pipecuronium、Buprenorphine和Voclosporin这三种与Trim59上三个不同口袋结合的配体的对接结果,在结合概率分布图上标记了与各自结合位点对应的谷。例如,Pipecuronium结合的口袋1对应于AI-Bind预测的5个谷,标记为1A、1B、1C、1D和1E。
附模拟过程:File preparation for docking simulations.
对AI-Bind预测的前100和后100中128种蛋白质-配体相互作用使用AutoDock Vina对接模拟:
(1)从PubChem获取SDF格式的3D配体结构,并使用PyMOL将其保存为.pdb格式。pdb格式的3D蛋白质结构,去除水分子,添加所有氢原子,并将Kollman电荷添加到蛋白质中。(2)创建包含整个蛋白质结构的对接网格。这种网格选择确保了blind对接设置,以便考虑蛋白质上的所有位置来确定结合亲和力。(3)为每个蛋白质创建包含网格细节的配置文件,并启动对接模拟。作者认为蛋白质分子是刚性的(rigid),而配体分子是柔性的,也就是说,允许配体有可旋转的键。