文章目录
- 1.Attentive Deep Canonical Correlation Analysis for Diagnosing Alzheimer’s Disease Using Multimodal Imaging Genetics
- 2.Bidirectional Mapping with Contrastive Learning on Multimodal Neuroimaging Data
- 3.CoLa-Diff: Conditional Latent Diffusion Model for Multi-Modal MRI Synthesis
- 4.DBTrans: A Dual-Branch Vision Transformer for Multi-modal Brain Tumor Segmentation
- 5.Gene-induced Multimodal Pre-training for Image-omic Classification
1.Attentive Deep Canonical Correlation Analysis for Diagnosing Alzheimer’s Disease Using Multimodal Imaging Genetics
Zhou R, Zhou H, Chen B Y, et al. Attentive Deep Canonical Correlation Analysis for Diagnosing Alzheimer’s Disease Using Multimodal Imaging Genetics[C]//International Conference on Medical Image Computing and Computer-Assisted Intervention. Cham: Springer Nature Switzerland, 2023: 681-691.
代码开源
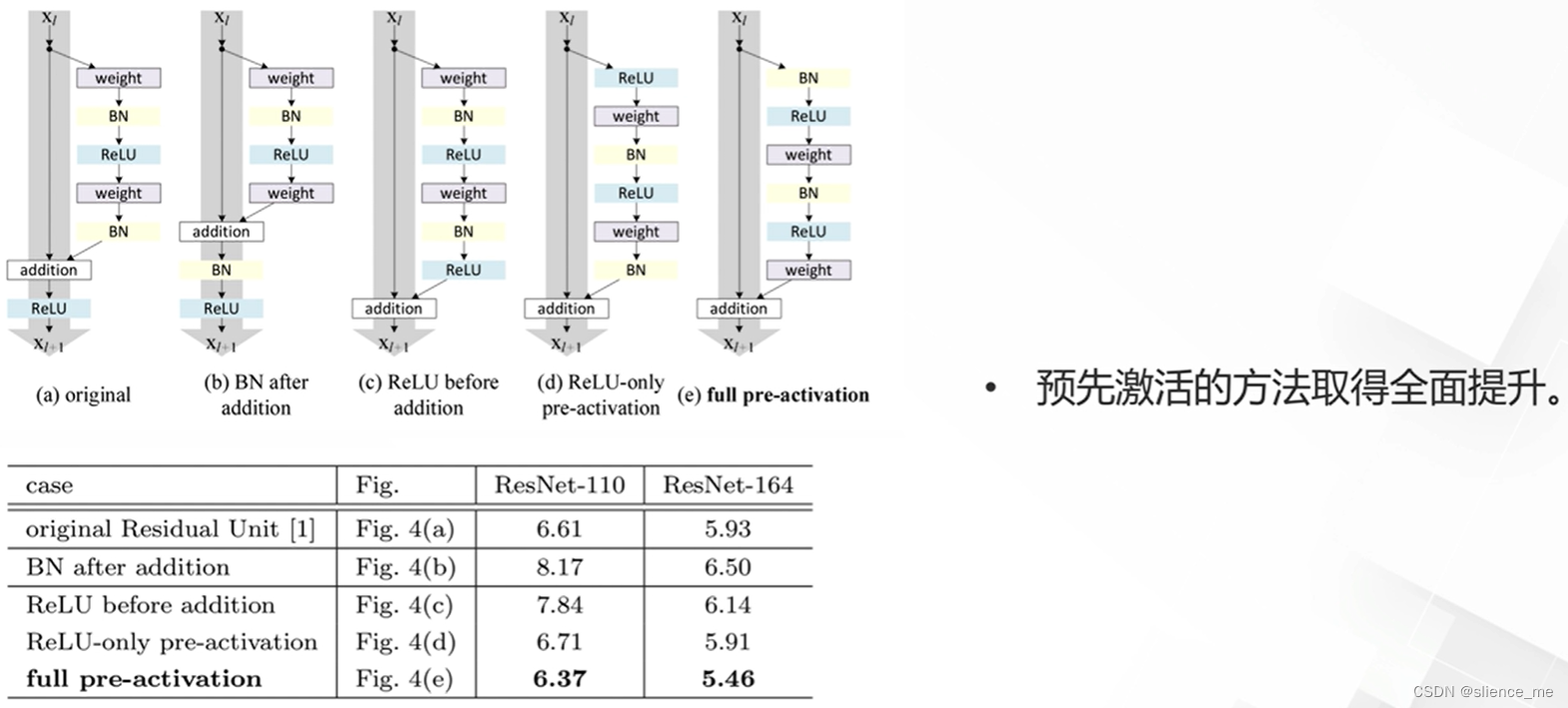
本文提出了一种新的模型——注意力深度典型相关分析(ADCCA),用于诊断阿尔茨海默病。该模型结合了深度神经网络、注意力机制和典型相关分析的优势,以整合和利用多模态脑成像遗传学数据。文章使用了VBM-MRI、FDG-PET和AV45-PET等脑成像模式以及基因SNP数据进行研究,结果表明这种方法可以实现出色的性能,并识别有意义的生物标志物。文章指出,这种方法可以发现与特定基因变异相关的脑区,并可能发现新的多模态生物标志物,影响特定的脑系统,为药物发现提供巨大的推动力。因此,ADCCA模型在阿尔茨海默病的诊断和研究中具有广泛的应用前景。
- 模型概述
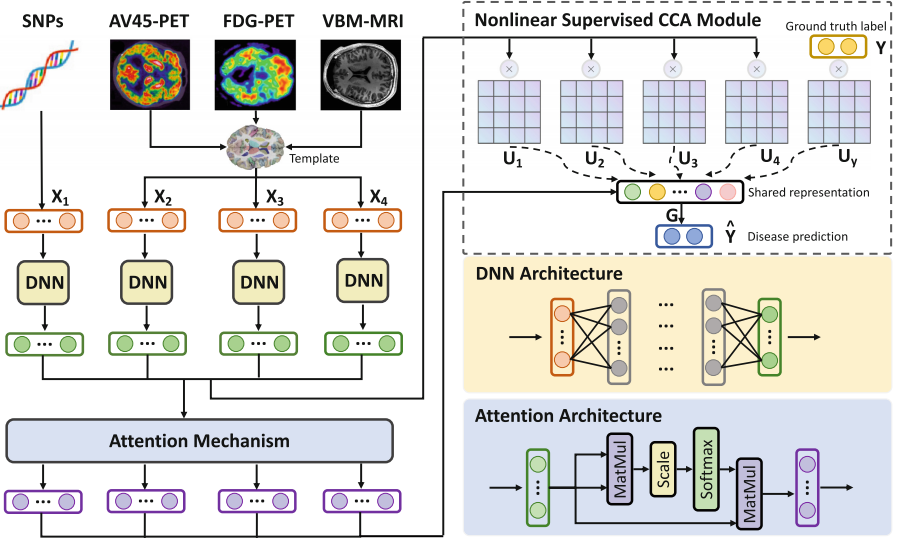
-
输入模态数据: X 1 , . . . , X 4 X_1, ..., X_4 X1,...,X4,这些是输入的不同模态的数据,例如图像、文本等。
-
标签信息: Y Y Y,这是与输入数据相关的标签信息,例如疾病的类别。
-
深度神经网络(DNNs):首先对每个模态的数据进行处理,生成每个模态的隐藏表示。
-
自注意力机制:将这些隐藏表示通过自注意力机制进行处理,生成改进的自注意力表示。
-
投影矩阵:同时,将隐藏表示和标签 Y Y Y分别与基于CCA(canonical correlation analysis,典型相关分析)的投影矩阵 U 1 , . . . , U 4 , U y U_1,..., U_4, U_y U1,...,U4,Uy相乘,将它们映射到一个共享表示 G G G。
-
疾病预测:最后,通过使用自注意力表示、投影矩阵和共享表示 G G G来计算疾病预测。
这个框架的主要优点是能够有效地整合来自不同模态的信息,并通过自注意力机制和投影矩阵来提高预测的准确性
-
数据集
模型训练用的数据集是ADNI(阿尔茨海默病神经影像倡议组织)数据库,该数据库包含了多种成像模式(如VBM-MRI、FDG-PET和AV45-PET)和基因SNP数据(http://www.alzgene.org/)。在实验中,数据被分为三组,包括AD vs. HC(阿尔茨海默病与健康对照组)、AD vs. MCI(阿尔茨海默病与轻度认知障碍组)和MCI vs. HC(轻度认知障碍与健康对照组)进行评估。
模型中用的基因SNP数据是从AlzGene数据库获取的。这个数据库记录了与阿尔茨海默病(AD)相关的基因和SNP(单核苷酸多态性)。在筛选这些基因时,通常会选择那些位于或邻近于AD风险基因APOE的SNP。这些基因数据通常是由基因芯片进行测序和分型的,例如Human 610-Quad或OmniExpress Array平台(Illumina,Inc.,San Diego,CA,USA)。在获取这些数据后,还需要进行预处理,包括质量控制和补缺值估计等步骤。
2.Bidirectional Mapping with Contrastive Learning on Multimodal Neuroimaging Data
Ye K, Tang H, Dai S, et al. Bidirectional Mapping with Contrastive Learning on Multimodal Neuroimaging Data[C]//International Conference on Medical Image Computing and Computer-Assisted Intervention. Cham: Springer Nature Switzerland, 2023: 138-148.
开放源码
本文提出了一种新型的双向映射模型——BMCL( Bidirectional Mapping with Contrastive Learning),通过ROI级别的对比学习减少结构与功能单向映射的偏差。该模型结合了BOLD((blood-oxygelevel-dependent)信号和结构网络信息,能够更准确地预测神经退行性疾病和临床表型。在公开数据集上的评估显示了BMCL的优越性,有助于深入理解神经退行性疾病的发病机制并发现新的生物标志物。
多模态方法在神经影像学中应用广泛,结合不同种类的神经影像数据,如结构MRI和功能MRI,以更全面地评估大脑健康状况。这种方法不仅可以提供关于大脑结构变化的信息,还可以提供关于大脑活动状态的信息。因此,多模态方法是神经影像学中有效的研究方法。
整合多源信息可以提高预测准确率,例如结合图像和文本数据可以提高图像分类和聚类任务的表现。在脑成像研究中,许多研究通过建模功能MRI与其结构对应物之间的通信来表达多模态MRI数据。虽然这些研究主要关注建立单向映射,但这种单向映射方法可能会产生偏差。
总之,通过结合多模态信息和双向映射方法,可以更准确地理解大脑结构和功能之间的相互作用,提高疾病和临床表型的预测准确率。这种方法有助于深入探究神经退行性疾病的发病机制并发现新的生物标志物,从而改善诊断和治疗。
-
模型概述

上图是使用对比学习进行双向映射的框架。该框架包括两个编码器-解码器结构,一个用于从结构网络构建BOLD信号,另一个执行相反的映射。在编码器和解码器之间使用ROI级别的对比学习,以最小化两个重构映射之间的潜在空间差异。最后,使用多层感知机(MLP)进行任务预测。
3.CoLa-Diff: Conditional Latent Diffusion Model for Multi-Modal MRI Synthesis
Jiang L, Mao Y, Wang X, et al. CoLa-Diff: Conditional latent diffusion model for multi-modal MRI synthesis[C]//International Conference on Medical Image Computing and Computer-Assisted Intervention. Cham: Springer Nature Switzerland, 2023: 398-408.
[【开放源码】](https://github.
com/SeeMeInCrown/CoLa Diff MultiModal MRI Synthesis)
本文提出了一种名为CoLa-Diff的新型多模态MRI合成模型,通过利用扩散模型,结合先验知识引导扩散过程,并自动调整权重有效利用多模态信息。相比传统方法,CoLa-Diff更好地平衡了多种条件,解决了潜在的压缩和噪声问题,并更好地保留了MRI中的解剖结构。实验结果表明,CoLa-Diff在多模态MRI合成方面具有出色的性能,有望成为有效的多模态MRI合成工具。
本文还介绍了该方法如何通过使用eanatomical结构和脑区掩码作为密度分布的先验来指导扩散过程。这种方法有助于更好地保持解剖结构,提高合成图像的质量。此外,CoLa-Diff还具有自监督损失函数,可以更好地衡量合成图像与原始图像之间的差异,进一步优化合成结果。
实验结果表明,CoLa-Diff在多模态MRI合成方面具有显著优势。与先进的MRI合成方法相比,CoLa-Diff能够更好地平衡多种条件,解决潜在的压缩和噪声问题,并更好地保留MRI中的解剖结构。这些优势使得CoLa-Diff成为一种具有潜力的多模态MRI合成工具,为医学图像处理领域提供了新的思路和方法。
- 模型概述
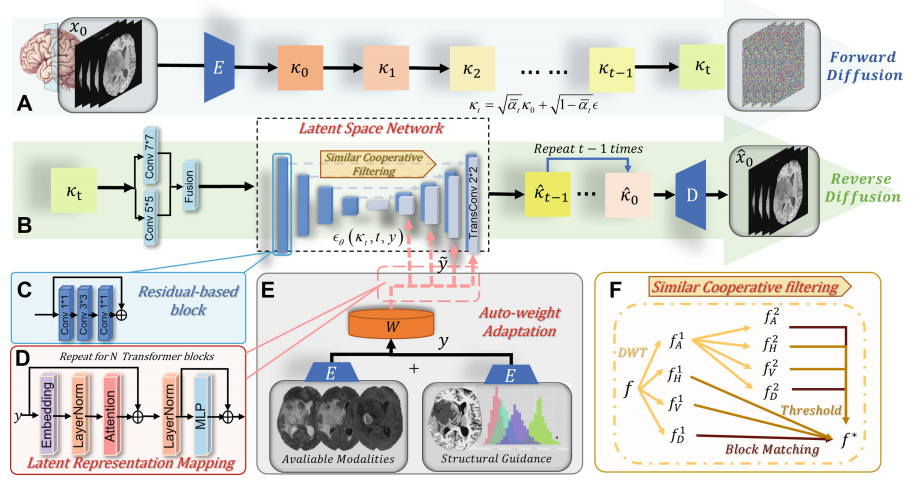
上图是CoLa-Diff模型的网络架构图。CoLa-Diff是一个基于深度学习的多模态MRI合成模型,该模型的主要贡献是提出了一种新颖的神经网络架构来解决多模态MRI合成的难题。具体来说,CoLa-Diff模型将多模态MRI的合成问题转化为一个条件下的扩散过程,并利用深度学习技术对扩散过程进行建模。
在图1中,CoLa-Diff模型包括两个主要部分:前向扩散过程和反向扩散过程。在前向扩散过程中,输入的原始图像( x 0 x_0 x0)通过编码器E被压缩成潜在空间中的表示( k 0 k_0 k0),然后通过添加噪声进行扩散,得到一系列潜在空间中的表示( K t , t = 1 , . . . , T K_t, t=1,...,T Kt,t=1,...,T)。在反向扩散过程中,通过一个潜在空间网络( ϵ θ \epsilon_θ ϵθ)对扩散过程中的潜在空间表示进行预测,从而得到最终的合成图像。
此外,CoLa-Diff模型还引入了自适应权重调整模块(Auto-Weight Adaptation),该模块的作用是平衡多个条件,最大化相关信息并最小化冗余信息。具体来说,自适应权重调整模块对编码条件 y y y进行自适应权重调整,得到调整后的编码条件 y ~ \tilde y y~,然后利用调整后的编码条件 y ~ \tilde y y~进行潜在空间网络的预测。
4.DBTrans: A Dual-Branch Vision Transformer for Multi-modal Brain Tumor Segmentation
Zeng X, Zeng P, Tang C, et al. DBTrans: A Dual-Branch Vision Transformer for Multi-Modal Brain Tumor Segmentation[C]//International Conference on Medical Image Computing and Computer-Assisted Intervention. Cham: Springer Nature Switzerland, 2023: 502-512.
[代码开源]
本文提出了一种名为DBTrans的基于Transformer模型,用于解决3D空间对齐的多模态MRI脑肿瘤分割(SAMM-BTS)任务的挑战。传统Transformer模型在处理此类任务时面临两个主要问题:计算复杂度高和对局部特征建模不足。DBTrans通过双分支架构解决了这些问题,既考虑了全局特征建模,又考虑了局部特征建模,降低了计算复杂度。此外,现有模型仅将空间对齐的多模态数据堆叠在通道维度上,DBTrans在模型内部设计中对多通道数据进行处理。
- 模型概述
首先,DBTrans模型采用双分支结构,包括一个编码器和一个解码器。在编码器中,输入的多个模态的医学图像被分割成多个小的窗口,并使用两个注意力机制(即近距离窗口注意力机制和远距离窗口注意力机制)来建模局部和全局特征。这种双分支结构可以使模型同时捕获局部和全局特征,并降低计算复杂度。其次,DBTrans模型使用了一种额外的路径来提高解码过程。除了传统的跳过连接结构外,DBTrans模型在解码器中引入了一个Shifted-W-MCA-based全局分支。这个全局分支可以提供更全面的上下文信息,帮助解码器更好地理解输入图像的内容,从而提高了模型的分割性能。此外,DBTrans模型还采用了多尺度特征融合策略,将不同尺度的特征进行融合,从而提高了模型的鲁棒性和分割精度。
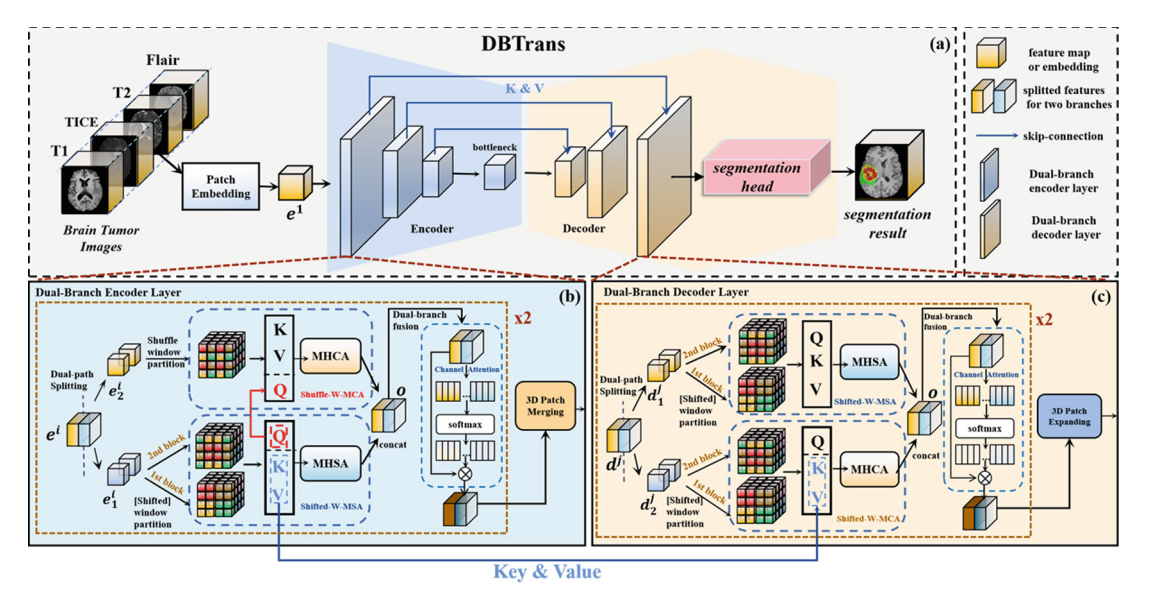
上图是DBTrans的整体框架图,它包括编码器和解码器两部分。
编码器部分,它由四个双分支编码器层(包括一个瓶颈)组成。每个编码器层包含两个连续的编码器块和一个3D patch 合并层,以向下采样特征嵌入。需要注意的是,在瓶颈中只有一个编码器块。编码器块包括一个双分支结构,包含本地特征提取分支,全局特征提取分支,以及一个瓶颈连接。在每个编码器层之后,我们将嵌入向量沿通道维度拆分,得到两个等大的特征映射,然后进行下一步操作。
解码器部分,它以编码器的输出作为输入,并通过三个双分支解码器层和一个分割头来生成最终的预测结果。每个解码器层包含两个连续的解码器块和一个3D patch 扩展层。
此外,基于交叉注意力的跳跃连接被建立在编码器和解码器之间,还应用了一个通道注意力双分支融合模块。
总的来说,DBTrans的整体框架图是基于一个双分支结构构建的,这个结构贯穿于整个网络中,包括编码器和解码器部分。还采用了跳跃连接和通道注意力机制来提高性能。
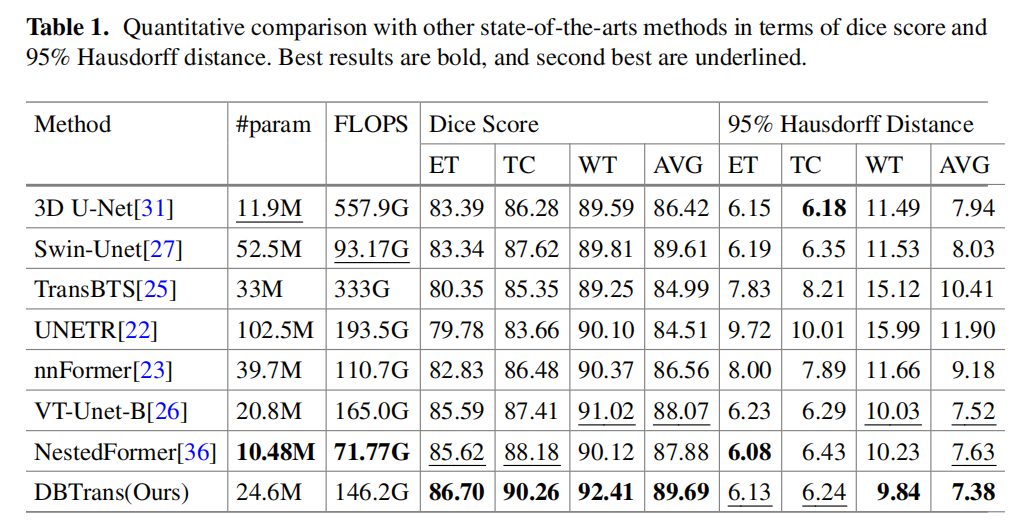
- 模型性能
5.Gene-induced Multimodal Pre-training for Image-omic Classification
Jin T, Xie X, Wan R, et al. Gene-Induced Multimodal Pre-training for Image-Omic Classification[C]//International Conference on Medical Image Computing and Computer-Assisted Intervention. Cham: Springer Nature Switzerland, 2023: 508-517.
[开放源码]
本文提出了一种名为Gene-induced Multimodal Pre-training (GiMP)的框架,旨在解决多模态图像-基因组分类的挑战。该框架联合整合基因组学和全视野数字切片(WSIs),通过 Group Multi-head Self Attention基因编码器和掩码Patch模型范例(MPM)来捕获基因表达队列中的全局结构特征和不同组织中的病理特征。
GiMP采用新型的跨模态学习网络架构,将图像和基因表达数据结合,通过三联学习模块学习高阶相关性和辨别性患者信息。预训练后,采用简单的微调即可获得分类结果。在TCGA数据集上的实验结果表明,该网络架构和预训练框架具有优越性,图像基因表达分类的准确率达到99.47%。本文展示了跨模态学习在病理图像和基因表达数据分析中的巨大潜力,有助于推动精准医学的发展。通过使用跨模态学习,我们可以更好地理解和利用基因表达和病理图像数据,为精准医学提供更准确和可靠的工具。
- 模型概述
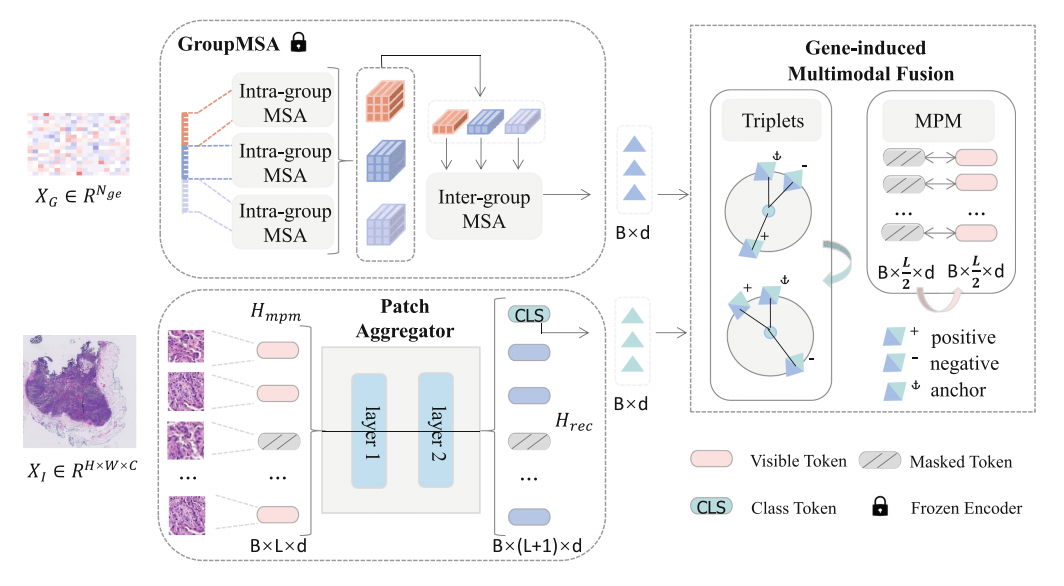
上图是GiMP预训练的示意图。给定一批图像-基因组对数据,随机选择一个固定长度的patch队列,并遮盖patch的一部分。然后使用两个特定于模态的编码器捕捉单模态特征。这里考虑了两个预训练目标:
- 通过合并每个模态的CLS标记构建三元组并按照类别关系增强判别能力。
- 通过其相邻patch重建缺失的patch嵌入。
在第一部分中, group-based genetic encoder GroupMSA(GroupMSA)被用来捕获图像和基因组数据中的特征。将基因组数据按照NLP方式处理:分段->提取嵌入->段内MSA->段间MSA,从而捕捉到它们之间的相关性。
在第二部分中, efficient patch aggregator被用来将来自同一图像或基因组的patch特征聚合在一起。考虑到WSI尺寸太大,使用标准attention开销大,文章使用 Nystrom-based attention algorithm(通过在特征图上采样并计算注意力权重,从而对特征进行加权融合。该算法主要由两部分组成:patch extraction和patch aggregation。在patch extraction阶段,算法从输入特征图中提取出多个patch作为输入;在patch aggregation阶段,算法使用Nystrom方法对这些patch进行加权融合,得到最终的特征表示。)。这有助于在预训练过程中提高模型的泛化能力。
在第三部分中, gene-induced multimodal fusion(gene-induced multi-modal fusion)被用来将来自不同模态的特征进行融合。这个过程使用了跨模态注意力机制,以便将来自图像和基因组数据的特征进行有效的融合。这部分使用两个任务:
(1)Masked Patch Modeling:对WSI进行,重建遮蔽patch的 特征嵌入而不是原始像素
(2)Gene-Induced Triplet Learning:被用来进一步增强图像特征的表示能力。Triplet学习是一种深度学习技术,通常用于学习图像数据的紧凑和有效的表示。在Triplet学习中,模型被提供三个图像(即锚点图像、正图像和负图像),并被训练以区分这三者之间的相似性和差异性。在GiMP中,Gene-Induced Triplet Learning首先使用Transformer网络对基因表达数据进行编码,以捕捉其全局结构特征。然后,这些基因特征被用作指导,以在图像数据中寻找和锚定相关的区域。对于每个图像,Gene-Induced Triplet Learning选择与锚点图像最相关的两个区域,一个作为正样本(与锚点图像更相似),另一个作为负样本(与锚点图像更不相似)。这种选择过程是基于基因表达数据中的信息来进行的,因此可以增强图像特征的表示能力。
GiMP模型通过这三部分的学习,可以有效地捕获图像和基因组数据中的特征,并将它们融合在一起,从而为之后的分类任务提供更有效的特征表示。




![[Docker]四.Docker部署nodejs项目,部署Mysql,部署Redis,部署Mongodb](https://img-blog.csdnimg.cn/19e61c961a3449b99b9dd5d52eb186e5.png)