文章目录
- 计算机辅助药物设计AIDD【小分子专题】
- AIDD概述及药物综合数据库学习
- 机器学习辅助药物设计
- 图神经网络辅助药物设计
- 自然语言处理辅助药物设计
- 药物设计与分子生成
- 计算机辅助药物设计【蛋白质专题】
- 蛋白质数据结构
- 激酶-Kinase相似性学习
- 基于序列的蛋白质属性预测
- 基于结构的蛋白质属性预测
- 蛋白质-配体相互作用预测PLI
计算机辅助药物设计AIDD【小分子专题】
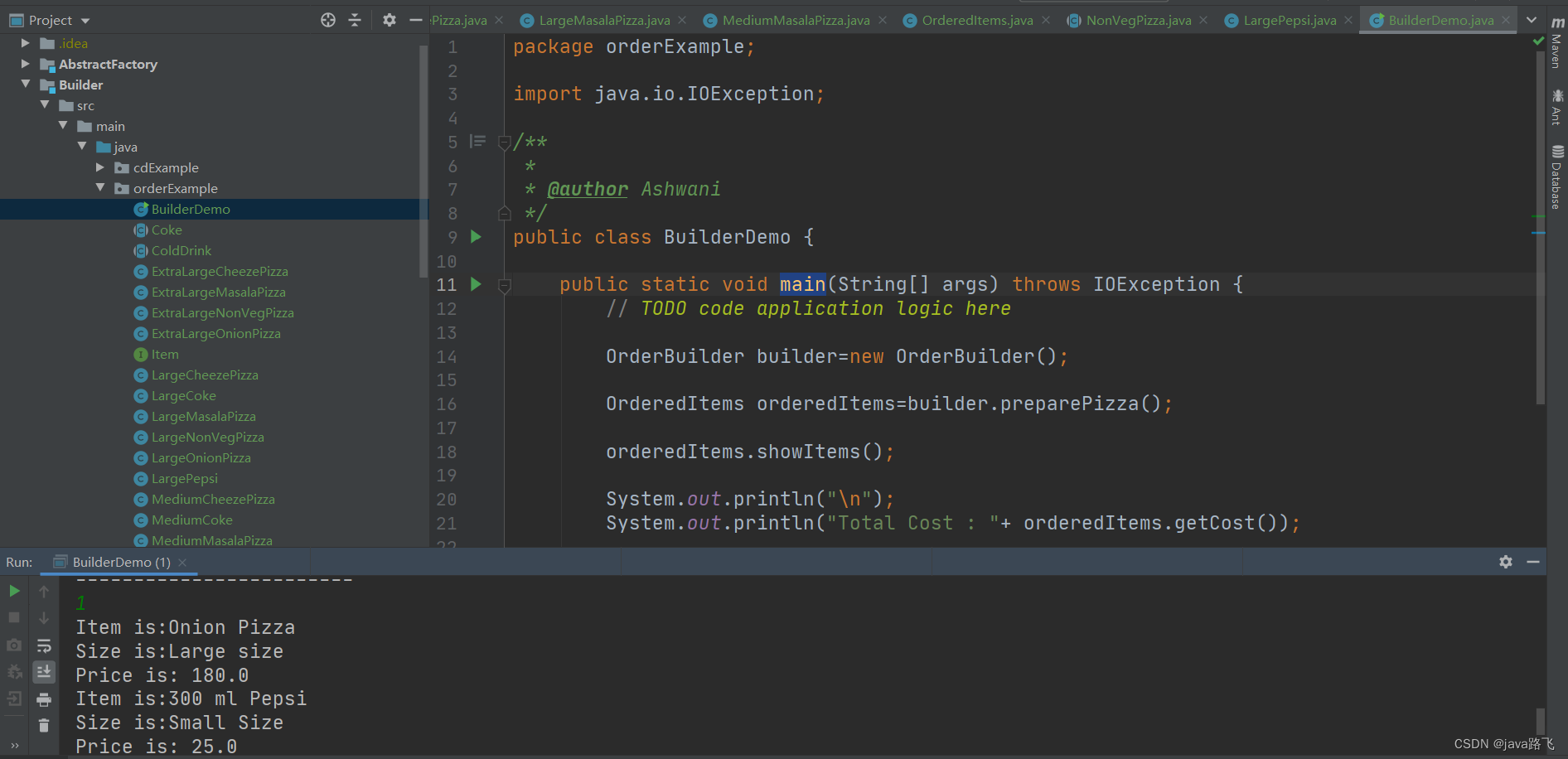
AIDD概述及药物综合数据库学习
- 人工智能辅助药物设计AIDD概述
- 安装环境
- anaconda
- vscode
- 虚拟环境
- 第三方库基本使用方法
- numpy (数据计算工具)
- pandas (数据清洗工具)
- matplotlib (结果可视化绘图工具)
- requests (数据库爬虫工具)
- 多种药物综合数据库的获取方式
- KEGG(requests爬虫)
- Chebi(libChEBIpy)
- PubChem(pubchempy / requests)
- ChEMBL(chembl_webresource_client)
- BiGG(curl)
- DeepChem集成MoleculeNet数据库介绍及下载
机器学习辅助药物设计
-
机器学习理论知识
- 机器学习种类:
- 监督学习
- 无监督学习
- 强化学习
- 典型机器学习方法
- 决策树
- 支持向量机
- 朴素贝叶斯
- 神经网络
- 卷积神经网络
- 模型的评估与验证:准确率、精确率、召回率、F1分数、ROC曲线、AUC计算,平均绝对误差、均方差、R2分数、可释方差分数,交叉验证等
- sklearn工具包基本使用
- 机器学习种类:
-
rdkit工具包的基本使用
-
RDKit安装
- 化合物编码方式和化合物相似性理论知识
- 基于RDKit的分子读写
- 基于RDKit的分子绘制
- 基于RDKit的分子指纹与分子描述符
- 基于RDKit的化合物相似性与子结构
- 基于 RDKit 与 Python3 的构象与 RMSD 计算
-
项目实战
- 基于 Pytorch 和 RDKit 建立 QSAR 模型
- 基于 scikit-learn 将 pytorch 用于 QSAR 模型构建
- 项目实战1:基于ADME和Ro5的分子筛选
- 项目实战2:基于化合物相似性的配体筛选
- 项目实战3:基于化合物相似性的分子聚类
- 项目实战4: 基于机器学习的生物活性预测
- 项目实战5:基于机器学习的分子毒性预测
图神经网络辅助药物设计
- 图神经网络基础知识
- 框架介绍: PyG,DGL,TorchDrug
- 图神经网络消息传递机制
- 图神经网络数据集设计
- 图神经网络节点预测、图预测任务和边预测任务实战
- 项目实战1:基于图神经网络的分子毒性预测
- SMILES分子数据集构建PyG图数据集
- 基于GNN进行分子毒性预测
- 项目实战2:基于图神经网络的蛋白质-配体相互作用预测
- 蛋白质分子图形化,构建PyG图数据集
- 基于GIN进行网络搭建及相互作用预测
自然语言处理辅助药物设计
- 自然语言处理概述
- 文本类语言的向量表示方法
- Encoder-Decoder模型
- 循环神经网络模型
- Seq2seq模型
- Attention注意力机制
- Transformer模型
- 项目实战
- 基于无监督的Seq2Seq模型进行分子表示学习
- 基于Transformer模型的反应表示方法
- 基于自然语言处理的反应分类任务
- 基于BERT模型的反应产量预测任务
药物设计与分子生成
- 分子生成模型概述
- 循环神经网络RNN
- 变分自动编码器VAE
- 生成对抗网络GAN
- 强化学习RL
- 基于RDKit提取反应规则预测分子生成
- 基于 RDKit 处理化学信息学中的反应方程式
- 基于 RDKit 绘制化学反应
- 基于 RDKit 和 SMARTS 的化学反应处理
- 基于RDKit的化学反应指纹与化学反应相似度计算
- 基于 RDKit 通过 SMARTS 定义反应模式来生成反应产物
- 基于深度学习的分子生成
- 基于图数据的小分子化合物生成模型
- 基于MolGAN的分子生成
- 分子合成可行性评估
计算机辅助药物设计【蛋白质专题】
蛋白质数据结构
- 数据库介绍与相关数据爬取
- PDB数据库
- UniProt数据库
- KLIFS数据库
- 【基于RDKit的蛋白质基本操作】
- 基于 RDKit 的氨基酸序列转换为 SMILES
- 基于 RDKit 的肽和核酸序列转换分子 Mol 对象
- 多肽 HELM 字符串格式与分子 Mol 格式间的转换
- 从 ChEMBL 数据库提取大分子 HELM 单体(XML 转换为 DataFrame 并搜索部分结构)
- 基于RDKit的药效团特征与可视化
- 【基于RDKit的药效团处理】
- RDKit 中的药效团特征
- RDKit:可视化药效团(Pharmacophore)
- RDKit | 基于 RDKit 从分子中提取 3D 药效团特征
- RDKit | 基于 RDKit 计算 3D 药效团指纹
- 【基于RDKit的骨架 (Scaffold)】
- RDKit | 基于 RDKit 操纵分子结构(骨架转换)
- RDKit:化合物骨架分析(基于 Python3)
- 【基于RDKit的片段 (Fragments)处理】
- RDKit 中的 RECAP 进行分子裂解
- RDKit:基于 RECAP 生成片段
- RDKit | 可视化重要片段
- RDKit | 基于片段的分子生成(骨架 A + 骨架 B)
- RDKit | 基于多片段的分子生成(骨架 A + 骨架 B + 骨架 C)
激酶-Kinase相似性学习
- 激酶基本理论介绍
- Kinase相似性:序列
- Kinase相似性:Kinase口袋(KiSSim指纹)
- Kinase相似性:交互指纹
- Kinase相似性:配体配置文件
- Kinase相似性:比较不同的view
基于序列的蛋白质属性预测
- 多重序列对比
- 基于蛋白质序列的深度学习和机器学习任务
- 预测突变对TEM-1β-内酰胺酶蛋白的影响
- 基于蛋白质的二级结构预测残基的属性
- 通过蛋白质的溶化温度预测蛋白质的稳定性
基于结构的蛋白质属性预测
- 蛋白质结构数据处理
- 基于RDKit的蛋白质动态图构建
- 基于几何感知关系图神经网络(GearNet)及其边缘消息传递的扩展(GearNet-Edge)的蛋白质结构表示模型
蛋白质-配体相互作用预测PLI
- 结合位点相似性和脱靶预测
- 结合位点预测
- 蛋白质-配体对接
- 蛋白质-配体相互作用
- NGLView高级教程
- 分子动力学模拟
- 分析分子动力学模拟
- 基于图神经网络的蛋白质-配体相互作用预测(分类任务)
- 基于机器学习的分子对接来预测蛋白质-配体的结合亲和力(回归任务)






![全文检索[ES系列] - 第495篇](https://img-blog.csdnimg.cn/img_convert/880d863c11479bc7084d22b7b0b1b11f.png)