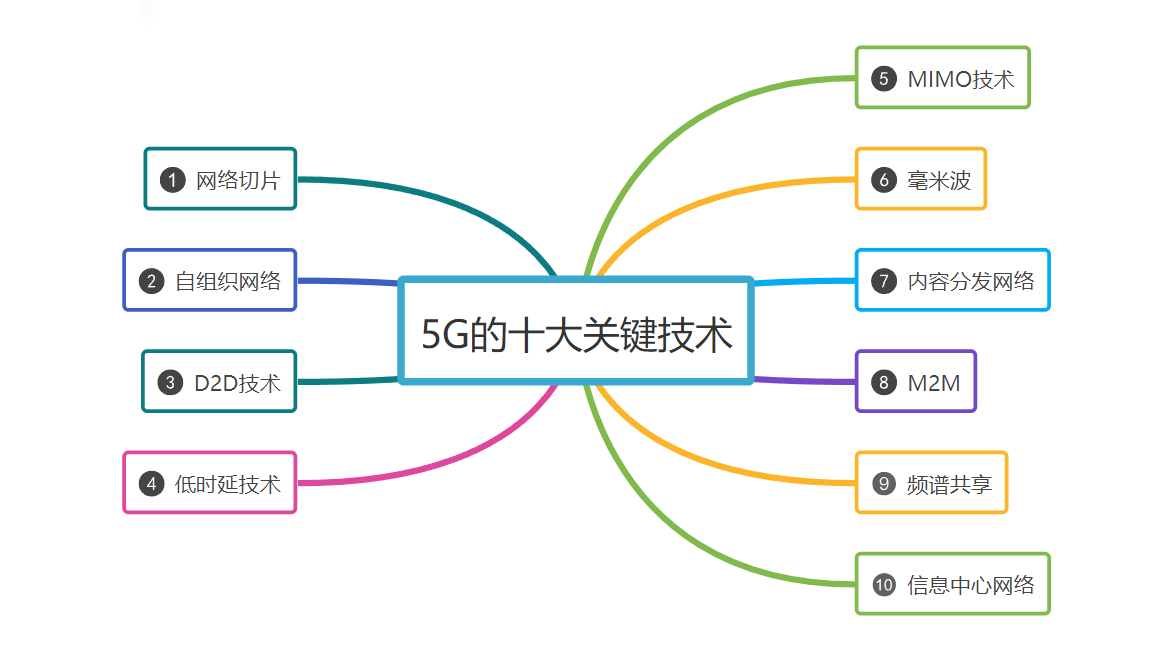
微生物序列数据库包含大量有关酶和其他可用于生物技术的分子的信息。但近年来,这些数据库已经变得非常庞大,以至于很难有效地搜索到感兴趣的酶。
2023年11月23日,博德研究所张锋及美国国立卫生研究院Eugene V. Koonin共同通讯在Science 在线发表题为“Uncovering the functional diversity of rare CRISPR-Cas systems with deep terascale clustering”的研究论文,该研究开发了基于位置敏感哈希的快速聚类(FLSHclust)算法,该算法在线性时间内对大量数据集进行深度聚类。
该研究将FLSHclust纳入CRISPR发现管道,并鉴定了188个以前未报道的CRISPR相关基因模块,揭示了许多与适应性免疫相关的其他生化功能。该研究通过实验表征了三种含HNH核酸酶的CRISPR系统,包括第一种具有特定干扰机制的IV型系统,并对它们进行了基因组编辑。该研究还鉴定并表征了一种候选的VII型系统,显示了它对RNA的作用。这项工作为利用CRISPR和更广泛地探索微生物蛋白质的巨大功能多样性开辟了新的途径。

该算法来自CRISPR先驱张锋实验室。他们使用大数据聚类方法来快速搜索海量基因组数据。这些作者利用他们的算法---基于快速位置敏感散列的聚类(Fast Locality-Sensitive Hashing-based clustering, FLSHclust)---分析了三个主要的公共数据库,这些数据库包含来自一系列不同寻常细菌的数据,包括在煤矿、酿酒厂、南极湖泊和狗唾液中发现的细菌。
作者发现,CRISPR系统的数量和多样性都令人惊讶,其中包括可以对人体细胞中的DNA进行编辑的CRISPR系统、可以靶向RNA的CRISPR系统,以及许多具有其他多种功能的CRISPR系统。
与目前的CRISPR/Cas9系统相比,这些新的CRISPR系统有可能被用来编辑哺乳动物细胞,其脱靶效应更少。有朝一日,它们还可以用作诊断工具,或作为细胞内活动的分子记录。
这些作者说,他们的研究凸显了CRISPR系统前所未有的多样性和灵活性,随着数据库的不断扩大,可能还有更多稀有的CRISPR系统有待发现。
张锋说,“生物多样性是一座宝库,随着我们继续对更多的基因组和宏基因组样本进行测序,我们越来越需要更好的工具,比如FLSHclust,来搜索序列空间,以便寻找分子宝石。”
什么是CRISPR?
CRISPR全名为“成簇的、规律间隔的短回文重复序列”,是细菌防御病毒侵入的一种机制。2012年法国科学家埃玛纽埃勒·沙尔庞捷和美国科学家珍妮弗·道德纳发表研究指出,她们开发出CRISPR/Cas9基因编辑技术。这项技术随后成为生物医学史上第一种可高效、精确、程序化修改细胞基因组包括人类基因组的工具。这种技术就是以核糖核酸(RNA)做向导,把Cas9酶带到相应的位置,然后用这种酶切割病毒DNA。
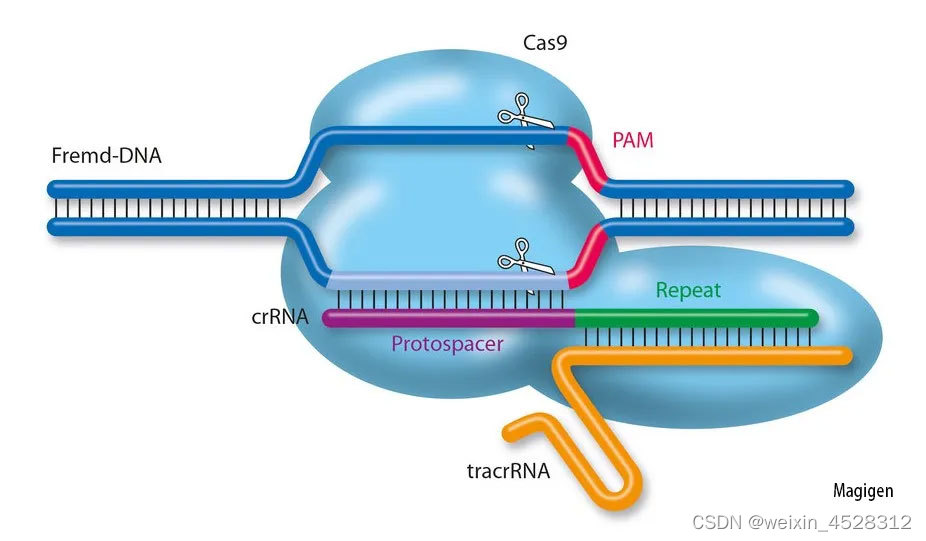
图一:CRISPR基因编辑技术示意图
相比此前的技术,CRISPR/Cas9技术具有成本低、易上手、效率高等优势,使得对基因的修剪改造“普通化”,因此风靡整个生物学界。科学界普遍认为,这是21世纪以来生物技术方面最重要的突破。这一技术曾三度入围美国《科学》杂志年度十大突破,并且在2015年被该杂志评为年度头号突破。
就像在科学领域时常发生的“偶然”那样,“基因剪刀”的发现过程也出乎意料。沙尔庞捷在研究化脓性链球菌时,发现了一种未知分子——tracrRNA。她的研究显示,tracrRNA是细菌的古老免疫系统“CRISPR/Cas”的一部分,能够通过切割病毒的DNA来使病毒“缴械”,从而消除其危害。
沙尔庞捷2011年发表了上述研究成果。同年,她与道德纳开始合作研究。在一次具有划时代意义的实验中,她们对“基因剪刀”进行改造。在天然形式下,这种“剪刀”能够识别出病毒中的DNA。但是沙尔庞捷和道德纳发现能对“剪刀”施加控制,这样一来就能在任何预先设定的位置切割任何DNA分子。一旦DNA被切割,那么重写生命的密码就变得简单了。
此后,“基因剪刀”技术的利用次数呈爆炸性增长。在基础科研领域,随着这一技术的应用,涌现出很多重大成果。例如植物研究者开发出能够耐霉菌、害虫和干旱的作物;在医学领域,与该技术相关的癌症新疗法临床试验正在开展,治愈遗传性疾病有望成为现实。
开发新算法寻找CRISPR
在张锋等人的这项研究中,为了从蛋白和核酸序列数据库中挖掘新型CRISPR系统,他们借鉴大数据领域的一种方法,开发了一种算法。它将相似但不完全相同的对象聚类在一起。
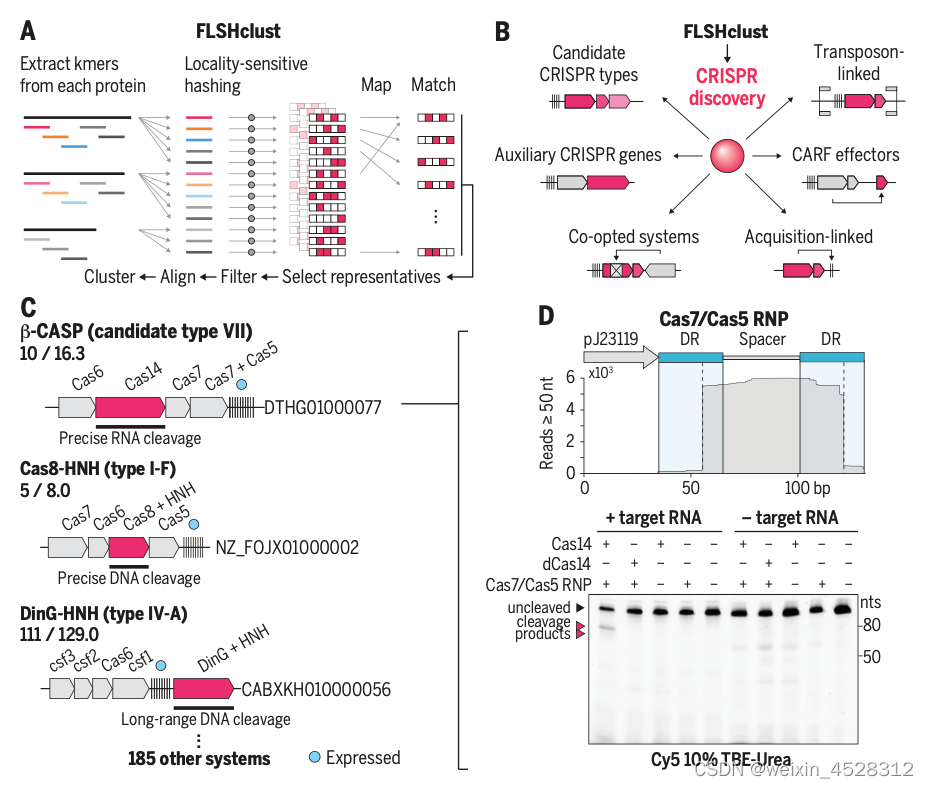
图二:FLSHclust探寻新的CRISPR-Cas系统的示意图
通过使用这种方法,作者可以在几周内探查数十亿个蛋白和DNA序列---这些序列来自NCBI、全基因组鸟枪数据库(Whole Genome Shotgun database)和联合基因组研究所(Joint Genome Institute),而以前寻找相同对象的方法需要几个月的时间。他们设计的算法旨在寻找与CRISPR相关的基因。
论文共同第一作者Soumya Kannan说,“这种新算法允许我们在足够短的时间内解析数据,从而可以实际恢复结果并提出生物学假设。”另一名论文共同第一作者为来自张锋实验室的Han Altae-Tran。
Altae-Tran说,“这证明了当你改进探索方法并使用尽可能多的数据时,你能做什么。能够提高我们搜索的规模,这真地很令人兴奋。”
新的CRISPR系统
在分析过程中,Altae-Tran、Kannan 和他们的同事们注意到,他们发现的数千个 CRISPR 系统分为几个现有类别和许多新类别。他们在实验室中对其中的几个新的CRISPR系统进行了更详细的研究。
他们发现了已知 I 型 CRISPR 系统的几种新变体,它们使用 32bp长的向导RNA(gRNA),而CRISPR/Cas9系统使用20 bp长的gRNA。由于具有较长的gRNA,这些 I 型CRISPR系统有可能被用于开发更精确而不易发生脱靶编辑的基因编辑技术。
张锋实验室发现其中的两种CRISPR系统可以对人类细胞的DNA进行较短的序列编辑。由于这些I型CRISPR系统的大小与CRISPR/Cas9系统相似,因此它们很可能可以用目前用于CRISPR的基因递送技术递送到动物或人类的细胞中。
其中的一种 I 型CRISPR系统还显示出“附带活性”---CRISPR 蛋白结合靶序列后,核酸会被广泛降解。科学家们已经用类似的系统制造出了传染病诊断仪,如SHERLOCK,这是一种能够快速检测单个DNA或RNA分子的工具。Zhang实验室认为,这些新的CRISPR系统也可能用于诊断技术。
这些作者还发现了一些IV型CRISPR系统的新作用机制,以及一种精确靶向RNA的VII型CRISPR系统,因此该VII型CRISPR系统有可能用于RNA编辑。其他的CRISPR系统有可能被用作记录工具---基因表达的分子记录,或用作活细胞中特定活动的传感器。
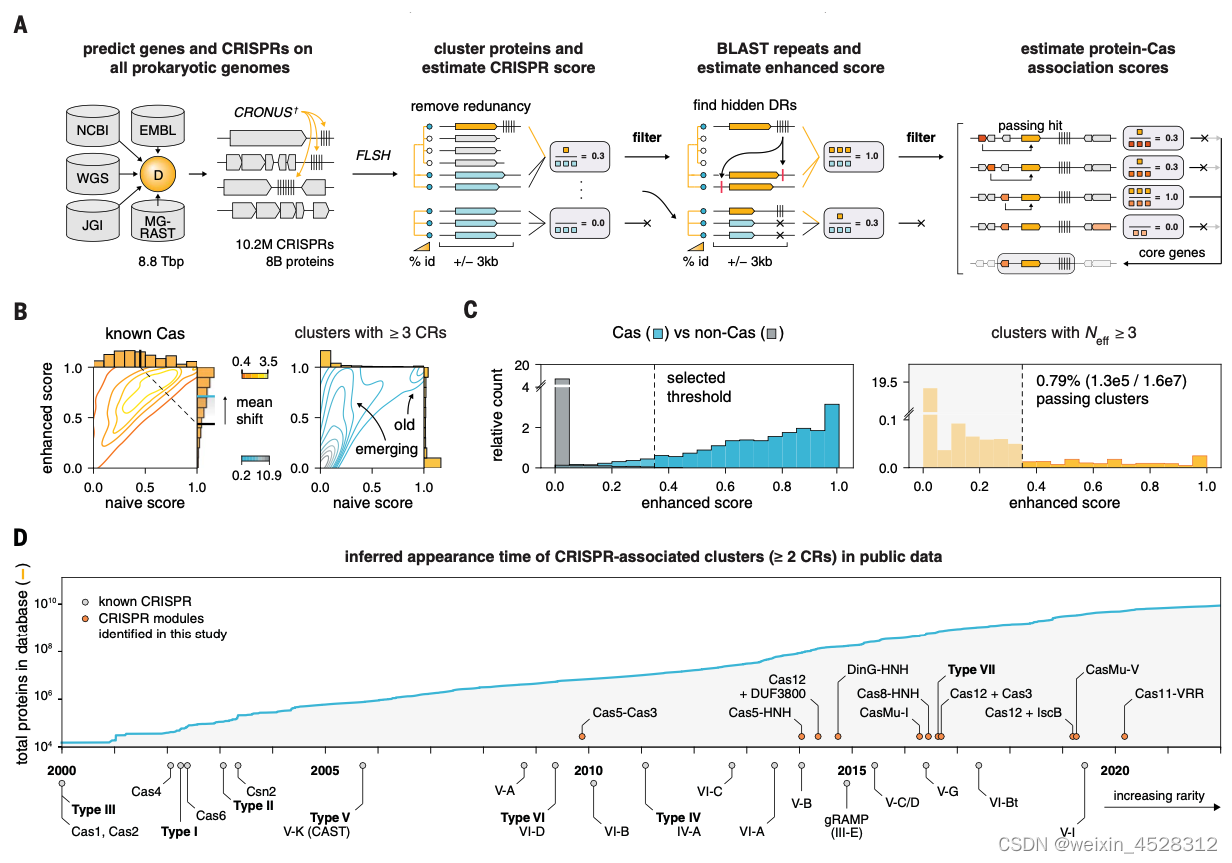
图三:新的聚类算法发现数百个罕见的以前未被发现的CRISPR系统,具有敏感的,可扩展的CRISPR关联管道。
利用新算法进行数据挖掘
这些作者说,他们的算法可能有助于寻找其他生化系统。Altae-Tran 说,“任何想利用这些大型数据库研究蛋白如何进化或发现新基因的人都可以使用这种搜索算法。”
他们补充说,他们的发现不仅说明了CRISPR系统的多样性,还说明了大多数CRISPR系统是罕见的,只存在于不常见的细菌中。
Kannan说,“其中的一些微生物CRISPR系统只存在于煤矿的水中。如果不是有人对此感兴趣,我们可能永远都不会看到这些CRISPR系统。扩大我们的取样多样性对于继续扩大我们所能发现的CRISPR系统多样性确实非常重要。”

图四:本研究鉴定的CRISPR新系统之一(CRISPR-Cas效应模块)。
研究展望
目前,CRISPR/Cas9技术正在不断革新基因组编辑领域。它能够实现高度灵活性和特异性靶向性,可进行修饰和重定向,成为了干细胞工程、基因治疗、组织和动物疾病模型以及设计抗病转基因植物等广泛应用中的强大基因组编辑工具。

麻省理工学院教授、博德研究所研究员张锋

通讯作者之一,NIH生信专家Eugene V. Koonin博士
CRISPR/Cas9被归类为CRISPR系统中的II型。实际上,这只是其中的一种类型——目前研究人员已经确定了六种类型的CRISPR系统,命名为I-VI型,它们有不同的特性,包括它们使用的酶的类型以及如何识别、结合和切割RNA或DNA。
而此次新发现的CRISPR类型的特征可以用于其他应用,并有可能改编成最新的基因组编辑工具。
新西兰达尼丁奥塔哥大学生物化学家克里斯·布朗(Chris Brown)认为,这种算法本身是一个重大进展,研究人员可以用它来寻找不同物种中的其他类型的蛋白质,他表示“我非常敬佩他们能做到这一点”。
“这是生物化学家的宝库。”对于新发现的VII型CRISPR系统,德国马尔堡大学的微生物学家伦纳特·兰道(Lennart Randau)提到,“下一步将是研究这些酶和系统是如何工作的,以及它们如何被改造用于生物工程。某些CRISPR蛋白质会随机切割DNA,对生物工程毫无用处,但它们在检测DNA或RNA序列方面非常精确,可能会成为很好的诊断或研究工具。”
可以说,CRISPR新类型的发现,对基因工程来说意义重大。
小结
在最新一期《Science》杂志上,MIT和Broad研究所的张锋教授团队与美国国立卫生研究院的Eugene Koonin教授合作,借助其开发的全新算法,从数十亿个蛋白质序列中发现了188个此前未知的新型CRISPR相关系统,并且可能将CRISPR系统的类型拓展至7大类。这一发现进一步证实CRISPR系统的多样性,并且有望带来新型基因编辑工具。
FLSHclust是一种基于序列相似性,利用大数据对蛋白质进行聚类的算法。FLSHclust可以对公开数据库中的基因序列进行分析,这些数据包含了收集自南极湖泊、狗的唾液、啤酒厂等广泛来源的细菌与古菌。最终的数据库包括80亿个蛋白和1020万个CRISPR阵列。通过寻找相似但不完全相同的序列,研究将其分为约5亿个簇,从宏基因组数据库中寻找CRISPR相关基因。
参考资料:
Han Altae-Tran et al. Uncovering the functional diversity of rare CRISPR-Cas systems with deep terascale clustering. Science, 2023, doi:10.1126/science.adi1910.