Polygenic Risk Scores in R
最朴素的理解PRS:
GWAS分析结果中,有每个SNP的beta值、se值、P值,因为GWAS分析中将SNP变为0-1-2编码,所以这些显著的SNP的beta值,就可以用于预测。
比如:GWAS分析中,显著的SNP效应值为:
SNP1: 0.3
SNP2: 0.2
SNP3: -0.1
对于target data(目标群体),检测了3个个体,3个SNP的分型分别为:
ID1 0 0 1
ID2 1 0 2
ID3 2 2 1
那么个体1的多基因评分为:00.3 + 00.2 + 1*-0.1 = -0.1
个体2的多基因评分为:0.3 + 0 + -0.1 = 0.2
个体3的多基因评分为:0.6 + 0.4 + -0.1 = 0.9
用数学公式表示:
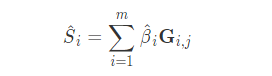
- beta是效应值
- G是0-1-2的编码
- m是m个SNP
实际项目的PRS计算

实际中的项目,考虑的因素比较多,比如:
- 数据质控
- 群体结构
- LD值(clumping)
- beta矫正值
- 通过P值筛选最优组合
相关软件实现PRS分析
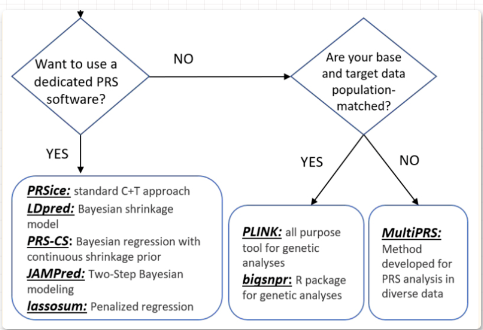
- plink
- biqsnpr,一个R包
- PRSice,应用最广泛,通过C+T的策略
- LDpred,通过贝叶斯收缩的模型
- PRS-CS
- JAMPred
- Lassosum
之前写过PRS的操作流程,可以作为参考:
多基因风险预测模型1–先立Flag
多基因风险预测模型2–相关概念和软件
不会安装使用PRSice-2软件就太不讲究了