简介
数字病理切片,也称为全幻灯片成像(Whole Slide Imaging,WSI)或数字切片扫描,是将传统的玻片病理切片通过高分辨率扫描仪转换为数字图像的技术。这种技术对病理学领域具有革命性的意义,因为它允许病理切片以电子形式保存和查看,从而便于数据共享、远程诊断和自动化分析。
数字病理切片的主要特点包括:
-
高分辨率扫描: 数字扫描仪能够以极高的分辨率扫描病理玻片,这意味着可以在电脑屏幕上查看到与显微镜下相同的详细信息,甚至更好。这些扫描图像可以放大和缩小,查看不同的细节层次。
-
便于存储和共享: 数字切片可以存储在服务器上,通过网络被远程访问和共享。这对于多地点的医疗机构、教育和研究机构尤其有用,因为它使得远程会诊、教育培训和科研合作变得更加简单和高效。
-
图像分析软件: 随着人工智能和机器学习技术的发展,数字切片可以被用于自动病变检测和量化分析。这些软件能够识别特定的组织结构、计算组织参数等,有助于标准化病理诊断并减少人为误差。
-
教育和培训: 数字切片可以用于病理学教育,学生可以在没有实体显微镜的情况下访问高质量的切片图像。此外,可以创建包含大量病例的数据库,供学生学习和测试。
-
持久性和可靠性: 与传统的玻璃切片相比,数字切片不会随时间退化,更易于长期保存。
应用领域
- 临床诊断: 提供远程病理诊断服务,特别是在资源有限的地区。
- 研究: 便于进行大规模的图像分析和数据挖掘,推动病理学的研究。
- 教育: 作为教学资源,提高病理学教育的可访问性和质量。
数字病理切片正在逐渐成为现代病理学的标准工具之一,其潜在的优势正在被越来越多的医疗机构和研究所认可和采用。
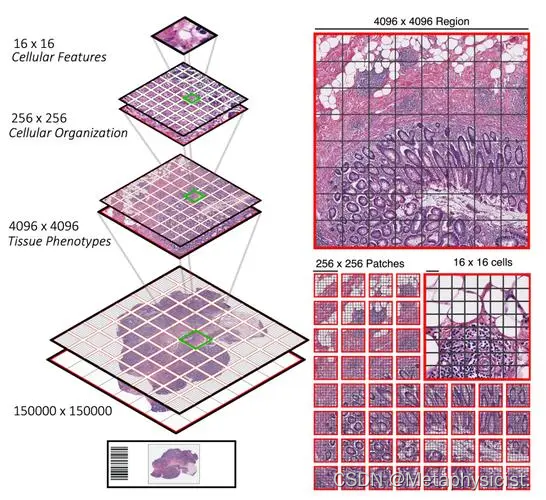
数字病理切片通常按照病理金字塔的方式采集和存储,以便在不同的放大倍数下观察切片的细节。在进行后续的图像处理和分析时,我们需要将这些切片数据转换为标准的图像格式,如 TIFF 或 SVG,以便与主流的图像处理工具兼容。

本教程将介绍如何使用 Python 和相关的开源工具,将WSI)数据从 .mds 格式转换为 TIFF 格式,并查看 WSI 的不同图层。
环境准备
- 安装 pma_python 库,用于与 Pathomation 数字显微镜软件进行交互。可以通过以下命令安装:
pip install pma_python
Pathomation是一个
2. 确保已经在系统上正确安装和配置了 openslide 库。这个库对于处理 WSI 数据至关重要。
转换 WSI 数据格式
对于以 .mds 格式保存的 WSI 数据,我们可以使用ConvertToTtiff.py 脚本将其转换为 TIFF 格式。这个转换对于与大多数图像处理工具的兼容性非常重要。
-
从 pma_python GitHub 仓库 下载
ConvertToTtiff.py脚本。 -
运行脚本,指定输入的 .mds 文件和输出的 TIFF 文件路径:
分析 WSI 图层
为了查看 WSI 数据的不同图层,我们可以参考https://www.cnblogs.com/ybqjymy/p/17384906.html中的教程,对应修改文件地址,即可打印出WSI图层信息。
注意事项
-
在运行上述脚本之前,请确保已经正确安装了所有的依赖项和库,以确保工作流程的顺利和高效。
-
转换和处理大型 WSI 数据可能需要较长时间,请耐心等待。
-
如果在使用过程中遇到任何问题,请参考 openslide 和 pma_python 的官方文档寻求帮助。
通过本教程,你应该能够将 .mds 格式的 WSI 数据转换为 TIFF 格式,并使用 Python 脚本分析 WSI 的不同图层。这为后续的图像处理和分析工作奠定了基础。
Reference
[1] https://www.pathomation.com/
[2] https://github.com/Pathomation/pma_python
[3] https://openslide.org/api/python/
[4] https://openslide.org/