MolDiff模型是一种考虑分子键生成的3D分子生成的新模型。MolDiff是清华大学智能产业研究院马剑竹课题组发表在PMLR 2023的工作,第一作者是Xingang Peng,文章题目为:《 Addressing the Atom-Bond Inconsistency Problem in 3D Molecule Generation 》。MolDiff的文章链接:https://arxiv.org/abs/2305.07508。马剑竹课题组在药物设计,特别是分子生成领域发表过不少文章,我们之前介绍过3DLinker就是他们课题组的工作。同时,Xingang Peng也是pocket2mol的作者。

在3D分子生成中,扩散模型先生成原子,保存到xyz格式中,然后通过obabel后处理生成完整的3D分子保存在sdf等文件中。在3D分子生成的过程中,生成的原子可能没有相应的化学键,因为它们的位置是在不考虑潜在化学键的情况下生成的。而这会导致原子和化学键不一致的问题,造成生成的分子“不真实”。
作者提出了一种新的3D分子生成扩散模型 MolDIff。通过对原子-原子键之间关系显式建模的方式,MolDiff可以同时生成原子和原子键。对比实验结果说明,MOlDiff在生成分子的成功率上提升了三倍,同时生成分子的质量更高。
一、MolDiff模型基本介绍
当前3D分子生成方法生成真实分子的效率并不高。因为,在3D分子生成过程中,采用两步法,先使用模型预测原子类型以及相应的原子坐标,然后根据距离判断原子之间的原子键。然而,两步法会生成一些不真实的拓扑结构(例如:大环)。此外,根据距离判断原子键会导致原子价键错误(例如:不完整的苯环)。

为此,作者提出了对原子和分子键同时进行概率采样的扩散模型MolDiff。MolDiff基于SE3等变神经网络,同时进行原子和化学键的消息传递。MolDiff采用不同层级的噪音去扰动原子以及化学键,并在分子生成过程中,使用一个原子键预测器的梯度引导生成的原子更适合生成正确的分子键,以提高生成真实分子的概率。
MolDiff模型中,一个分子被表示为原子类型A,原子坐标R,化学键B。MolDiff使用常见的7种元素(C, N, O, F, P, S, 和 Cl)和5种常见的化学键(单键、双键、三键和芳香键,以及dummy类型(none-type))。
MolDiff根据预先定义的噪声表逐渐向数据添加噪声,然后在逆向过程利用神经网络去除噪声,最终从噪声中恢复真实数据。经过所有的向前过程,原子位置近似遵循标准高斯分布,原子和键类型将所有概率质量放在吸收类型(dummy类型)上。注意,MolDiff将原子类型分散到吸收类型上,使用的稀疏的建模方式,而不是其他扩散模型的标准化高斯分布。
在反向过程中,MolDiff反转马尔可夫链以从先验分布重建真实样本,并使用 E(3) 等变神经网络来参数化转换过程。MolDiff利用高斯分布来预测原子位置,利用类型分布来预测原子类型和化学键类型。
在SE3等变神经网络部分,MolDiff设计一种新的等变网络架构和涉及原子和键的消息传递算法。MolDiff构造一个完整的图,其坐标为原子,所有顶点都相连。与ENGG等变神经网络不同,MolDiff更看重等变的作用。这些边还与其他边和节点传播消息以更新边表示。 该SE3等变模型是专门为MolDiff这种扩散框架设计的,其中节点和边的特征用于预测原子和键类型。
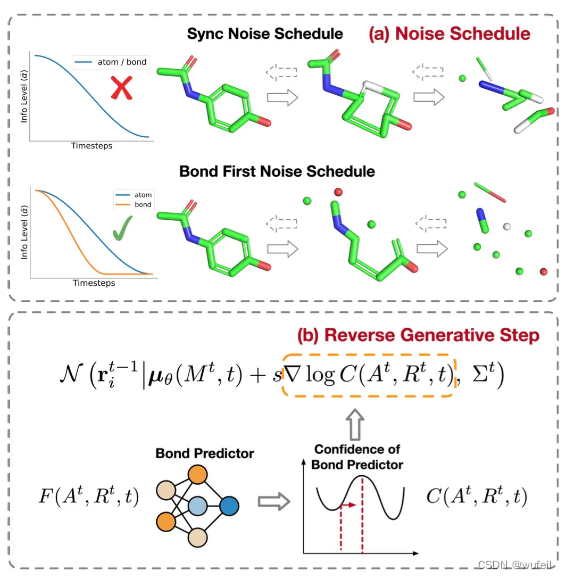
在向前过程中,与传统的扩散网络不同,MolDiff设计了新的噪音调度器(noise schedule),将原子类型和化学键的扩散过程分为两个阶段。在第一阶段,MolDiff使化学类型扩散到先验分布,所有化学键逐渐变成吸收性的dummy类型。 在此阶段,原子类型和位置仅受到轻微干扰。这样,模型变得更加鲁棒,并且可以获得当两个原子的距离在一定范围内而不是固定值时需要添加化学键的知识。在第二阶段,MolDiff不断扰动原子类型和位置以获得其相应的先验分布。总的来说,这种策略使键和原子之间的关系在扩散过程中保持更接近真实的关系。
MolDIff在生成过程中,利用键类型和键长度之间的紧密关系来指导原子位置的生成,以便模型可以将原子放置在正确的位置,从而生成准确的分子 3D 结构。为此,MolDiff设计了一个新的SE3等变网络,以原子类型和原子坐标为输入,预测化学键。利用在不同去噪t时刻,该神经网络预测的化学键的对原子坐标和原子类型的梯度,引导原子坐标和原子类型,以让生成的化学键置信度更高,生成的化学键的长度更加合理。
关于新的噪音调度器和化学键梯度引导,示意图如下图:
二、MolDiff性能介绍
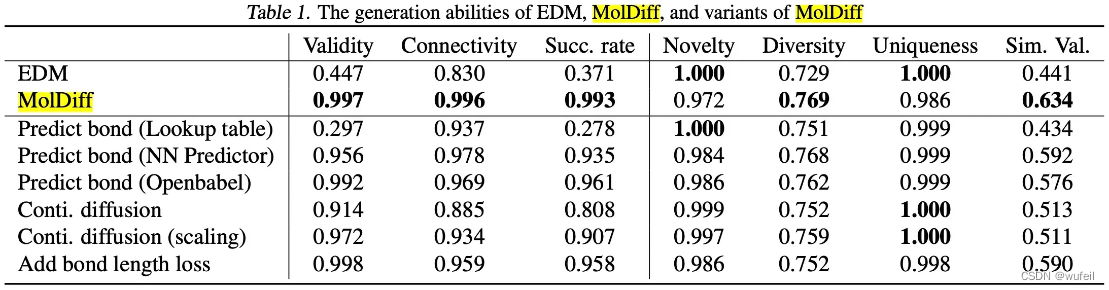
作者以EDM模型(https://arxiv.org/abs/2203.17003)作为对比。在有效率,链接率,成功率方面,MolDiff均明显优于EDM模型。需要注意的是,MolDiff文章中显示EDM生成分子的有效率是44.7%,但是在EDM模型的文章中,有效率是97.5%,这可能是因为有效率的计算方式不同(有待查证)。此外,MolDiff生成的分子在与原分子的相似率上也优于EDM模型。似乎添加见的损失也不错。。。毕竟都接近100%,一点点提升也没啥意思。
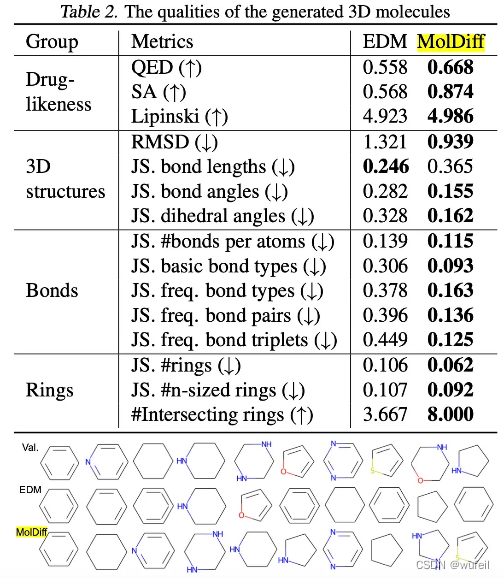
在生成分子的化学性质和3D构象方面,MolDiff在生成分子的类药性,与原参考分子的RMSD,键长,键角分布,二面角,化学键类型,以及环的分布上全面占优,如下表。这一点是非常好的结果,因为3D分子生成模型生成分子不类药,很大的原因就是这个。
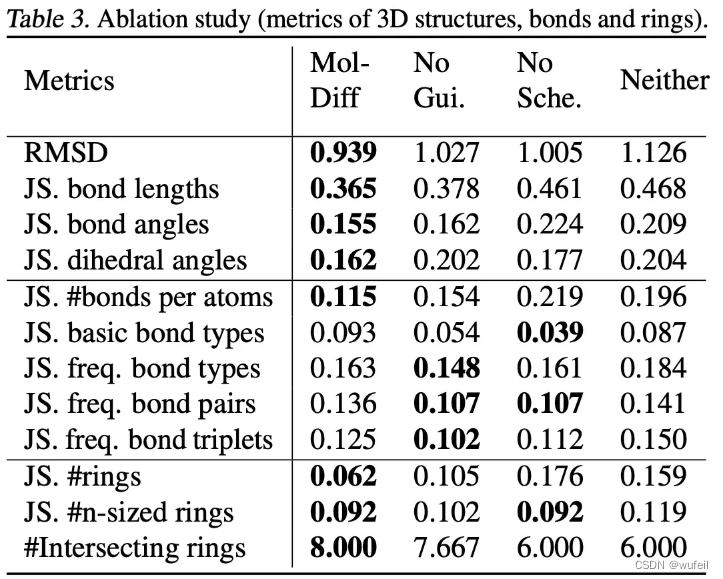
新的噪音调度器和化学键引导生成,是MolDiff模型的两个大创新。作者做了消融实验,对比完整的MolDiff,与没有梯度引导,没有噪音调度器,没有梯度引导也没有噪音调度器的结果。发现在RMSD相关的键长、键角、二面角指标,以及环相关的指标上上,MolDiff全面占优,体现了新的噪音调度器和化学键引导生成优势。
下图作者计算了计算去扩散过程中不同步骤长度时超出范围的真实键的比例。可以看出MolDiff明显比没有噪音调度器的结果更好,噪音调度器让模型更关注真实的数据分布。

三、MolDiff 评测
3.1 复制代码&安装环境
复制项目代码
git clone https://github.com/pengxingang/MolDiff.git复制环境并激活
conda env create -f env.yaml
conda activate MolDiff作者提供的环境代码非常齐全,安装非常顺利。但是,如果你安装的cuda版本太高,可能会出现安装的pytorch 1.10不能运行的问题。这时需要尝试新的pytorch版本,以兼容cuda。然后重装pyg等。
因为我的cuda版本是12.0,因此,我选择卸载pytorch等,然后重装。
卸载pytorch
conda uninstall pytorch重新安装pytorch,这里安装的是2.1.2,cuda_11.8版本。本机cuda的版本是12.0,但是向下兼容11.8。
conda install pytorch==2.1.2 torchvision==0.16.2 torchaudio==2.1.2 pytorch-cuda=11.8 -c pytorch -c nvidia
检查torch和cuda版本:
conda list | grep torch输出:
ffmpeg 4.3 hf484d3e_0 pytorch
libjpeg-turbo 2.0.0 h9bf148f_0 pytorch
pytorch 2.1.2 py3.8_cuda11.8_cudnn8.7.0_0 pytorch
pytorch-cuda 11.8 h7e8668a_5 pytorch
pytorch-mutex 1.0 cuda pytorch
torchaudio 2.1.2 py38_cu118 pytorch
torchtriton 2.1.0 py38 pytorch
torchvision 0.16.2 py38_cu118 pytorch安装pytorch 2.1.2以及cuda 11.8安装torch-scatter, torch-sparse, torch-cluster,torch-geometric(安装过程参考:https://blog.csdn.net/qq_54478153/article/details/122683064)。whl安装包链接:https://pytorch-geometric.com/whl/torch-2.1.2%2Bcu118.html
pip install torch-scatter torch-sparse torch-cluster torch-spline-conv -f https://pytorch-geometric.com/whl/torch-2.1.2%2Bcu118.html
pip install torch-geometric安装过程非常顺利,代码和checkpoint均无需修改,即可直接使用。感谢作者提供了如此完整以及高鲁棒性的代码,兼容不同的pytorch的版本。完整的项目代码以及说明文档,一直是马剑竹课题组的工作的特点。手动大写👍!!国内这样的严谨的研究人员很少!!!
下载作者预训练好的checkpoint,下载链接为:https://drive.google.com/drive/folders/1zTrjVehEGTP7sN3DB5jaaUuMJ6Ah0-ps
作者提供了三个checkpoint,分别是:MolDiff.pt,MolDiff_simple.pt, bond_predictor.pt 分别是,预训练的完整 MoDiff 模型, 未使用键噪音进行训练的预训练简化 MolDiff 模型,以及预训练的键预测器,用于采样过程中的键指导。
将下载好的checkpoint放置在./ckpt文件夹内。
3.2 分子生成测试
使用GitHub中案例,尝试生成分子:
python scripts/sample_drug3d.py \--outdir ./outputs \--config ./configs/sample/sample_MolDiff.yml \--device cuda:0 \--batch_size 32注意:要修改作者中的batch_size 以及 cuda的编号。毕竟我们是小机器。output指定分子生成的保存路径;config指定分子生成的配置文件;device指定GPU设备及其编号;batch_size设定批次大小,如果未设置将按照config文件中的配置进行生成。
./configs/sample/sample_MolDiff.yml 配置文件内容如下:
model:checkpoint: ckpt/MolDiff.ptbond_predictor: ckpt/bondpred.ptsample:seed: 2023batch_size: 512num_mols: 1000save_traj_prob: 0.02guidance:- uncertainty- 1.e-4其中,guidance配置了使用键预测模型的梯度对分子生成过程进行引导,引导强度为1.e-4。分子生成过程使用的是MolDiff的完整版。
以下是一些输出示例:
[2024-04-06 09:41:00,504::sample::INFO] Namespace(batch_size=32, config='./configs/sample/sample_MolDiff.yml', device='cuda:0', outdir='./outputs')
[2024-04-06 09:41:00,504::sample::INFO] {'model': {'checkpoint': 'ckpt/MolDiff.pt'}, 'bond_predictor': 'ckpt/bondpred.pt', 'sample': {'seed': 2023, 'batch_size': 512, 'num_mols': 1000, 'save_traj_prob': 0.02, 'guidance': ['uncertainty', 0.0001]}}
[2024-04-06 09:41:00,504::sample::INFO] Loading data placeholder...
。。。 。。。
2<00:00, 10.87it/s]
[09:42:32] Can't kekulize mol. Unkekulized atoms: 0 4 5 10 11 14 17 19 20
[09:42:32] Can't kekulize mol. Unkekulized atoms: 0 4 5 10 11 14 17 19 20
[2024-04-06 09:42:32,977::sample::INFO] Success: O=C1CC(c2ccc(O)cc2)NC2CCC(c3ccccc3)CC12
[2024-04-06 09:42:32,978::sample::INFO] Success: COc1ccc(CNC(=O)c2ccc(C(=O)NCc3ccc(OC)cc3)nc2)cc1
[2024-04-06 09:42:32,978::sample::INFO] Success: O=C(NCCN1CCC(c2ccccc2)(c2ccccc2)C1)c1ccccc1
。。。 。。。
[09:42:33] non-ring atom 2 marked aromatic
[09:42:33] non-ring atom 0 marked aromatic
[09:42:33] non-ring atom 2 marked aromatic
[09:42:33] non-ring atom 0 marked aromatic
[09:42:33] non-ring atom 3 marked aromatic运行结束后,会生成两个含有运行日期的文件夹,例如:
.
├── sample_MolDiff_20240420_021957
└── sample_MolDiff_20240420_021957_SDF在SDF文件夹可以查看生成分子的3D结构,同时包含了几个文件名带traj的sdf文件,保存了相应分子的去噪生成过程。生成分子的3D结构如下:
MolDiff_all
其中,29号分子的轨迹如下(未完整):
MolDiff_traj
部分生成分子的2D结构如下图:
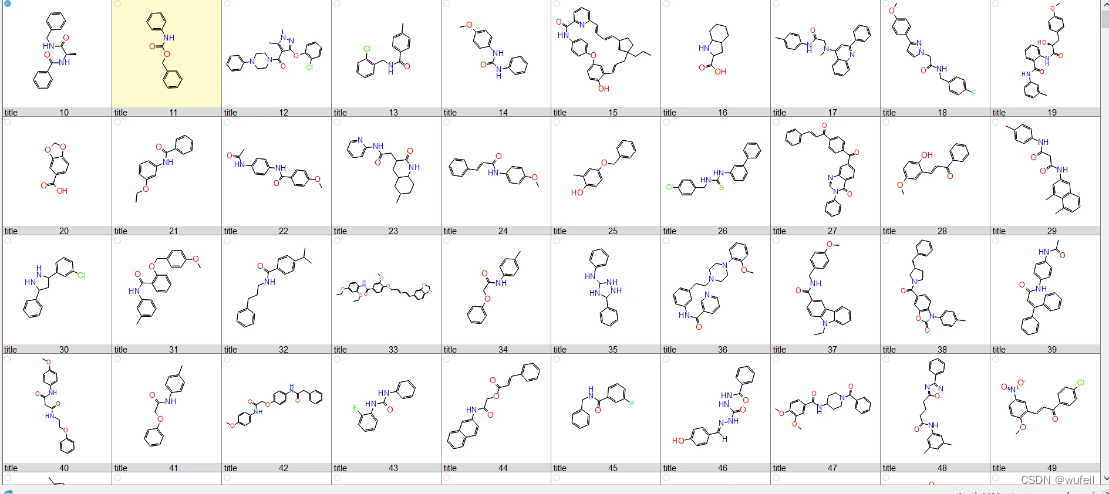
3.3 生成分子测试
评估生成的分子,运行如下代码:
python scripts/evaluate_all.py \--result_root ./outputs \--exp_name sample_MolDiff_20240420_021957 \--from_where generated注:在一步需要一个数据集的mol_summary.csv文件。下面四的内容,下载作者与处理好的数据,里面包含有这个文件。放置在./data/geom_drug文件夹中即可。
运行结束以后,结果保存在:./outputs/sample_MolDiff_20240420_021957/mols.csv,其中记录了每一个生成分子的各种性质,包括:qed,sa,logp,lipinski,n_atoms,n_bonds,n_rings,n_rotatable,weight,n_hacc,n_hdon,rmsd_max,rmsd_min,rmsd_median,cnt_eleC,cnt_eleN,cnt_eleO,cnt_eleF,cnt_eleP,cnt_eleS,cnt_eleCl,cnt_bond1,cnt_bond2,cnt_bond3,cnt_bond4,cnt_ring3,cnt_ring4,cnt_ring5,cnt_ring6,cnt_ring7,cnt_ring8,cnt_ring9。
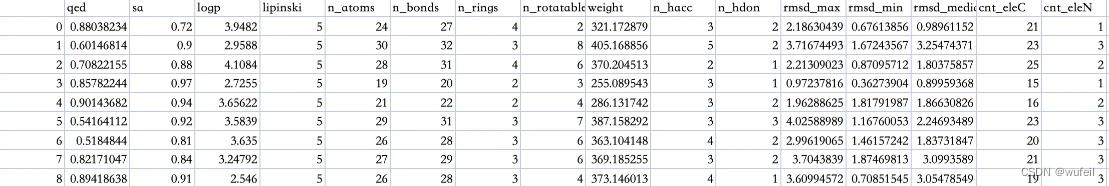
所有生成的QED,sa,logp,lipinski,n_atoms均值分别为:0.6699,0.8657,3.76,4.98,24.7。从这结果来看,生成分子的类药性还不错。
四、训练模型测试
4.1 训练模型
这里从头开始训练MolDiff模型。因此,先下载原始的数据集,下载链接:https://drive.google.com/drive/folders/1WkYIv471SjVwQe6_FfDxFOPC7dSnPY9c 下载其中的sdf.tar.gz文件。

下载完成后,放置在data/geom_drug目录,并解压。
训练完整的MolDiff模型
python scripts/train_drug3d.py \--config ./configs/train/train_MolDiff.yml \--device cuda:0 \--logdir ./logs出现问题,不能正确特征化数据集,所有分子全部跳过,最后输出报错没有分子。
Traceback (most recent call last):File "scripts/train_drug3d.py", line 59, in <module>train_iterator = inf_iterator(DataLoader(File "/home/wufeil/software/anaconda/envs/MolDiff/lib/python3.8/site-packages/torch_geometric/loader/dataloader.py", line 87, in __init__super().__init__(File "/home/wufeil/software/anaconda/envs/MolDiff/lib/python3.8/site-packages/torch/utils/data/dataloader.py", line 349, in __init__sampler = RandomSampler(dataset, generator=generator) # type: ignore[arg-type]File "/home/wufeil/software/anaconda/envs/MolDiff/lib/python3.8/site-packages/torch/utils/data/sampler.py", line 140, in __init__raise ValueError(f"num_samples should be a positive integer value, but got num_samples={self.num_samples}")
ValueError: num_samples should be a positive integer value, but got num_samples=0在try语句中,发生报错的问题在./utils/parser.py文件中的
bond_type = np.array(bond_type, dtype=np.long)
bond_index = np.array([row, col],dtype=np.long)是因为numpy的版本问题,numpy版本高于1.16以后移除了long类型,这一部分修改为int_即可。
bond_type = np.array(bond_type, dtype=np.int_) # wufeil
bond_index = np.array([row, col],dtype=np.int_) # wufeil再次运行训练命令即可开始正确处理数据集,在./data/geom_drugs目录下生成数据集文件processed.lmdb 和 processed.lmdb-lock。(注,如果要从头开始训练,那么要删除这两个文件)。
训练过程比较顺利,使用3090显卡,大约花费了4个半小时。训练的结果保存在logs目录下,目录如下:
.
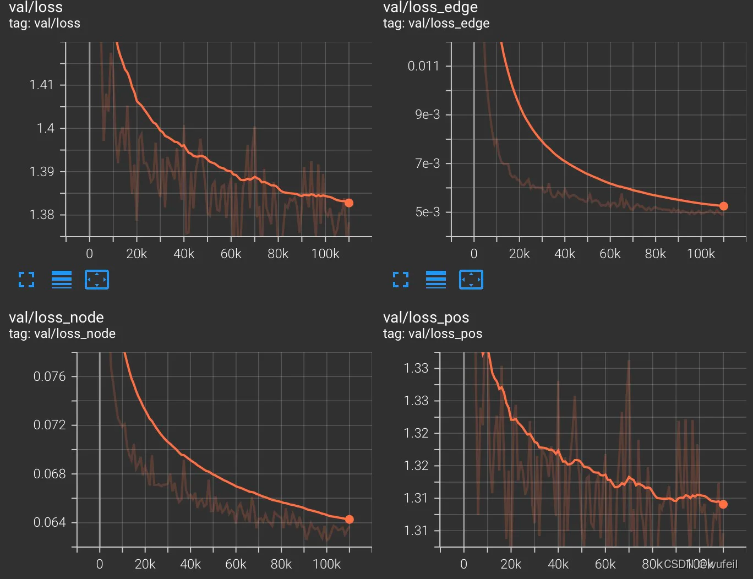
└── train_MolDiff_20240422_142032├── checkpoints├── events.out.tfevents.1713795632.ps.3132011.0├── log.txt└── train_MolDiff.yml2 directories, 3 files训练好的模型的权重保存在checkpoint文件夹内,每隔100次迭代,保存一次。log.txt是寻来你过程的终端输出内容。train_MolDiff.yml是训练/模型参数设置。events.out.tfevents.是tersorboard保存的训练指标,可以打开看损失等曲线,如下:
训练过程的损失:

评估过程的损失:
4.2 训练键预测器
要在采样过程中使用键预测器进行指导,还需要训练键预测器:
python scripts/train_bond.py \--config ./configs/train/train_bondpred.yml \--device cuda:0 \--logdir ./logs注意,配置文件中batch_size按照自己的GPU显存适当的调整。
训练过程的损失,同样在tensorboard中查看。
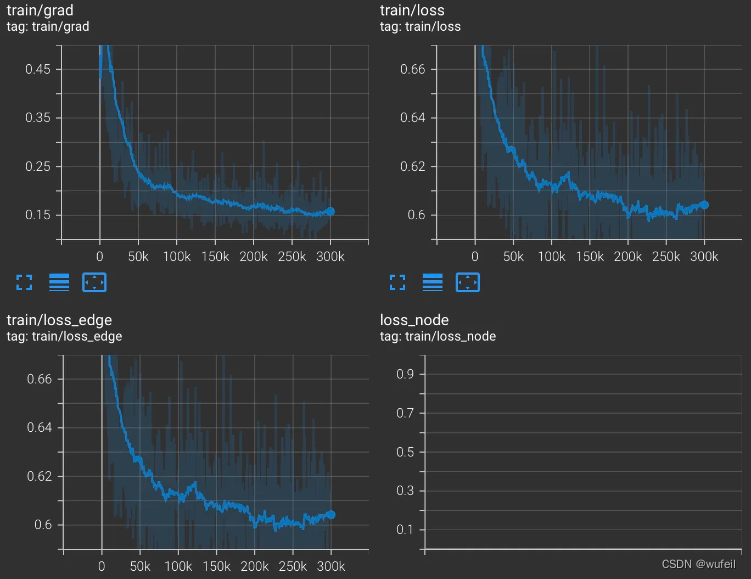
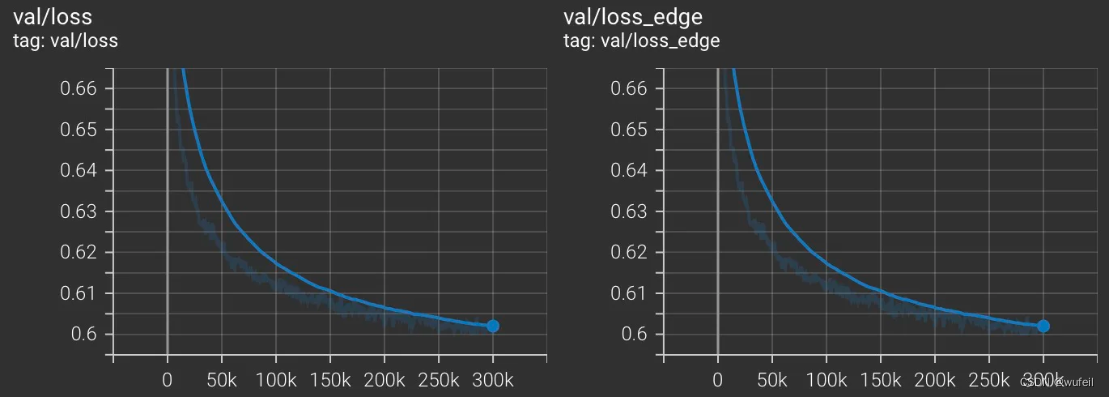
4.3 使用训练的checkpoint生成分子
python scripts/sample_drug3d.py \--outdir ./outputs \--config ./configs/sample/sample_MolDiff-2.yml \--device cuda:0 \--batch_size 32sample_MolDiff-2.yml根据sample_MolDiff.yml修改,改为:
model:checkpoint: logs/train_MolDiff_20240422_142032/checkpoints/99000.ptbond_predictor: logs/train_bondpred_20240426_154859/checkpoints/99000.ptsample:seed: 2023batch_size: 512num_mols: 1000save_traj_prob: 0.02guidance:- uncertainty- 1.e-4运行结束后,生成一个./output/sample_MolDiff-2_20240501_132703文件夹。评估生成的分子:
python scripts/evaluate_all.py \--result_root ./outputs \--exp_name sample_MolDiff-2_20240501_132703 \--from_where generated运行结束后,在./output/sample_MolDiff-2_20240501_132703目录产生mol.csv文件,可以统计到所有生成的QED,sa,logp,lipinski,n_atoms均值分别为:0.6768,0.8689,3.6092,4.5870,24.47。从这结果来看,生成分子的类药性还不错,也与作者提供的checkpoint相当,说明模型可训练性得到验证。
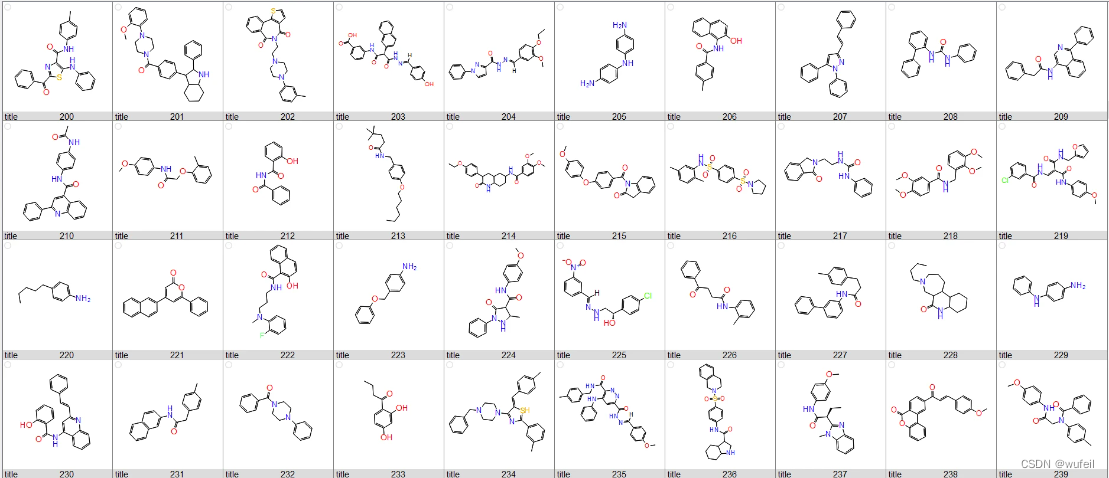
一下是一些生成分子的示例:
五、总结
MolDiff提供了一个新的分子生成框架。在这个框架中,除了原子的扩散,还包括了化学键的扩散。通过特制的化学键添加噪音调度器,合理添加噪音。在分子生成过程中,键类型和键长度之间的紧密关系来指导原子位置的生成,使生成的分子更合理。同时,MolDiff基于SE3网络训练了一个键预测器,在分子生成过程中,利用键预测器对生成分子的坐标和类型进行梯度引导,使得生成的分子更符合真实的化学规则。
作者提供了完整的,便于安装的源代码,非常值得尝试。