新降维可视化
NGS系列文章包括NGS基础、转录组分析 (Nature重磅综述|关于RNA-seq你想知道的全在这)、ChIP-seq分析 (ChIP-seq基本分析流程)、单细胞测序分析 (重磅综述:三万字长文读懂单细胞RNA测序分析的最佳实践教程 (原理、代码和评述))、DNA甲基化分析、重测序分析、GEO数据挖掘(典型医学设计实验GEO数据分析 (step-by-step) - Limma差异分析、火山图、功能富集)等内容。

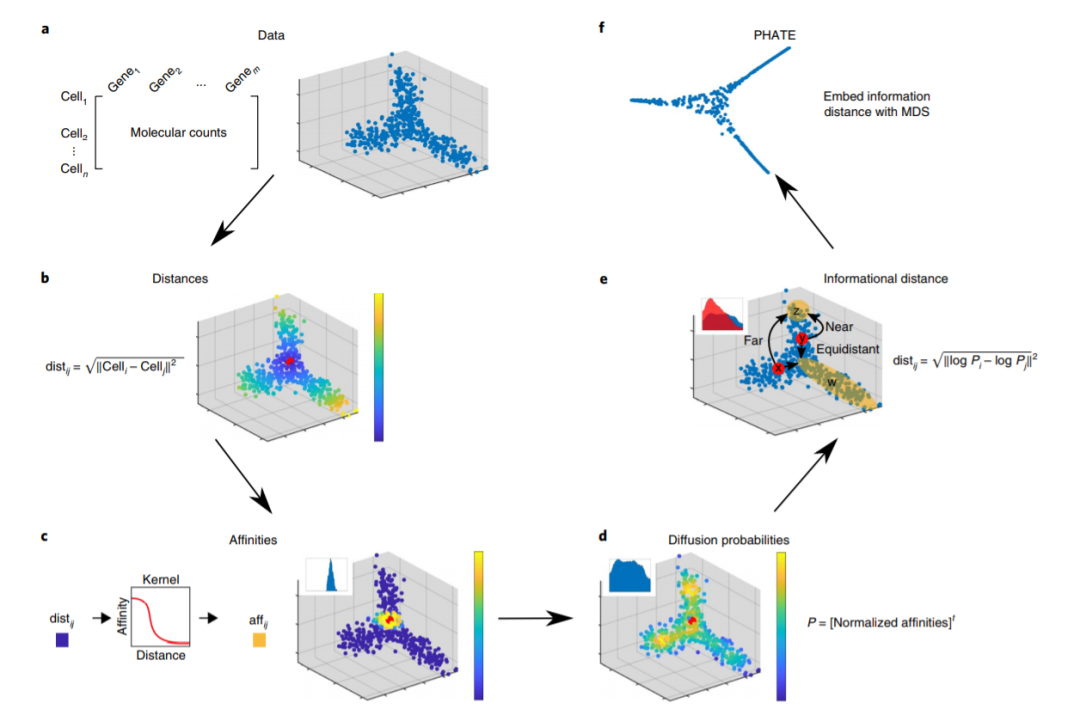
2019年12月3日,美国耶鲁大学Smita发表题为Visualizing structure and transitions in high-dimensional biological data的研究内容,提出PHATE(https://github.com/KrishnaswamyLab/phateR),一种降维可视化方法,使用数据点之间的几何距离信息(即:样品表达谱之间的距离)来捕获局部和全局非线性结构。
PHATE除了可以应用于单细胞分析,还适用于很多数据类型,如质谱数据、Hi-C数据和肠道菌群数据等。
PHATE算法主要为三个步骤:
-
通过局部相似性对局部数据信息进行编码;
-
使用潜在距离在数据中编码全局关系;
-
将潜在距离信息嵌入低维度以进行可视化;

优点与创新点
用于可视化的降维方法很多,其中最常用的是主成分分析(PCA)和t分布随机邻居嵌入(t-SNE)。但是,这些方法对于探索高维生物学数据而言并不是最佳方法:
-
首先,这些方法对噪声敏感。
生物医学数据通常非常嘈杂,例如
PCA和Isomap之类的方法无法显著消除噪声以进行可视化,从而导致无法识别细颗粒度的局部结构。 -
其次,非线性可视化方法(例如t-SNE)通常会扰乱数据中的全局结构。
-
第三,许多降维方法(例如,
PCA和diffusion maps)无法针对二维(2D)可视化进行优化,因为它们不是专门为可视化设计的。
PHATE生成专门用于可视化的低维嵌入,该嵌入可在所需数量的维度中提供数据的局部和全局结构的精确而去噪,无需对数据的结构施加任何假设。
将PHATE与各种人工和生物学数据集上的其他工具进行了比较,发现与其他工具相比,PHATE能够始终如一地保留数据的固有模式,包括连续性过程、分支模式分析和聚类分析。
看文章中的一个小栗子:
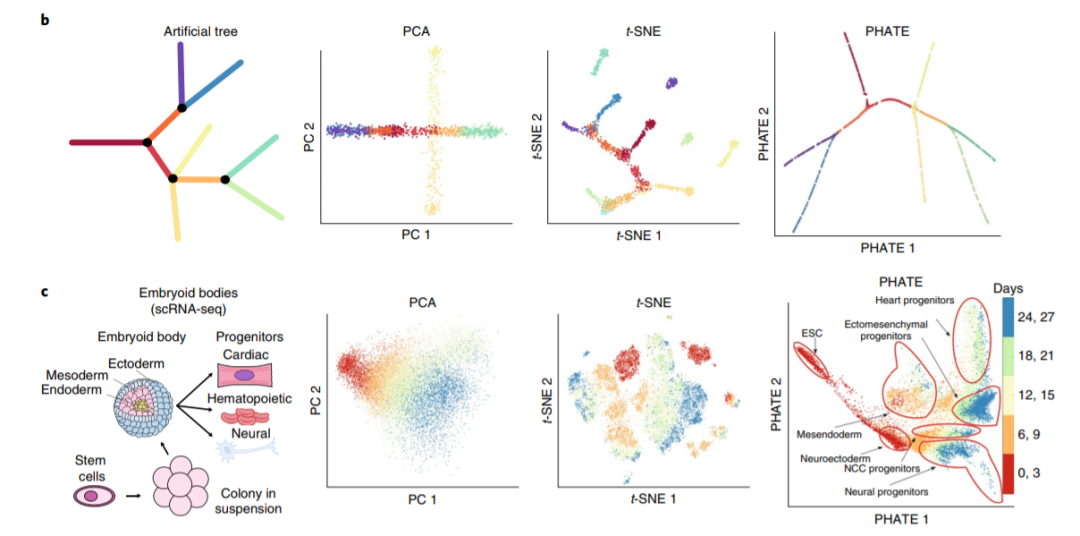
其实从图中就可以看出PATHE能反映局部和全局结构,并且并没有像umap在一定情况下会出现图像的变形。
运行示例
为了在R中使用PHATE,还必须安装Python解释器和包管理工具。建议使用Miniconda3一起安装Python和pip,或者可以从https://pip.pypa.io/en/stable/installing/安装pip。
安装软件包
# 先安装Python包
pip install --user phate# 再安装R包
install.packages("phateR")
if (!require(viridis)) install.packages("viridis")
if (!require(ggplot2)) install.packages("ggplot2")
if (!require(readr)) install.packages("readr")
if (!require(Rmagic)) install.packages("Rmagic")如果从未使用过MAGIC,则还应该从命令行安装MAGIC,如下所示:
pip install --user magic-impute加载软件包:
library(phateR)
library(ggplot2)
library(readr)
library(viridis)
library(Rmagic)加载数据:
在本教程中,我们将按照Paul等人的描述(2015年)分析小鼠骨髓中的髓样和红系细胞。您可以通过下载https://raw.githubusercontent.com/KrishnaswamyLab/phateR/master/inst/examples/bonemarrow_tutorial.Rmd,在RStudio中将其打开并运行本教程。示例数据位于PHATE GitHub存储库中,可以直接从Web上加载它们。
# 加载数据
bmmsc <-read_csv("https://github.com/KrishnaswamyLab/PHATE/raw/master/data/BMMC_myeloid.csv.gz")
## Warning: Missing column names filled in: 'X1' [1]## Parsed with column specification:
## cols(
## .default = col_double(),
## X1 = col_character()
## )与之前数据不同的是,这个数据的每一行是一个样品,每一列是一个基因。如果自己的数据格式不符,记得转换下。
bmmsc <- bmmsc[,2:ncol(bmmsc)]
bmmsc[1:5,1:10]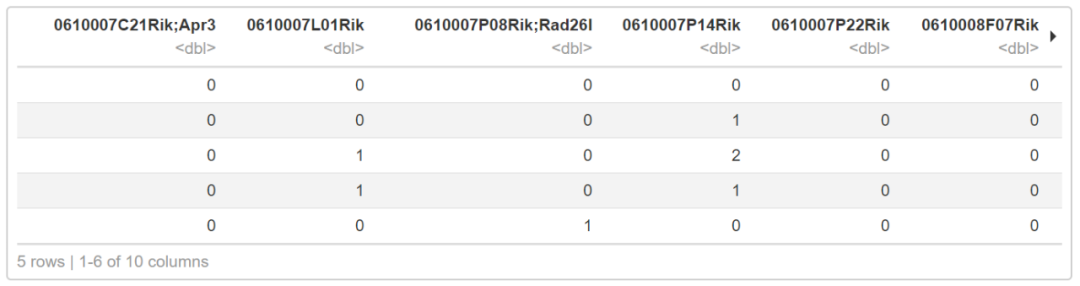
过滤数据:
删除低表达的基因和细胞。
# 至少在10个细胞内表达基因
keep_cols <- colSums(bmmsc > 0) > 10
bmmsc <- bmmsc[,keep_cols]
# look at the distribution of library sizes
ggplot() +geom_histogram(aes(x=rowSums(bmmsc)), bins=50) +geom_vline(xintercept = 1000, color='red')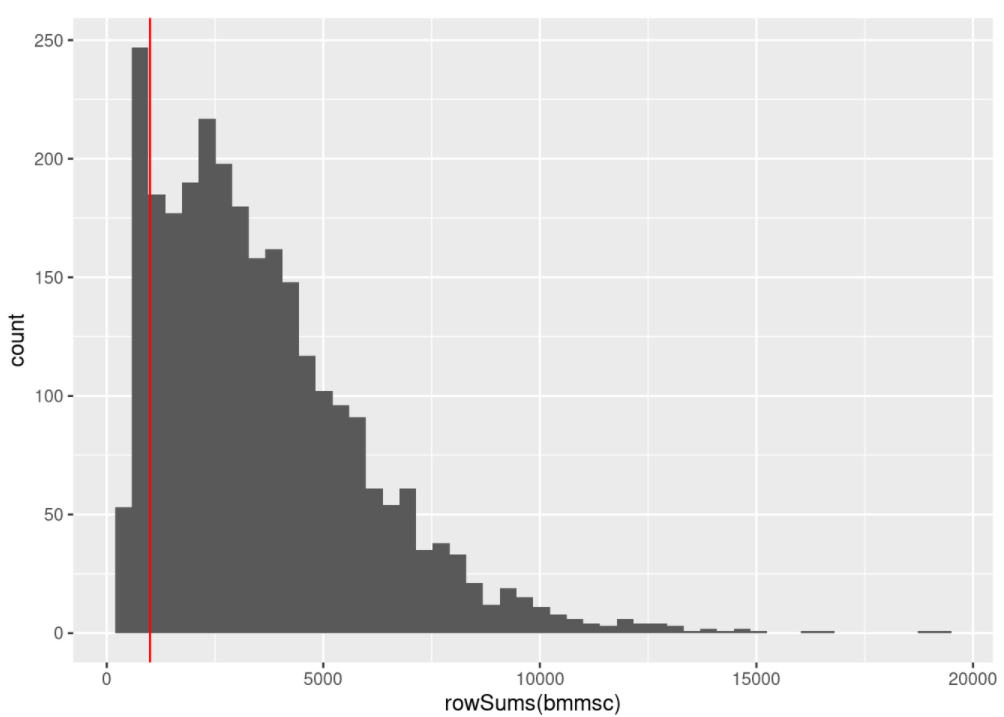
# 保证细胞至少检测到1000UMI
keep_rows <- rowSums(bmmsc) > 1000
bmmsc <- bmmsc[keep_rows,]Normalizing data:
在使用PHATE之前将文库大小归一化并转换数据, 这里作者取平方根 (因为Log转换时需要加一个”pseudocount”以避免log(0),而开方则不需要考虑这一点):
bmmsc <- library.size.normalize(bmmsc)
bmmsc <- sqrt(bmmsc)运行PCA:
bmmsc_PCA <- as.data.frame(prcomp(bmmsc)$x)使用ggplot2绘制结果。通过髓样标记基因Mpo为图上色:
ggplot(bmmsc_PCA) +geom_point(aes(PC1, PC2, color=bmmsc$Mpo)) +labs(color="Mpo") +scale_color_viridis(option="B")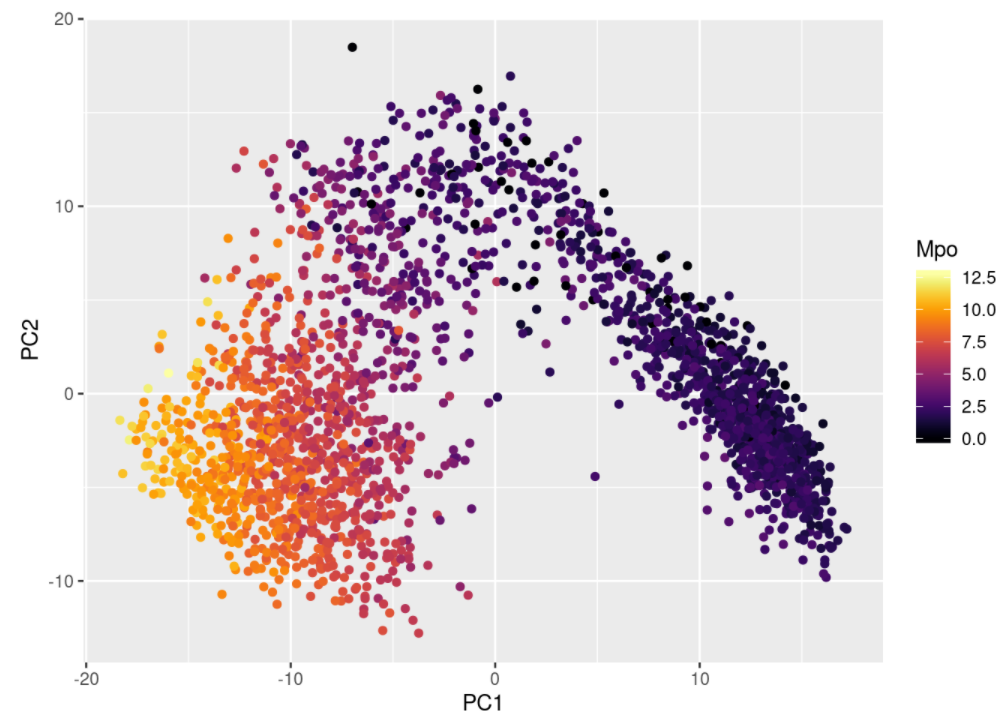
ggsave('BMMSC_data_R_PCA.png', width=5, height=5)由于数据自身结构复杂,而PCA又致力于保存全局差异,样品并不能区分太开 ——但我们可以看到,这些数据沿第一主成分被髓样红细胞广泛分隔开来。
运行PHATE:
# run PHATE
bmmsc_PHATE <- phate(bmmsc)使用ggplot2绘制结果。
ggplot(bmmsc_PHATE) +geom_point(aes(PHATE1, PHATE2, color=bmmsc$Mpo)) +labs(color="Mpo") +scale_color_viridis(option="B")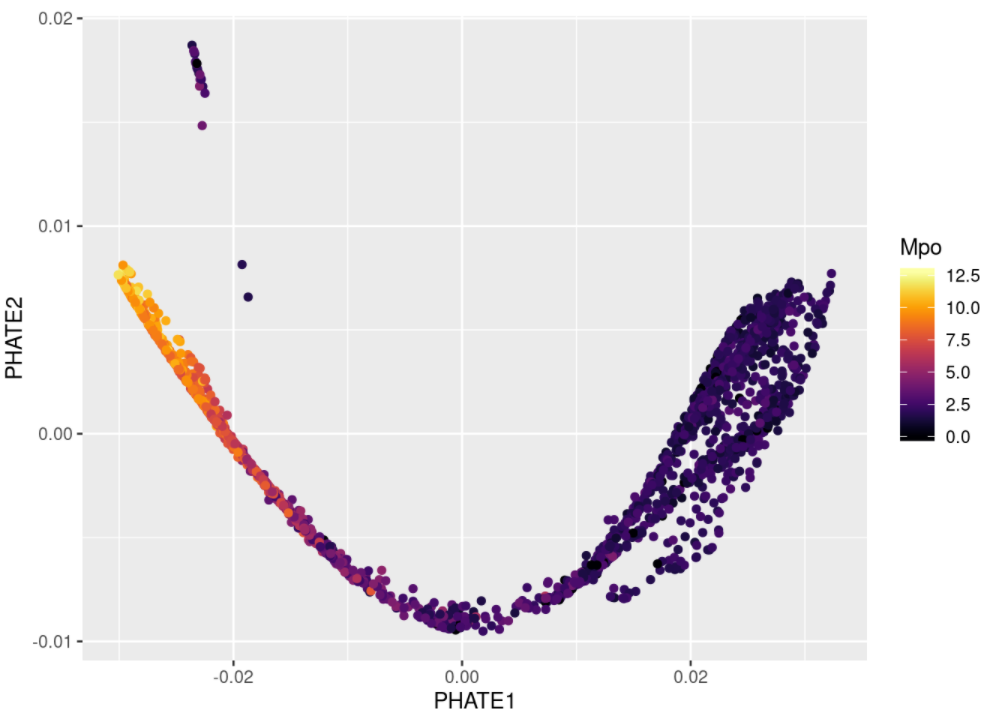
使用新参数重新运行PHATE:
bmmsc_PHATE <- phate(bmmsc, knn=4, decay=100, t=10, init=bmmsc_PHATE)
ggplot(bmmsc_PHATE) +geom_point(aes(PHATE1, PHATE2, color=bmmsc$Mpo)) +labs(color="Mpo") +scale_color_viridis(option="B")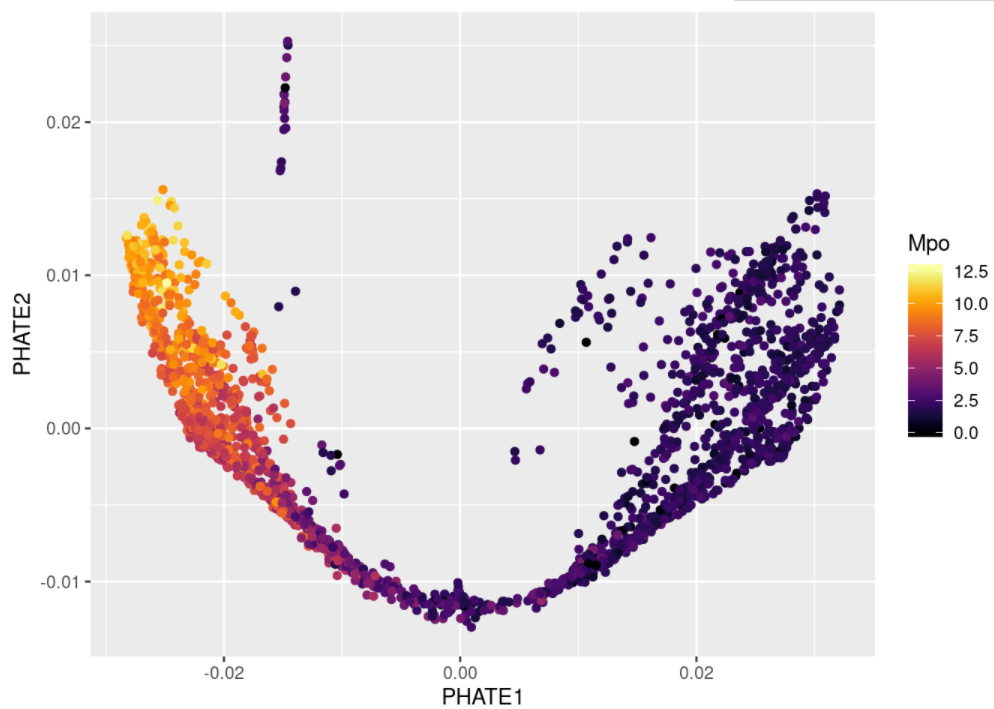
用MAGIC可视化PHATE上的基因:
许多基因都受到dropout的困扰,以致无法检测到其表达。以髓系干细胞标记Ifitm1为例。
ggplot(bmmsc_PHATE) +geom_point(aes(PHATE1, PHATE2, color=bmmsc$Ifitm1)) +labs(color="Ifitm1") +scale_color_viridis(option="B")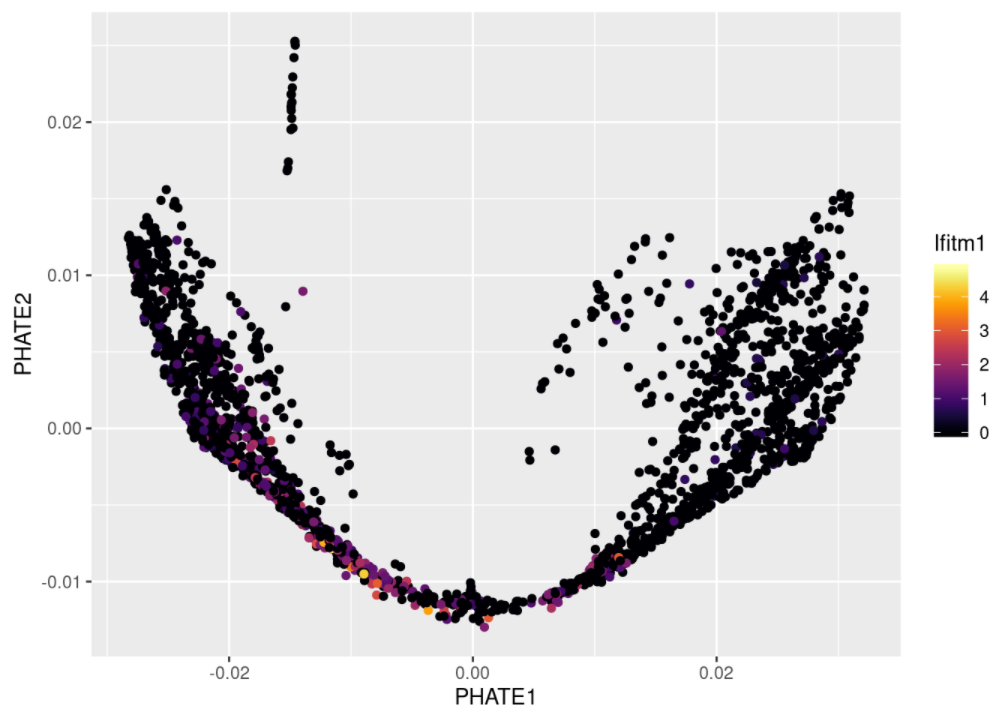
即使我们期望中间亚群完全是干细胞,但这些细胞中许多仍不表达Ifitm1。让我们运行MAGIC(是一种用于去除高维数据噪声的算法,该算法最常应用于单细胞RNA测序数据),然后again。
获取更多有关MAGIC的内容:https://github.com/KrishnaswamyLab/MAGIC
bmmsc_MAGIC <- magic(bmmsc, t=4, genes="Ifitm1")
ggplot(bmmsc_PHATE) +geom_point(aes(x=PHATE1, y=PHATE2, color=bmmsc_MAGIC$result$Ifitm1)) +scale_color_viridis(option="B") +labs(color="Ifitm1")
这样看起来合理多了,有时候图像显示表达程度并不明显,这时我们可以通过调整colorbar的参数,帮助显示更为合理。
撰文:Tiger
编辑:生信宝典
参考文献:
Moon KR, van Dijk D, Wang Z, Gigante S, Burkhardt DB, Chen WS, Yim K, Elzen AVD,


















