Total body segmentation using MONAI Deploy on an AMD GPU — ROCm Blogs

2024 年 4 月 4 日 作者: Vara Lakshmi Bayanagari.
医疗开放网络人工智能(MONAI)是一个开源组织,提供最先进的医疗成像模型的 PyTorch 实现,涵盖从分类和分割到图像生成的各个方面。MONAI 的生命周期为研究人员、临床医生和同领域的贡献者提供了三种不同的端到端工作流工具:MONAI Core、MONAI Label 和 MONAI Deploy。
MONAI Core 提供可用于端到端训练和推理的模型。MONAI Label 是一个智能工具,可基于用户交互自动标记数据集。MONAI Deploy 是一个打包工具,提供命令行接口(CLI)命令(如 monai-deploy exec、`monai-deploy package`)来部署可以投入生产的 AI 应用程序。此外,它允许将您的应用程序打包成 MAP/Docker 镜像,可以使用 monai-deploy run 命令运行。MONAI Deploy 提供了打包的示例应用程序(例如脾脏分割和乳腺密度分类)。
正如MONAI 的教程所解释的那样,创建一个应用程序/SDK 需要我们定义一组运算符,这些运算符将按顺序执行。运算符是应用程序中的一个工作单元,可以有多个虚拟输入/输出端口。每个端口通过将前一个运算符的输出端口的输出传递给当前运算符的输入端口,从而促进应用程序的进展。例如,脾脏分割应用程序的官方教程使用了五个预定义的运算符:
-
DICOMDataLoaderOperator: 从输入文件夹中的
.dcm文件加载 DICOM(标准医疗体积存储格式)研究。 -
DICOMSeriesSelectorOperator: L根据规则集从每个 DICOM 研究中加载特定数据系列。该示例中使用的规则集使用正则表达式过滤每个研究中的 CT 文件。或者,我们还可以添加多个条件,例如:`Spleen & CT 1` 为第一个条件,`Abdomen & CT 2` 为第二个条件,如此处所示:
{ "selections": [{"name": "CT Series","conditions": {"StudyDescription": "(.*?)","Modality": "(?i)CT","SeriesDescription": "(.*?)"}} ] } -
DICOMSeriesToVolumeOperator: 将 DICOM 实例转换为 NumPy 数组进行进一步处理。
-
MonaiBundleInferenceOperator: AI 运算符加载通用的 TorchScript 模型,选择设备,执行输入预处理和输出后处理。用于加载感兴趣模型的
model.ts捆绑包位于数据文件夹中,并在命令model-deploy exec -m model.ts -i dcm -o output中传递。此运算符通常适用于任何 MONAI 捆绑包,并适应不同的配置参数,例如输入转换、数据类型、输入参数和模型推理(如 `bundle('model/extra/metadata.json' 中定义。警告 - 不要将捆绑包名称更改为 model.ts 以外的任何名称。这样做可能会在读取元数据配置文件时导致错误)。 -
DICOMSegmentationWriterOperator: AI 模型的每像素二进制输出由此运算符处理以生成预测的分割脾脏。然后将其转换为 DICOM 实例并保存到磁盘。
在这篇博客中,我们将讲解使用 NIfTI 体积进行全身分割的 MONAI Deploy 示例。现有示例使用运算符探索 DICOM 数据和元数据,并不容易与 NIfTI 数据集成。本博客作为编写自定义 NIfTI 数据运算符的入门。
您可以在这个GitHub 文件夹中找到与本博客相关的文件。
实现
要跟随本博客,你需要:
-
安装基于 ROCm 的
torch和torchvision。这个实验是在 ROCm 5.7.0 上进行的。参阅系统要求 了解硬件和操作系统的支持。pip install torch==2.0.1 torchvision==0.15.2 -f https://repo.radeon.com/rocm/manylinux/rocm-rel-5.7/
-
安装 MONAI Deploy 应用程序 SDK 0.5.1 版本。这将帮助你使用
monai-deploy run和monai-deploy package等命令来启动应用程序。pip install monai-deploy-app-sdk==0.5.1
-
从 zenodo.org 下载一个用于推理的单一测试对象到输入文件夹,即 ts_data
curl https://zenodo.org/records/10047263/files/Totalsegmentator_dataset_small_v201.zip --output ts.zipunzip ts.zip s0011/ct.nii.gzmv s0011/ct.nii.gz ts_data// Remove downloadsrm -r s0011rm ts.zip
-
安装其他所需的库。
pip install monai pydicom highdicom SimpleITK Pillow nibabel scikit-image numpy-stl trimesh
代码
TotalSegmentator 是一个开源项目,它使用与 ResNet 等效的网络从整个人体 CT 扫描图像中分割出 105 个部位,包括颅骨、胃、心脏、肺和消化道。所有类别的列表存储在 这里。

我们使用 MONAI 预训练模型结合 MONAI Deploy App SDK 来构建 TotalSegmentator 应用程序。您可以在 Project-MONAI GitHub 仓库 中找到用于预训练该模型的训练配置。我们将使用此配置文件构建模型的预处理和后处理管道,并加载 预训练的检查点。确保下载检查点并将其放在输入文件夹中,即 ts_data,否则脚本将引发如下代码块所示的错误。
curl https://drive.google.com/file/d/1c3osYscnr6710ObqZZS8GkZJQlWlc7rt/view?usp=share_link -o ./ts_data/model.pt
构建 TotalSegmentator 应用程序的第一个操作符 - PreprocessNiftiOperator,它从输入路径加载 NIfTI 文件和预训练模型。
@md.output('image', np.ndarray, IOType.IN_MEMORY)
@md.output('model', str, IOType.IN_MEMORY)
class PreprocessNiftiOperator(Operator):def __init__(self):super().__init__()def compute(self, op_input, op_output, context):input_path = op_input.get().pathlogging.info(input_path)files = os.listdir(input_path)if model not in files:logging.error('Cannot find model.pt file in the input path')returninput_file = files[0] # ct.nii.gz filenifti_input = nib.load(os.path.join(input_path,input_file)).get_fdata()op_output.set(nifti_input, 'image')op_output.set(os.path.join(input_path, 'model.pt'), 'model')
接下来,定义 TotalSegmentator 应用程序的核心操作符 SegInferenceOperator,该操作符预处理数据,加载 SegResNet 模型,并对输出进行推理和后处理。该操作符的装饰器定义了临时数据存储中 InputContext 和 OutputContext 端口的数据类型和存储类型。此数据存储是在应用程序启动期间在 MONAI 工作目录 <current_dir>/.monai_workdir/ 内创建的,用于存储中间结果。`SegInferenceOperator` 期望两个输入端口分别保存 NumPy 数据和预训练 ckpt 的路径。它输出一个‘输出已保存’消息,以成功结束操作符。
@md.input('image', np.ndarray, IOType.IN_MEMORY)
@md.input('model', str, IOType.IN_MEMORY)
@md.output('output_msg', str, IOType.IN_MEMORY)
class SegInferenceOperator(Operator):def __init__(self, roi_size=96,pre_transforms = True,post_transforms = True,model_name = 'SegResNet'):super().__init__()def compute(self, op_input, op_output, context):input_image = op_input.get("image") #ndarrayinput_image = np.expand_dims(input_image, axis=-1)# input_image = torch.tensor(input_image).float()# 没有使用标准的 MonaiSegInferenceOperator 因为它深度集成了 DICOM 样式元数据渲染,对 NIfTI 数据不够灵活。此外,它从应用上下文中加载 monai.core.model.Model 而不是 torch.nn.Modulenet = SegResNet(spatial_dims= 3,in_channels= 1,out_channels= 105,init_filters= 32,blocks_down= [1,2,2,4],blocks_up= [1,1,1],dropout_prob= 0.2)#加载从 InputContext 中提取的预训练检查点net.load_state_dict(torch.load(op_input.get('model')))device = torch.device("cuda" if torch.cuda.is_available() else "cpu")net.to(device)# 定义要应用的变换列表pixdim = [3.0, 3.0, 3.0]self.pre_transforms = Compose([EnsureChannelFirstd(keys='image', channel_dim=-1),Orientationd(keys='image', axcodes='RAS'),# Resized(keys="image", spatial_size=(208,208,208), mode="trilinear", align_corners=True),Spacingd(keys='image', pixdim=pixdim, mode='bilinear'),NormalizeIntensityd(keys='image', nonzero=True),ScaleIntensityd(keys='image', minv=-1.0, maxv=1.0)])self.post_transforms = Compose([Activationsd(keys='pred', sigmoid=True),AsDiscreted(keys='pred', argmax=True),Invertd(keys='pred', transform=self.pre_transforms, orig_keys='image'),SaveImaged(keys='pred', output_dir='/home/aac/monai-2/output', meta_keys='pred_meta_dict')])dataset = Dataset(data=[{'image':input_image}], transform=self.pre_transforms)dataloader = DataLoader(dataset, batch_size=1)for i in dataloader:logging.info(f'Preprocessed input size is {i["image"].shape}')o = net(i['image'].to(device))logging.info(f'Output size is {o.shape}')i['pred'] = o.detach()out = [self.post_transforms(x) for x in decollate_batch(i)]op_output.set("Output saved",'output_msg')
您现在可以使用这两个操作符来构建这个应用程序:
import logging# 需要设置 SegmentDescription 属性。从 pydicom.sr.codedict 导入,因为它不是应用程序 SDK 包的一部分。
from pydicom.sr.codedict import codes
from operators import PreprocessNiftiOperator, SegInferenceOperator
from monai.deploy.core import Application, resource@resource(cpu=1, gpu=1, memory="7Gi")
class TotalBodySegmentation(Application):def __init__(self, *args, **kwargs):super().__init__(*args, **kwargs)def run(self, *args, **kwargs):# 此方法调用基类来运行。可以省略,如果只是简单地调用它。logging.info(f"Begin {self.run.__name__}")super().run(*args, **kwargs)logging.info(f"End {self.run.__name__}")def compose(self):logging.info(self._context.model_path)"""创建应用程序特定的操作符并将它们连接到处理 DAG 中。"""logging.info(f"Begin {self.compose.__name__}")preprocess_op = PreprocessNiftiOperator()inference_op = SegInferenceOperator()self.add_flow(preprocess_op, inference_op, {'image': 'image', 'model': 'model'})logging.info(f"End {self.compose.__name__}")if __name__ == "__main__":logging.basicConfig(level=logging.DEBUG)app_instance = TotalBodySegmentation(do_run=True)
通过以下命令对测试示例进行分割;结果作为 NIfTI 对象保存在输出文件夹中。
monai-deploy exec total_body_segmentation.py -i ts_data/ -o output/ -l DEBUG
结果
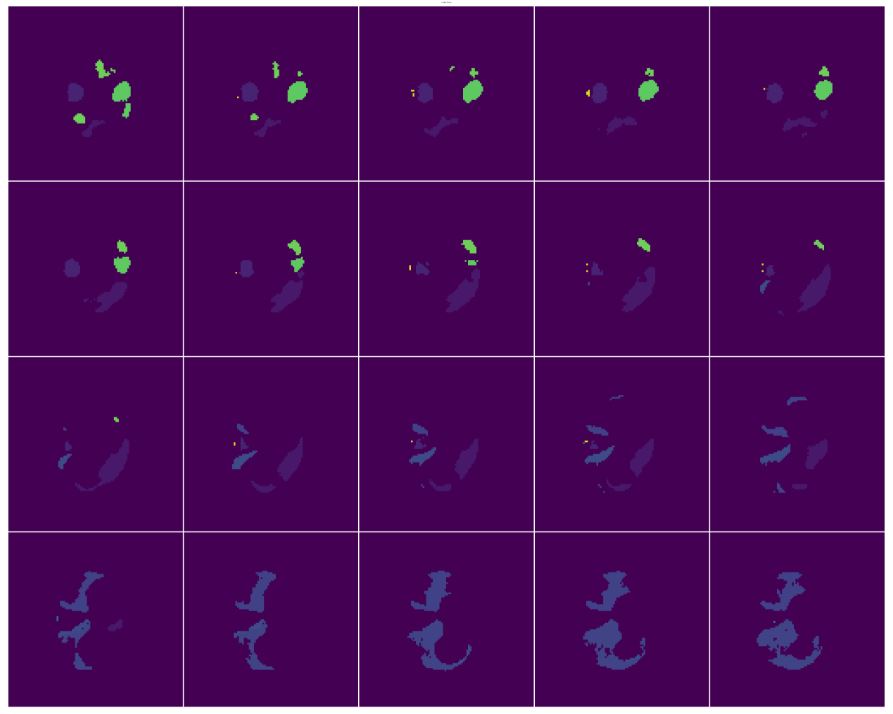
使用MONAI的`matshow3d`库进行可视化:
from monai.visualize import blend_images, matshow3d
import matplotlib.pyplot as plt
import nibabel as nib
import osoutput_image = nib.load(os.path.join('./output/0/0_trans.nii.gz')).get_fdata()
print(output_image.shape)
matshow3d(volume=output_image[:,:,200:300],fig=None,title="output image",figsize=(100, 100),every_n=5,frame_dim=-1,show=True,# cmap="gray",
)
注意,每个输入体积的切片上都有多个部分被分割出来。你可以使用MONAI Label来进行每个分割部分的3D可视化。
你可以使用monai-deploy命令将应用程序打包并上传为Docker镜像。使用打包应用程序时使用的Docker标签来检索并运行它
实际案例
开发了TotalSegmentator的团队还使用MONAI Deploy创建了另一个基于他们自定义模型和训练参数的应用程序,您可以在此查看: application 。他们最近在生产环境中发布了一个原型,以促进医学研究与最新AI工作流程的结合,从而提高其采用率。这是MONAI Deploy的一个实际生产用例。该应用程序不仅生成分割结果,还生成PDF报告,这对医生和研究人员非常有用。
鸣谢
我们要感谢Project MONAI在AI医学影像应用中的开源贡献。