随着 ChatGPT 的快速崛起,大型语言模型(LLM)已经在人类语言建模领域展示出了其非凡的能力。无论是证明数学公式、编写代码,还是以不同的风格创作诗歌,LLM 都能胜任。然而,尽管 LLM 在人类语言的掌握上已达到了精通的程度,但在面对分子语言系统时,它仍显得较为稚嫩。
分子语言由数百万个组成成分经过数十亿年的相互作用和演化形成,其复杂性极高,对于人类来说仍然是一个挑战。最近的研究开始尝试将大型语言模型的强大能力扩展到分子领域,试图理解和预测复杂的分子语言,以提高药物发现的速度和质量,从而为该领域带来革命性的变革。
本文梳理了多篇分子语言模型的相关研究,希望能带来更多对 LLM 在药物小分子领域应用发展的深入理解和启发。
01
/ /
论文标题:
Language models can learn complex molecular distributions
论文地址:
https://www.nature.com/articles/s41467-022-30839-x
为了有效地探索庞大的分子空间,深度生成模型,特别是语言模型,已逐渐成为应对此一挑战的最有潜力的手段之一。这些模型能够理解训练分子语言的语法,并获得生成有效且相似的分子的能力,这对于各种下游应用,如设计功能性化合物,具有至关重要的意义。

▲ 图1:RNN 模型能更精准地学习训练分布的更多特征,而 CGVAE 和 JTVAE 几乎无法掌握主要分布特征。
在这篇文章中,作者研究了简单语言模型学习分子更复杂分布的能力。为此,作者通过编译更大,更复杂的分子分布来引入几个具有挑战性的生成建模任务,并评估语言模型对每个任务的能力。语言模型可以准确地生成:ZINC15 中得分最高的被惩罚 LogP 分子的分布,PubChem 中最大分子的多模态分子分布。这一发现证明,语言模型具有学习复杂分子分布的强大能力,并且其性能优于图模型。
02
/ /
论文标题:
Chemformer: a pre-trained transformer for computational chemistry
论文地址:
https://iopscience.iop.org/article/10.1088/2632-2153/ac3ffb/pdf
项目链接:
https://github.com/MolecularAI/Chemformer
本研究旨在解决计算化学领域中的资源挑战,通过使用自监督预训练和迁移学习的方法,为各种任务提供一个快速应用的模型。研究者提出了一种基于 SMILES,即“化学语言”的预训练模型——Chemformer。通过自监督预训练,模型可以在下游任务上提高收敛速度和性能。Chemformer 模型基于 BART 语言模型,采用了 Transformer 的 Encoder-Decoder 架构堆栈,可以很好地应用于 Seq2seq 任务,如反应预测和分子优化。同时,BART 模型也可以很容易地应用于判别性任务,如性质预测,只需使用其 Encoder 部分即可。
Chemformer 的训练过程分为两个阶段:自监督预训练和针对下游任务的微调。在自监督预训练阶段,使用大量未标记的 SMILES 数据集,通过三种不同的自监督预训练任务和模型架构进行训练。在针对下游任务的微调阶段,将预训练的 Chemformer 模型应用到特定的下游任务中,并进行微调。在微调过程中,使用了多任务学习的方法,可以同时优化多个任务,提高了化学信息学研究的效率。
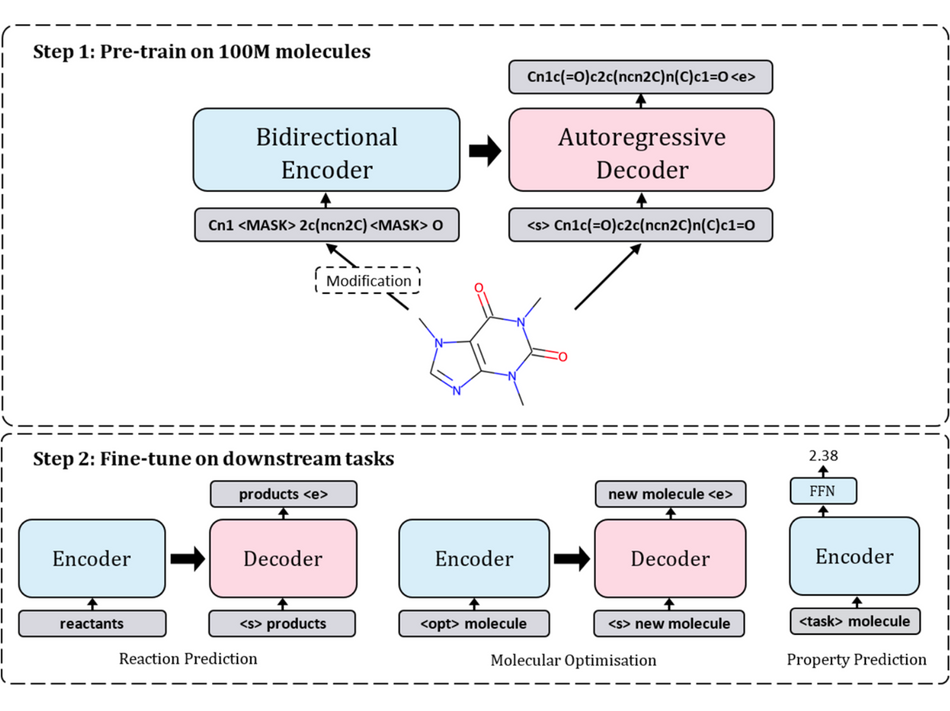
▲ 图2:Chemformer 的模型架构
Chemformer 在化学反应预测、分子优化和性质预测任务上的实验结果表明:Chemformer 模型可以轻松地应用于各种下游任务;自监督预训练可以提高 Chemformer 模型在下游任务上的收敛速度,同时可以显著提高这些任务的结果;当训练时间有限时,迁移学习和新的数据增强策略的组合能够在所有下游 Seq2seq 任务(化学反应预测、分子优化)中产生最先进的结果。
03
/ /
论文标题:
MolGPT: Molecular Generation Using a Transformer-Decoder Model
论文地址:
https://pubs.acs.org/doi/pdf/10.1021/acs.jcim.1c00600
项目链接:
https://github.com/devalab/molgpt
为了开发一种能够生成具有特定性质分子的生成模型,本文使用了一种基于 Transformer-decoder 的生成式模型 MolGPT,其架构包括以下几个部分:
输入编码器:将 SMILES 字符串编码为向量表示,以便输入到模型中。
Transformer-decoder 模型:由多个 transformer 模块和一个 decoder 模块组成。每个 transformer 模块包括多头自注意力机制和前馈神经网络,用于学习输入序列中的上下文信息。decoder 模块使用自注意力机制和编码器-解码器注意力机制来生成下一个 SMILES 字符。
输出解码器:将生成的 SMILES 字符串解码为分子结构。
在训练过程中,MolGPT 使用掩码自注意力机制来预测下一个 SMILES 字符。在生成过程中,MolGPT 使用贪心搜索或基于束搜索的方法来生成具有所需性质的分子。
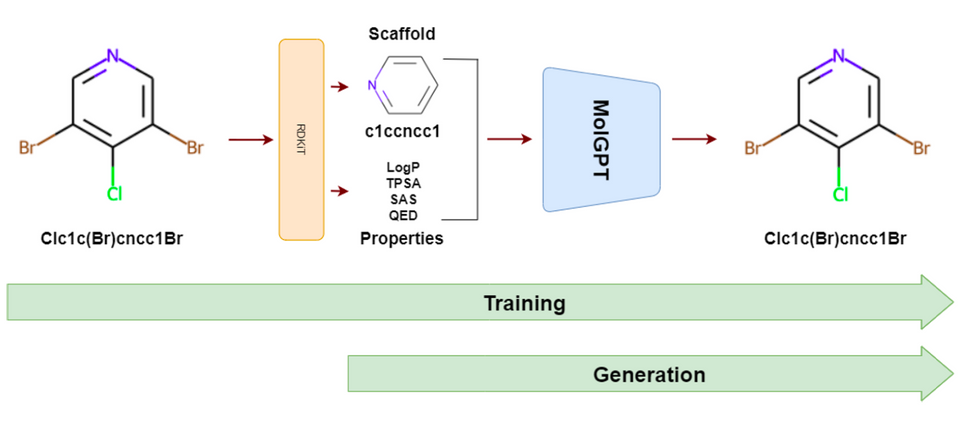
▲ 图3:MolGPT 的模型架构
这篇文章主要进行了分子生成任务的实验,旨在评估 MolGPT 模型的性能和多样性。实验结果表明,MolGPT 生成的分子在多个性质上与目标分子具有相似的性质,包括分子量、脂水分配系数、旋转键数等。此外,MolGPT 生成的分子具有较高的多样性和独特性,与其他现有的分子生成模型相比,MolGPT 生成的分子更加多样化和有效。
04
/ /
论文标题:
Domain-Agnostic Molecular Generation with Self-feedback
论文地址:
https://arxiv.org/pdf/2301.11259.pdf
项目链接:
https://github.com/zjunlp/MolGen
本文旨在开发一种能够生成具有特定化学性质的分子的深度学习模型。作者提出了一种名为 MOLGEN 的预训练分子语言模型。该模型使用 SELFIES 作为分子语言,SELFIES 是一种无语法和语义障碍的分子表示方法,比 SMILES 更加鲁棒。MOLGEN 采用 Encoder-Decoder 的架构,使用自回归的方式生成分子。
该方法分为三个主要部分:
预训练第一阶段:使用大规模的分子数据集对模型进行预训练,以获取分子的结构和语法信息。这个预训练过程使用了超过 1 亿个分子 SELFIES 进行重构,从而获得分子的内在结构和语法洞察力。
预训练第二阶段:引入了不依赖特定领域的分子前缀,以增强模型对不同领域的理解能力。这种前缀调整方法不仅仅更新前缀矩阵,还能有效增强模型理解不同领域的能力。
微调阶段:提出一种自反馈机制,根据生成的分子的性质评估结果来调整模型的参数,以逐步优化生成的分子。这种自反馈机制使得模型能够更好地生成具有期望性质的分子。通过将模型与优化目标对齐,鼓励模型对更优的候选分子分配更高的概率,从而实现分子性质的优化。

▲ 图4:MolGen 的模型架构
实验部分,作者对 MOLGEN 进行了广泛的评估和比较。实验结果表明,在天然产物和人工合成分子领域,MOLGEN 在生成具有所需属性的分子方面均表现出色,并且能够捕捉到训练数据中的分子属性和结构特征。
05
/ ICML 2023 Poster /
论文标题:
A deep-learning system bridging molecule structure and biomedical text with comprehension comparable to human professionals
论文地址:
https://www.nature.com/articles/s41467-022-28494-3
项目链接:
https://github.com/thunlp/KV-PLM
为了弥合分子结构和生物医学文本之间的差距,以增强生物医学知识获取的机器阅读系统。现有系统仅能处理分子结构信息或生物医学文本信息中的一种,限制了它们的通用性和性能。通过联合建模这些异构数据源,可以开发出一个知识丰富的机器阅读系统,以获取元知识并更好地理解分子。本文提出 KV-PLM,它是一个统一的预训练语言模型,可以处理分子结构和生物医学文本。
KV-PLM 模型的具体架构是基于 BERT 预训练语言模型的统一模型,用于处理分子结构和生物医学文本。该模型通过自监督的语言模型预训练技术,在大规模生物医学数据上学习元知识,实现对分子的知识丰富和多功能阅读任务。
具体架构如下所示:
输入处理:分子结构首先被序列化为 SMILES 字符串,并使用字节对编码(BPE)进行分段,以便与文本数据一起处理。
BERT作为骨干网络:KV-PLM 使用 BERT 作为预训练语言模型的骨干网络,用于学习分子结构和生物医学文本的表示。
分子结构和文本交互:KV-PLM 通过在模型中建立分子结构和文本之间的交互通道,实现两者之间的联合建模。这样,模型可以同时学习分子结构和文本的表示,并捕捉它们之间的关联。
自监督预训练:KV-PLM 使用自监督的语言模型预训练技术,在大规模生物医学数据上进行预训练,学习分子结构和文本的表示。这种无监督的预训练方法使得模型能够获取元知识,并在下游任务中展现出良好的性能。
KV-PLM 的模型架构。
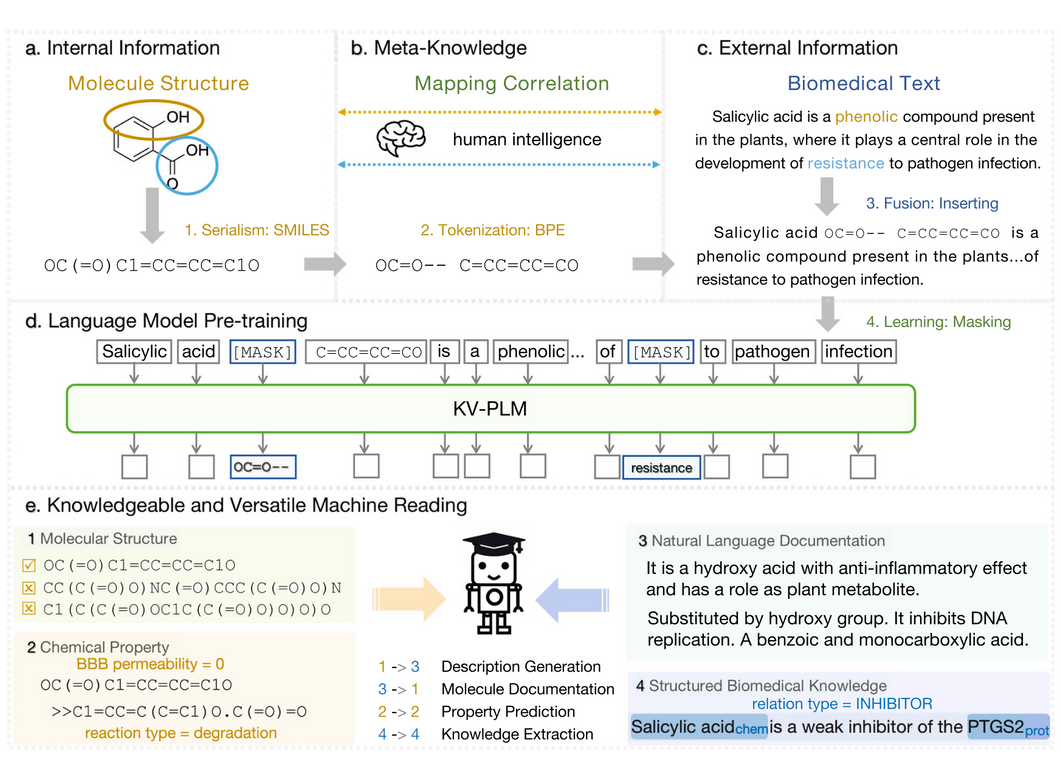
▲ 图5:KV-PLM 的模型架构
该研究进行了以下任务和实验:
1. 分子结构和生物医学文本的知识丰富和多功能阅读任务:通过 KV-PLM 模型,在 MoleculeNet 数据集上进行了 SMILES 属性分类任务,包括 BBBP、SIDER、TOX21 和 HIV 等四个常用的分类任务主题。实验结果表明,KV-PLM 模型在这些任务上取得了良好的性能,能够进行知识丰富和多功能的阅读任务。
2. 生物医学命名实体识别(NER)任务:使用 BC5CDR NER 数据集和 ChemProt 数据集进行了命名实体识别任务。实验结果表明,KV-PLM 模型在 BC5CDR 数据集上能够准确地识别化学分子和疾病等实体,并在 ChemProt 数据集上能够有效地识别化学物质和蛋白质之间的关系。
实验结果表明,KV-PLM 模型能够有效地处理分子结构和生物医学文本的信息;能够准确地识别实体,并在关系抽取任务中能够识别化学物质和蛋白质之间的关系;通过预训练能够有效地学习内部结构知识,并在自然语言任务中展现出良好的性能。
06
/ /
论文标题:
Translation between Molecules and Natural Language
论文地址:
https://arxiv.org/pdf/2204.11817.pdf
项目链接:
https://github.com/blender-nlp/MolT5
为了实现分子和自然语言之间的双向翻译,以便更好地控制分子的发现和理解,作者提出了一个自监督学习框架 MolT5,用于预训练模型,使其能够同时处理大量未标记的自然语言文本和分子字符串。
为此,作者定义了两个新的任务来实现这个目标:分子字幕和基于文本的 de novo 分子生成。对于任何给定的分子,分子字幕的目标是描述分子及其功能。在高层次上,分子字幕与图像字幕非常相似。但是,由于可能的字幕语言多样性增加,分子字幕的情况要困难得多。对于基于文本的 de novo 分子生成,目标是根据给定的文本描述生成新的分子。这种方法可以生成具有特定功能(如味道)而不是属性的分子,从而实现定制分子。
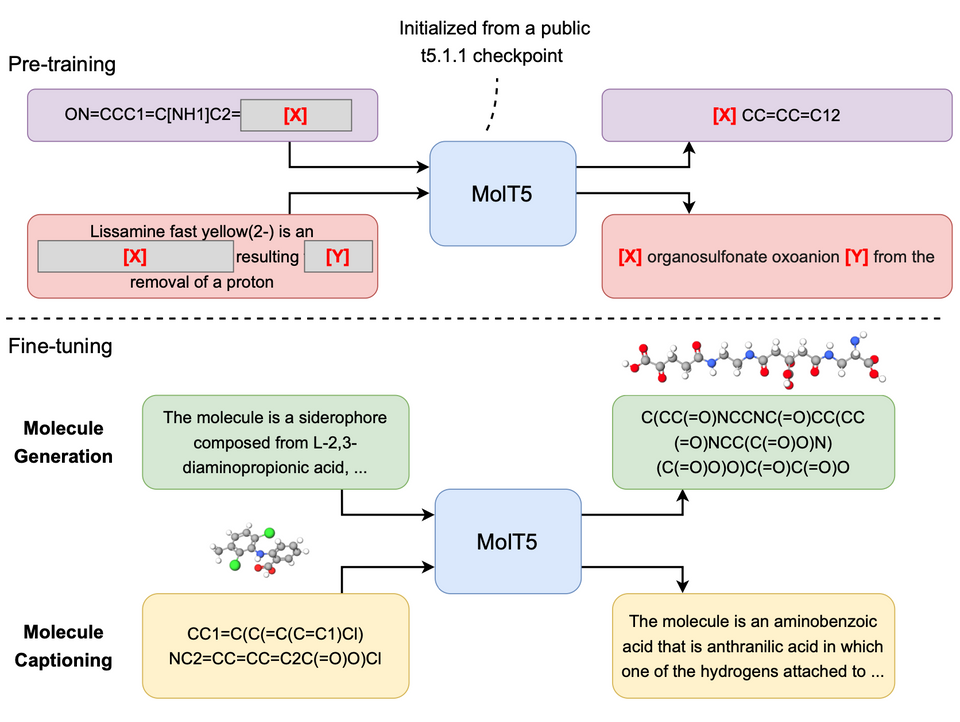
▲ 图6:MolT5 的模型架构
MolT5 是一个基于 Transformer 的模型,使用了 T5 的预训练架构。MolT5 的输入是一个分子 SMILES 字符串和一个自然语言文本,输出是一个分子字符串或一个自然语言文本,具体取决于所执行的任务。在分子字幕任务中,模型的输入是一个分子字符串,输出是一个自然语言文本,描述该分子及其功能。在基于文本的 de novo 分子生成任务中,模型的输入是一个自然语言文本,输出是一个新的分子字符串,该分子具有与文本描述相对应的功能。
作者在 CheBI-20 数据集上进行了实验,并使用了 Text2Mol 和 BLEU 两个指标来评估模型的性能。实验结果表明,MolT5 在两个任务上都取得了很好的性能,特别是在使用更大的模型时,性能得到了进一步提高。作者还进行了一些分析,以探索模型的行为和性能,例如,他们发现模型在生成分子时更喜欢使用较短的分子字符串,而在生成文本时更喜欢使用较长的文本。
07
/ /
论文标题:
Unifying Molecular and Textual Representations via Multi-task Language Modelling
论文地址:
https://arxiv.org/pdf/2301.12586.pdf
项目链接:
https://github.com/GT4SD/gt4sd-core
本文旨在将自然语言和化学语言的表示相结合,以便更好地处理化学数据和文本数据。为此,作者提出了一种跨领域、多任务的语言模型(Text+Chem T5),它可以有效地在自然语言和化学语言之间进行翻译,从而解决化学反应预测、反合成、文本生成和分子字幕等多种任务。
该模型的架构是基于 Transformer 的神经网络模型。它是一种跨领域、多任务的语言模型,可以在自然语言和化学语言之间进行翻译。作者提出了一种有效的训练策略,该策略利用单领域和多领域任务的优势,以适应单领域模型的跨领域任务。这消除了在大型单领域数据集上昂贵的预训练和任务特定的微调的需要,同时通过在任务和领域之间共享信息来提高跨领域翻译的性能。作者在基准数据集上进行了实验验证,证明了该模型在单领域和跨领域任务上与专门针对单个任务的最先进方法相当。
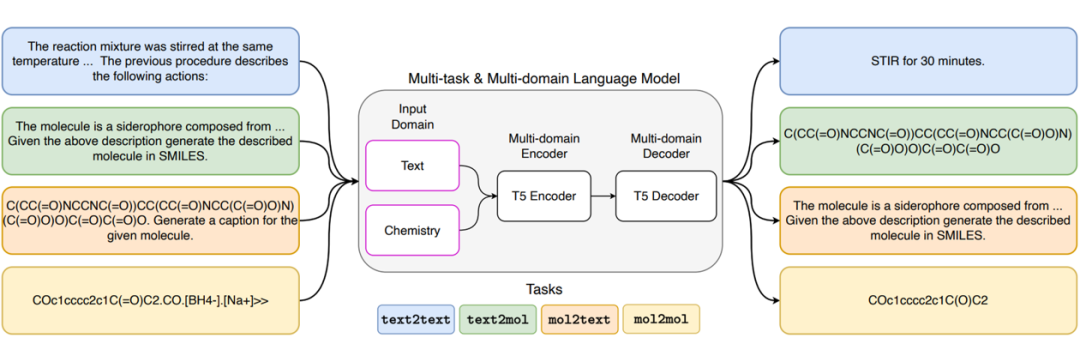
▲ 图7:Text+Chem T5 的模型架构。
作者在基准数据集上进行了实验验证,证明了该模型在单领域和跨领域任务上与专门针对单个任务的最先进方法相当。实验结果表明,跨领域任务的性能可以通过在单领域和多领域任务之间共享信息来提高。此外,作者还发现,使用多任务学习可以提高模型的性能,因为它可以利用不同任务之间的相似性和差异性。
08
/ /
论文标题:
Mol-Instructions: A Large-Scale Biomolecular Instruction Dataset for Large Language Models
论文地址:
https://arxiv.org/pdf/2306.08018.pdf
项目链接:
https://github.com/zjunlp/Mol-Instructions
本文提出一个全面的指令数据集 Mol-Instructions,旨在增强大型语言模型在生物分子领域的理解和预测能力。该数据集由分子导向指令、蛋白质导向指令和生物分子文本指令三个组成部分组成。
Mol-Instructions 的构建过程主要包括四个步骤:
人工智能与人类协作任务描述的创建:为了模拟人类提问的多样性,首先提供了明确简洁的人类编写的任务描述,并将其输入到gpt-3.5-turbo模型中生成多样的任务描述,以模仿各种不同的人类问题提出方式。
从现有数据中获取信息:生物分子数据往往需要专家实验室实验和专家分析,作者选用权威和公认的生物化学数据库作为 Mol-Instructions 数据的理想来源。通过适当的处理,作者将所收集到的原始数据提取并转化为所需的指令数据。
通过模板将生物数据转化为文本格式:为了将结构化的生物数据转化为文本格式,作者设计了一系列的模板。通过使用上述设计的模板,将从现有数据中获取的生物数据转化为文本格式的指令。
质量控制:由于生物分子语言的语法对于大语言模型来说相对陌生,作者采取了措施来加快模型对生物分子语言的适应和理解。这些质量控制措施包括对生物分子数据的严格筛选和鉴别,以确保数据的准确性和可信度。
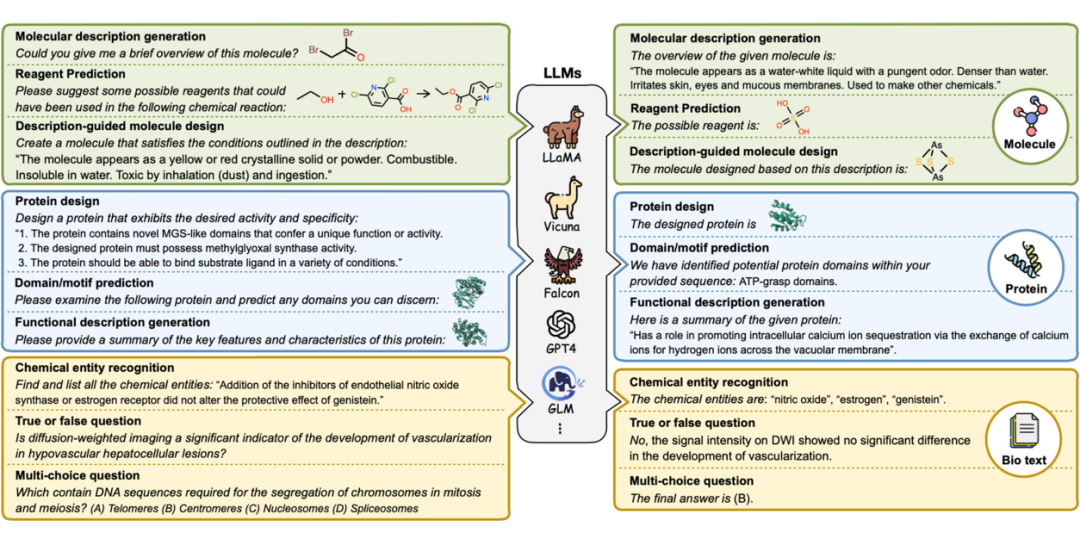
▲ 图8:Mol-Instructions 数据集能够帮助大型语言模型解锁生物分子领域。
通过大量的指令微调实验,该数据集被证明可以提高 LLMs 在复杂的生物分子研究领域的适应性和认知能力,以推动生物分子研究社区的进步。
09
/ /
论文标题:
DrugChat: Towards Enabling ChatGPT-Like Capabilities on Drug Molecule Graphs
论文地址:
https://www.techrxiv.org/articles/preprint/DrugChat_Towards_Enabling_ChatGPT-Like_Capabilities_on_Drug_Molecule_Graphs/22945922
项目链接:
https://github.com/UCSD-AI4H/drugchat
这篇文章旨在开发一个能够在药物分子图上实现 ChatGPT 类似功能的系统,以便回答关于药物的问题和生成文本描述。所提出的 DrugChat 模型利用图神经网络(GNN)对药物的分子图结构进行编码。GNN 捕捉图中原子和键之间的关系,并生成节点和边的嵌入表示。这些嵌入表示经过线性投影层,得到图结构特征表示。此外,DrugChat 还结合了 LLM,用于生成关于药物的文本描述和回答问题。采用与 Mini-GPT4 类似的架构,LLM 将图结构特征作为输入,根据提供的药物结构生成描述性文本。
在训练过程中,DrugChat 模型使用来自 ChEMBL 和 PubChem 数据集的数据。这些数据包括问题-答案对和药物特征,以指导模型生成准确的回答和描述。
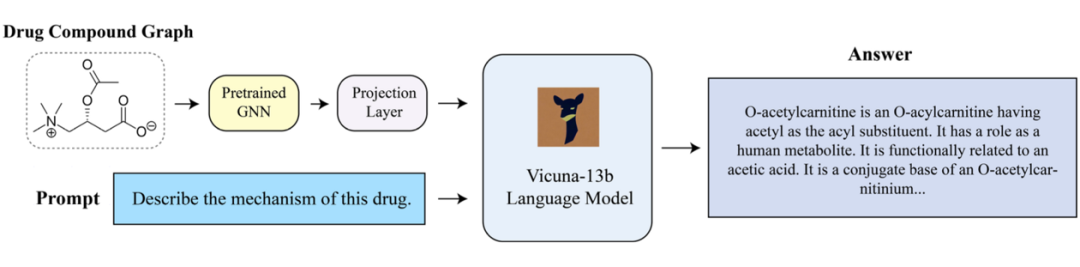
▲ 图9:DrugChat 的模型架构
实验结果显示,DrugChat 能够回答关于化合物的多样化多轮问题,并提供有信息量的答案。它可以生成关于药物作用机制、独特特征和潜在治疗应用的文本描述和回答问题。对 DrugChat 的系统定量评估仍在进行中,作者将与制药科学家合作进一步评估其性能和能力。

参考文献

[1] Flam-Shepherd D, Zhu K, Aspuru-Guzik A. Language models can learn complex molecular distributions[J]. Nature Communications, 2022, 13(1): 3293.
[2] Irwin R, Dimitriadis S, He J, et al. Chemformer: a pre-trained transformer for computational chemistry[J]. Machine Learning: Science and Technology, 2022, 3(1): 015022.
[3] Bagal V, Aggarwal R, Vinod P K, et al. MolGPT: molecular generation using a transformer-decoder model[J]. Journal of Chemical Information and Modeling, 2021, 62(9): 2064-2076.
[4] Fang Y, Zhang N, Chen Z, et al. Domain-AgnosticMolecular Generation with Self-feedback. arXiv preprint arXiv:2301.11259, 2023.
[5] Zeng Z, Yao Y, Liu Z, et al. A deep-learning system bridging molecule structure and biomedical text with comprehension comparable to human professionals[J]. Nature communications, 2022, 13(1): 862.
[6] Edwards C, Lai T, Ros K, et al. Translation between molecules and natural language[J]. EMNLP, 2022.
[7] Christofidellis D, Giannone G, Born J, et al. Unifying molecular and textual representations via multi-task language modelling[J]. ICML, 2023.
[8] Fang Y, Liang X, Zhang N, et al. Mol-Instructions: A Large-Scale Biomolecular Instruction Dataset for Large Language Models[J]. arXiv preprint arXiv:2306.08018, 2023.
[9] Liang Y, Zhang R, Zhang L, et al. DrugChat: Towards Enabling ChatGPT-Like Capabilities on Drug Molecule Graphs[J]. 2023.
更多阅读




#投 稿 通 道#
让你的文字被更多人看到
如何才能让更多的优质内容以更短路径到达读者群体,缩短读者寻找优质内容的成本呢?答案就是:你不认识的人。
总有一些你不认识的人,知道你想知道的东西。PaperWeekly 或许可以成为一座桥梁,促使不同背景、不同方向的学者和学术灵感相互碰撞,迸发出更多的可能性。
PaperWeekly 鼓励高校实验室或个人,在我们的平台上分享各类优质内容,可以是最新论文解读,也可以是学术热点剖析、科研心得或竞赛经验讲解等。我们的目的只有一个,让知识真正流动起来。
📝 稿件基本要求:
• 文章确系个人原创作品,未曾在公开渠道发表,如为其他平台已发表或待发表的文章,请明确标注
• 稿件建议以 markdown 格式撰写,文中配图以附件形式发送,要求图片清晰,无版权问题
• PaperWeekly 尊重原作者署名权,并将为每篇被采纳的原创首发稿件,提供业内具有竞争力稿酬,具体依据文章阅读量和文章质量阶梯制结算
📬 投稿通道:
• 投稿邮箱:hr@paperweekly.site
• 来稿请备注即时联系方式(微信),以便我们在稿件选用的第一时间联系作者
• 您也可以直接添加小编微信(pwbot02)快速投稿,备注:姓名-投稿

△长按添加PaperWeekly小编
🔍
现在,在「知乎」也能找到我们了
进入知乎首页搜索「PaperWeekly」
点击「关注」订阅我们的专栏吧
·
·
·