中文医疗文本处理大模型 PCL-MedBERT
- 提出背景
- 对ELECTRA限制的深入分析
- eHealth的创新方法
- 实体识别
- 关系抽取
- 总结
最近再做医学项目,需要从文本中抽取医学概念和关系,通用模型的抽取效果还可以。
但还想找医学嵌入模型,能够更准确地从文本中识别出医学实体(如病症、药物、治疗方法等)并理解实体间的关系(如疾病与症状之间的关系)。
提出背景
论文:https://arxiv.org/pdf/2110.07244v1.pdf
代码:https://github.com/trueto/medbert
本文介绍了eHealth,一个新的中文生物医学预训练语言模型,旨在解决中文生物医学领域缺乏高质量、公开可用的预训练模型的问题。
通过采用一个创新的自监督学习框架,该模型实现了在令牌级别和序列级别对生物医学文本的深入理解。
该框架包括一个用于生成损坏输入的生成器和一个通过多层次文本鉴别进行训练的鉴别器,使得eHealth能够在不依赖外部知识的情况下,仅通过文本本身学习语言语义。
这个框架有两个主要部分:一个生成器和一个鉴别器。
生成器的工作就像是在一个完整的句子中故意搞乱一些词语,而鉴别器的任务就是找出这些被搞乱的词语,并尝试修正它们。
- 生成器:它创建测试题目,通过对原始文本中的词语或句子进行随机修改,生成“损坏”的输入。这就像是给文本加上谜题,让模型去解决。
- 鉴别器:它尝试解决这个谜题,通过识别出哪些部分是被搞乱的,并且尽可能找出正确的原始形式。在这个过程中,鉴别器通过两个层面进行学习:一是找出单个词语(令牌级别)哪里不对劲,二是整个句子(序列级别)的结构是否有问题。
通过这种方式,eHealth模型可以更深入地理解文本的含义,因为它不仅学会了识别单个词语的正确与否,还学会了理解整个句子或文本段落的大致意图和结构,而且它不需要任何额外的信息或知识库,完全依赖于它所分析的文本本身来学习。
这种方法让eHealth能够有效地理解并处理生物医学领域的中文文本。
在11项中文生物医学语言理解任务上的测试结果显示,eHealth一致性地超越了同等规模的现有模型,包括那些专门针对生物医学领域或通用领域的模型。
在6.5亿字符中文临床自然语言文本语料上基于BERT模型,预训练获得了MedBERT模型。
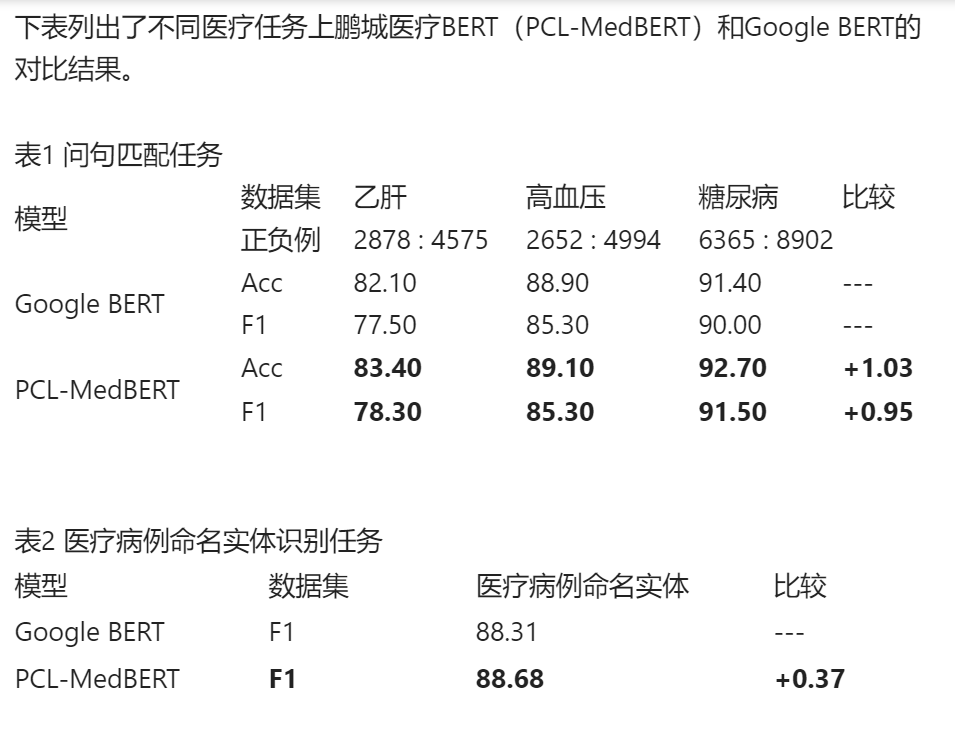
本工作的主要贡献在于提出了一种新的预训练算法,并验证了其在生物医学领域内的有效性,同时展示了该算法的通用性,为将来将此类方法扩展到其他领域提供了可能。
近年来,预训练语言模型(PLMs)已在自然语言处理(NLP)领域取得显著成功。这些模型,如BERT和其变种,通过在大规模的未标注文本上进行自监督学习来训练,学会了理解和生成语言。
这些文本主要来自一般领域,比如Wikipedia、新闻稿和网页内容。通过这种方法,PLMs能够捕获广泛的语言特征和模式,为各种NLP任务提供强大的基础。
尽管PLMs在一般领域取得了巨大成功,但在生物医学等专业领域内,直接应用这些一般领域的PLMs并不总是有效的。
这是因为专业领域具有独特的词汇、术语和语言结构,这些在一般领域的文本中很少或根本没有出现。
因此,为了在这些专业领域内达到最佳性能,研究人员开始构建专门的PLMs。然而,面临着两个主要挑战:
-
效率低下: 传统的掩码语言模型(MLM)方法在训练时只能从每次输入中的15%被掩码的令牌中学习,导致高计算成本。
-
缺乏专业领域的PLMs:尤其是中文生物医学领域,缺乏高质量、公开可用的PLMs,部分现有模型需要依赖额外、通常不公开的专业知识,限制了其应用范围。
为了解决上述挑战,研究人员提出了新的方法和技术:
- ELECTRA:通过一个新颖的预训练框架,ELECTRA引入了替换令牌检测(RTD)任务,其中一些令牌被随机替换,模型必须预测每个令牌是否被替换。这使得模型能够从所有输入令牌中学习,而不仅仅是被掩码的部分,显著提高了训练效率。
- 多令牌选择(MTS)和序列级信号:为了让模型捕获更丰富的语言信息和更好地理解语言结构,研究人员提出使用MTS和结合序列级信号的方法。MTS是RTD的泛化,要求模型从可能的候选中选择原始令牌,而序列级信号(如下一句预测等)则帮助模型理解文本之间更复杂的关系。
在ELECTRA框架中,生成器和鉴别器通过一种对抗的方式合作:生成器尝试“欺骗”鉴别器,而鉴别器则努力识别生成器的“欺骗”。
这种机制促进了模型在令牌级别上的学习,而多令牌选择(MTS)和序列级信号的引入,则进一步扩展了模型的学习范围,包括对复杂选择的处理和对整个文本结构的理解。
-
多令牌选择(MTS):这是一种训练任务,要求模型从一系列可能的候选令牌中选择最合适的令牌来恢复原始文本。
在ELECTRA框架中,这种方法可以被视为对鉴别器任务的扩展或改进,提供了更复杂的决策过程,因为模型不仅要识别哪些令牌被替换,还要从多个候选中选择正确的令牌。
-
序列级信号:这类信号涉及到对整个句子或文本段落的理解,而不仅是单个令牌。通过训练模型识别句子之间的逻辑关系、预测句子顺序或进行句子级别的对比学习,可以增强模型对文本整体结构的理解。
在ELECTRA框架中,虽然主要关注的是令牌级别的判断(由鉴别器完成),但通过引入序列级信号,可以进一步提高模型对语言结构的理解,尤其是在理解长文本或复杂文本结构时。
eHealth模型的开发是基于对ELECTRA存在限制的深入分析,并通过创新的方法来克服这些限制,以更好地服务于中文生物医学文本的处理。
对ELECTRA限制的深入分析
-
令牌级别的二元分类限制: ELECTRA模型通过生成器产生的损坏输入来训练鉴别器,仅在令牌级别进行二元分类(即,判断令牌是否被正确地替换)。
这种方法虽然有效,但主要局限于识别单个令牌的正确性,可能不足以捕捉到更复杂的语言模式,特别是在专业领域中,文本的含义往往需要更深层次的语义理解。
-
缺乏序列级别的学习: ELECTRA主要关注单个令牌的处理,而不涉及更广泛的文本结构或序列间的关系。
这限制了模型对于长篇文本或具有复杂结构和关系的文本的理解能力,而这在生物医学文本中尤为重要。
eHealth的创新方法
-
**丰富的令牌级别鉴别:**为了克服仅有的二元分类的限制,eHealth在令牌级别引入了更为丰富的鉴别任务。
不仅判断令牌是否被替换,而且还要恢复被替换令牌的原始身份。这种方法能够提供更多的语言信息给模型,帮助模型更细致地理解语言细节和文本的语义层面。
-
序列级别鉴别的引入: eHealth通过引入序列级别的鉴别任务来加强模型对文本整体结构的理解。
这种方式使得模型不仅能理解单个词语,还能把握整个句子或段落中词语之间的关系,以及它们如何共同表达特定的意义或概念。
实体识别
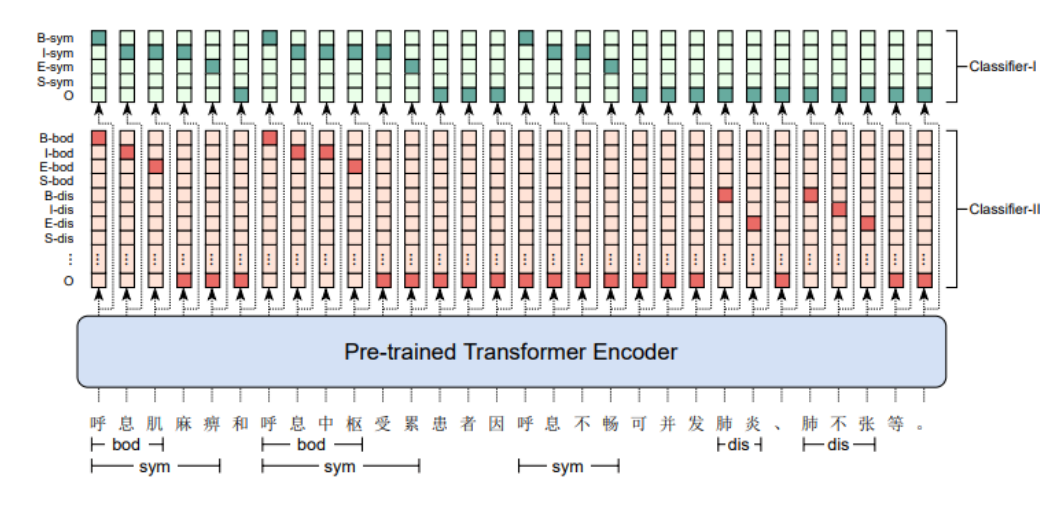
图展示的是用于中文医学实体识别(CMEE)任务的序列标注示例。在该任务中,目标是从医学文本中识别并标注特定的实体,如症状、疾病等。
深色阴影的条目代表实际标签为1(表示该令牌是一个实体的一部分),而浅色阴影的条目代表实体标签为0(表示该令牌不是实体的一部分)。
序列标注模型通常使用BIO标注体系,其中"B"代表实体的开始,"I"代表实体内部的令牌,"O"代表非实体令牌。
如图所示,可能还会有其他前缀,比如"S"和"E",分别代表单独的实体和实体的结束。
这些标签与预训练的Transformer编码器的输出相连接,编码器输出每个令牌的上下文表示,然后通过分类器来预测每个令牌的最终标签。
关系抽取
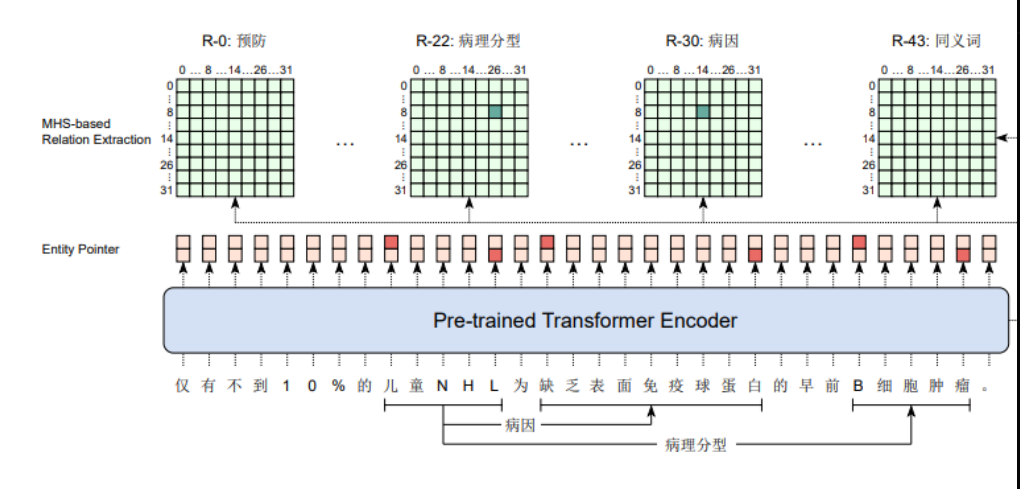
图展示的是一个多头选择层,用于中文医学实体和关系提取(CMIE)的联合任务。
在这个任务中,模型不仅要识别文本中的实体,还要识别实体之间的关系。
深色阴影的条目表示真实标签为1(表示选择的头部和关系是正确的),而浅色阴影的条目表示真实标签为0(表示选择不正确或无关)。
实体指针和关系提取的头部基于预训练的Transformer编码器的输出。
这个多头选择层可能是用来同时捕获实体之间的多种关系。
每个关系类型都有一个头部(比如R0、R22、R30),模型会预测文本中的实体对是否属于该关系类型。
总结
-
提出背景与动机
- 预训练语言模型(PLMs)在自然语言处理(NLP)领域取得了显著的成功,特别是BERT及其变体通过在大规模未标注文本上的自监督学习,显著推动了NLP的发展。
- 尽管在通用领域PLMs取得了巨大成功,但直接将这些模型应用于生物医学等专业领域的效果并不总是理想的,因为专业领域具有独特的词汇、术语和语言结构,需要专门的预训练模型来处理。
-
ELECTRA模型的限制
- ELECTRA模型采用令牌级别的二元分类任务进行预训练,虽有效,但可能不足以捕捉更复杂的语言模式。
- ELECTRA缺乏在序列级别进行学习的能力,限制了模型对复杂文本结构的理解。
-
eHealth模型的创新
- eHealth模型采用了ELECTRA的生成器-鉴别器框架,并通过引入丰富的令牌级别鉴别和序列级别鉴别的创新方法,旨在克服ELECTRA在生物医学文本处理上的限制。
- eHealth能够在不依赖外部知识的情况下,仅通过文本本身学习语言语义,有效地理解并处理生物医学领域的中文文本。
-
eHealth模型的实验验证
- 在11项中文生物医学语言理解任务上的测试结果显示,eHealth一致性地超越了同等规模的现有模型,证明了其有效性和优越性。
eHealth模型的开发基于对ELECTRA存在限制的深入分析,并通过创新的方法来提升中文生物医学文本的处理能力,展现了预训练算法在生物医学领域内的有效性和通用性。