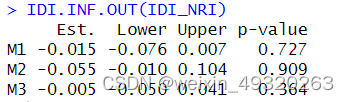
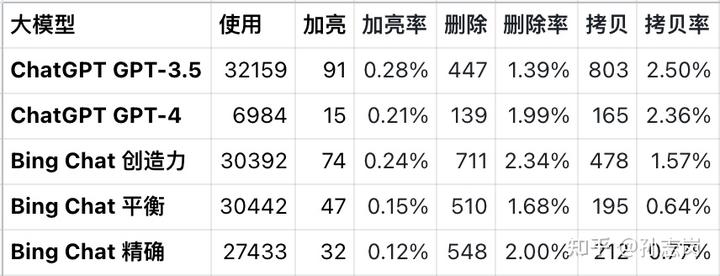
两个模型比较,与第一个模型相比,NRI=(重新分对的 - 重新分错的)/总人数。IDI=(新模型患者平均预测概率-旧模型患者平均预测概率)-(新模型非患者平均预测概率-旧模型非患者平均预测概率)。NRI=0无改善,>0新模型优于旧模型。IDI正值且越大越好。
1、非生存资料:广义线性模型
library(PredictABEL)
#数据集
data1<-data.frame(a=rnorm(n=1000,mean=6,sd=5),b=rnorm(1000,30,9),c=rnorm(1000,17,8),cOutcome=sample(c(0,1),1000,replace=T))
#建模
model_1<-glm(cOutcome~.,data1,family="binomial")
model_2<-glm(cOutcome~a+c,data1,family="binomial")
#计算NRI和IDI
pre1<-predRisk(model_1)
pre2<-predRisk(model_2)#=model_2$fitted.values
reclassification(data = data1, cOutcome = 4, predrisk1 = pre1, predrisk2 = pre2, cutoff = c(0,0.6,1))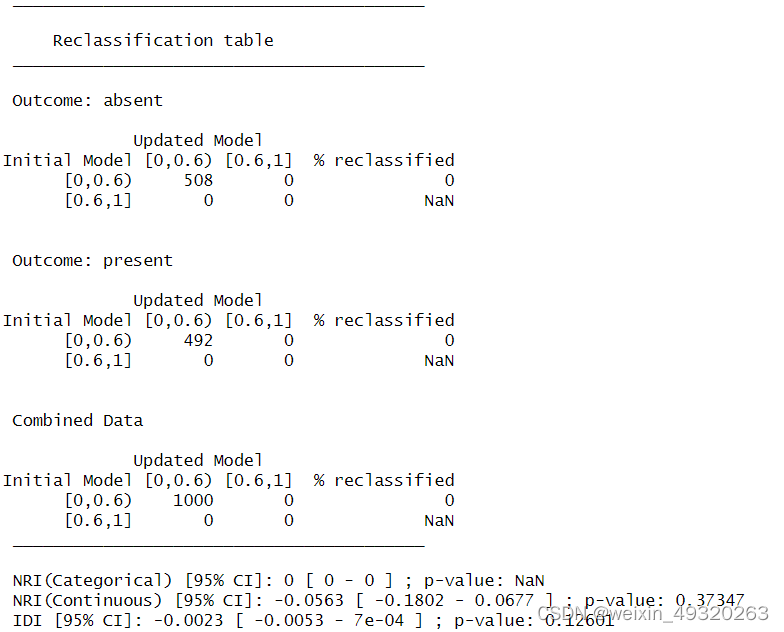
NRI<0,第二个模型较第一个模型差,降低了0.23%(IDI),但是均为统计学意义。
2、生存资料
#生存资料:CoIDI_NRI回归
library(survival)
data(cancer)
colnames(lung)
lung<-na.omit(lung)
coIDI_NRI1<- coIDI_NRIph(Surv(time, status) ~age+ph.ecog,data=lung,IDI_NRI=T)
coIDI_NRI2 <- coIDI_NRIph(Surv(time, status) ~.,data=lung,IDI_NRI=T)
#install.packages("nricens")
#1 利用nricens包
library(nricens)
nricens(mdl.std = coIDI_NRI1, mdl.new = coIDI_NRI2, t0 = 100,cut = c(0.2, 0.4),niter = 10)
#2 利用survIDINRI包
#install.packages("survIDINRI")
library(survIDINRI)
help(package="survIDINRI")
data_2 <- lung[,c("time","status")]
covs0 <- lung[,c("age","sex")]
covs1 <- lung[,c("age","sex","ph.ecog","ph.karno")]
IDI_NRI<-IDI.INF(data_2,covs0 = as.matrix(covs0),covs1=as.matrix(covs1), t0=500, npert=10)
IDI.INF.OUT(IDI_NRI)