基于AlphaFold2进行蛋白质结构预测的文章解析
RoseTTAFold: Tunyasuvunakool, K., Adler, J., Wu, Z. et al. Highly accurate protein structure prediction for the human proteome. Nature 596, 590–596 (2021)
AlphaFold2: Accurate prediction of protein structures and interactions using a three-track neural network
上海交大超算平台用户手册 Documentation
AF2方法总结
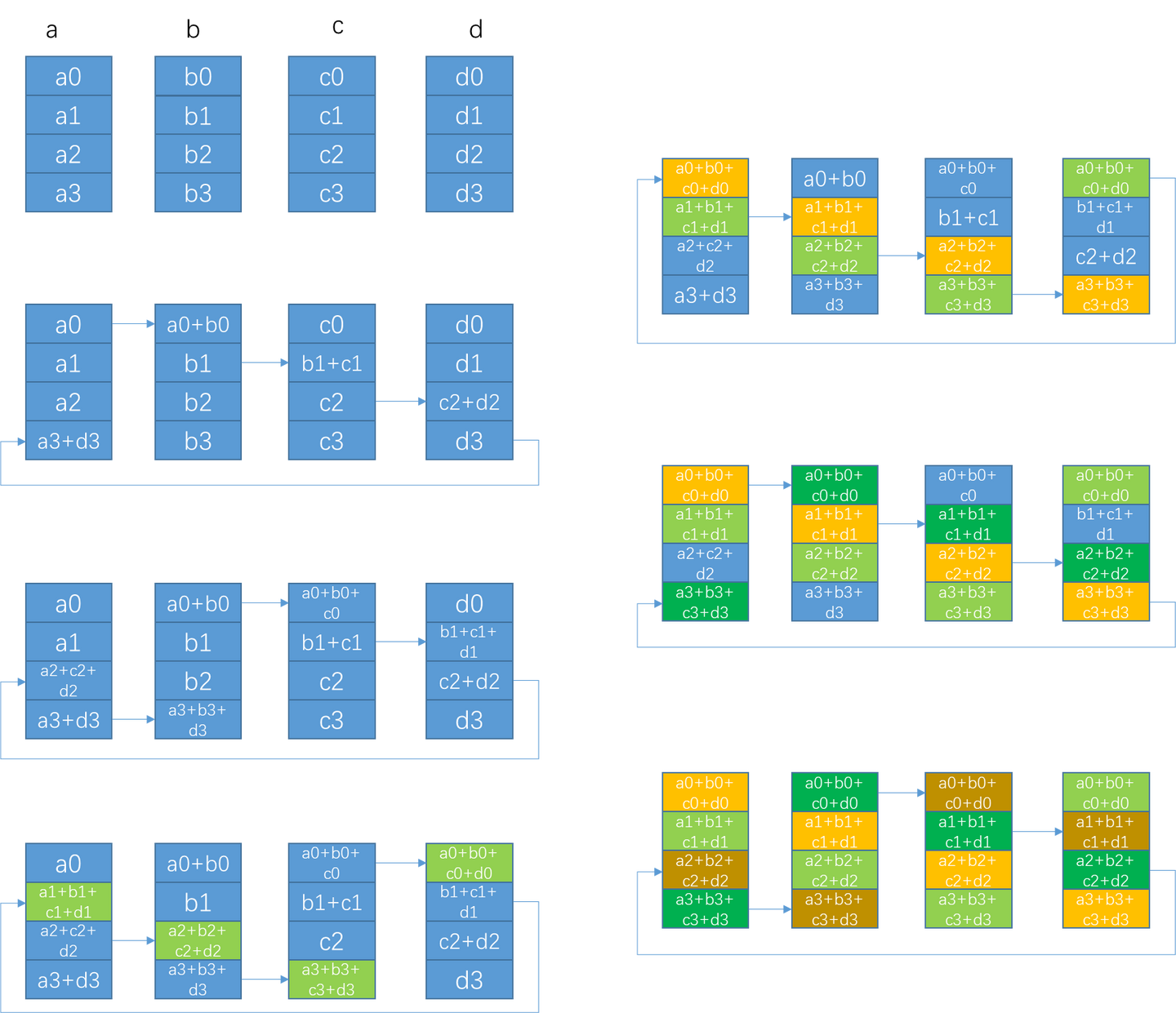
展示了一种联合嵌入多序列比对 (MSA) 和成对特征的输出和损失估计新架构,可实现准确的端到端结构预测 训练神经网络来对regression target进行逐步迭代精化(Iterative refinement) 广泛运用了Attention架构。一个二维的表可以横着做再竖着做attention,一个图可以在各种局部结构上做attention,从而不断精化embedding的结构。
还用到了Noisy student self-distillation,用带标签数据(氨基酸序列与三维坐标的对应)先训练一遍网络,然后用训练完的网络在无标签数据(仅有氨基酸序列)上预测一遍生成新的数据集,只保留预测得好的部分,然后把这两者混合拿来再进行训练 类似BERT的masking操作,对各种输入信息加噪音要求输出稳定,提高了鲁棒性和泛化能力。

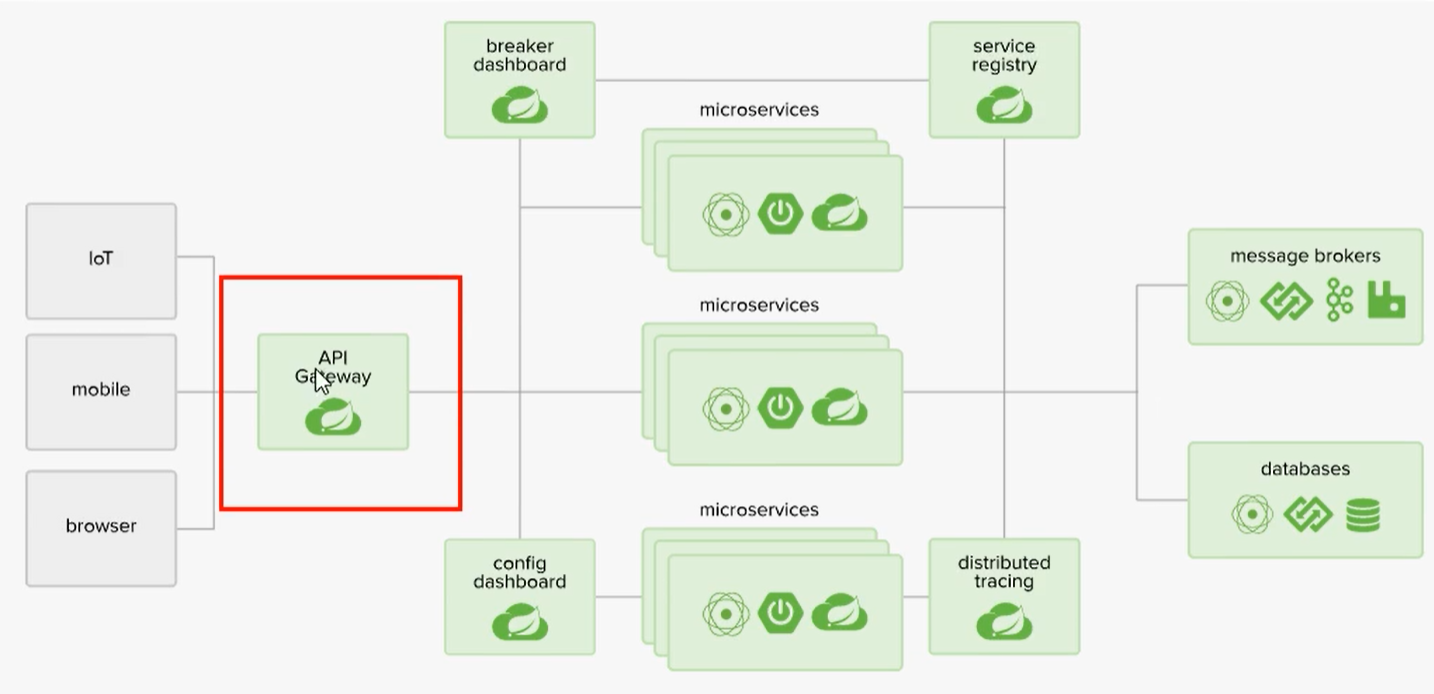
![[人工智能-综述-15]:第九届全球软件大会(南京)有感 -4-大语言模型全流程、全方面提升软件生产效能](https://img-blog.csdnimg.cn/43e3de8f115c4ccba6dccb91391ccee9.png)




![【LeetCode】145. 二叉树的后序遍历 [ 左子树 右子树 根结点]](https://img-blog.csdnimg.cn/88587c23768a40c8be2df9f79e6b8235.png)
![[SQL | MyBatis] MyBatis 简介](https://img-blog.csdnimg.cn/b8551321cd504535bbc61a2e3f5c4234.png)