现在的单细胞分析,往往避免不了scanpy的使用,我们可以通过对比seurat来学习scanpy

今天的格式怎么都改不了。。。手机阅读有点费劲,,推荐电脑阅读。
单细胞数据分析概览
单细胞分析,总流程

python教程

seurat教程

seurat中与scanpy对等的函数操作
数据预处理Seurat (R)CreateSeuratObject(): 创建Seurat对象。 NormalizeData(): 数据标准化。 FindVariableFeatures(): 识别高变异基因。 ScaleData(): 数据缩放和中心化。 Scanpy (Python)sc.read() / sc.read_10x_mtx(): 读取数据创建AnnData对象。 sc.pp.normalize_total(): 数据标准化。 sc.pp.highly_variable_genes(): 识别高变异基因。 sc.pp.scale(): 数据缩放和中心化。 降维分析Seurat (R)RunPCA(): 主成分分析(PCA)。 RunUMAP(): UMAP降维。 RunTSNE(): t-SNE降维。 Scanpy (Python)sc.tl.pca(): 主成分分析(PCA)。 sc.tl.umap(): UMAP降维。 sc.tl.tsne(): t-SNE降维。 聚类分析Seurat (R)FindNeighbors(): 计算邻居图。 FindClusters(): 基于图的聚类。 Scanpy (Python)sc.pp.neighbors(): 计算邻居图。 sc.tl.louvain() / sc.tl.leiden(): 基于图的聚类。 差异表达分析Seurat (R)FindMarkers(): 寻找差异表达基因。 Scanpy (Python)sc.tl.rank_genes_groups(): 寻找差异表达基因。 数据可视化Seurat (R)DimPlot(): 降维数据可视化。 FeaturePlot(): 基因表达水平可视化。 VlnPlot(): 小提琴图展示基因表达分布。 Scanpy (Python)sc.pl.umap() / sc.pl.tsne(): 降维数据可视化。 sc.pl.dotplot() / sc.pl.violin(): 基因表达水平可视化。 |
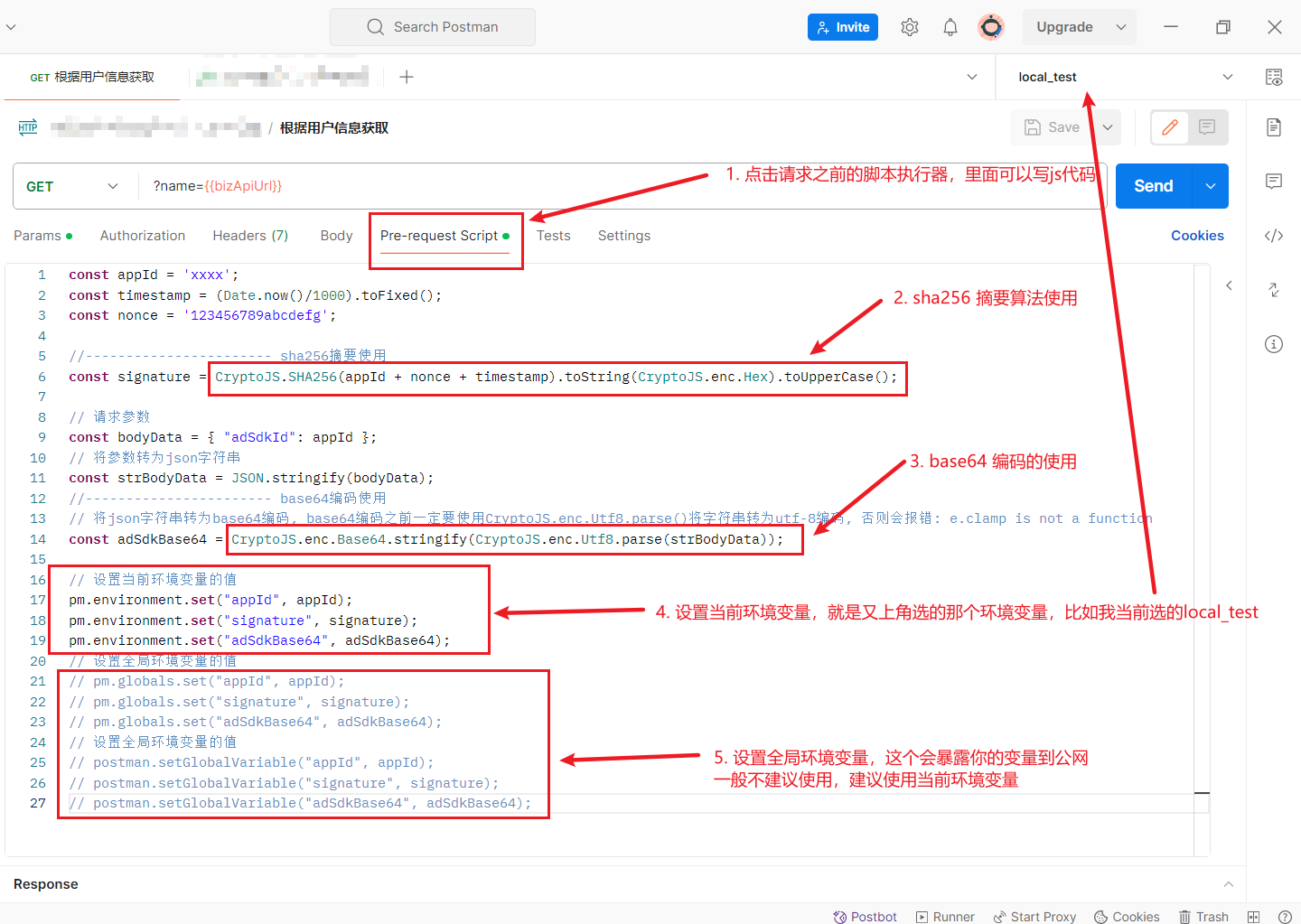
为何seurat中没有与scanpy中的sc.pp.log1p(adata)对应步骤
有几个方面考虑:
1. 数据标准化方法的差异Seurat和Scanpy在数据预处理和标准化方面采取了不同的方法。Seurat的NormalizeData函数默认使用的是LogNormalize方法,这个方法首先对每个细胞的基因表达量进行归一化处理,使得每个细胞的总表达量相同(默认是1e4),然后对归一化后的表达量加1后取对数(使用自然对数)。这个过程实质上包含了与Scanpy中sc.pp.normalize_total后跟sc.pp.log1p相似的步骤,只是Seurat将其整合在了一个步骤中进行。 2. 集成的处理流程Seurat的设计哲学是提供一个相对简化和集成的分析流程,使得用户可以通过少量的函数调用完成从数据预处理到高级分析的整个过程。因此,Seurat在其标准化函数中内置了对数变换,而无需用户手动进行这一步。这种设计选择简化了分析流程,减少了需要记住的函数数量,但也意味着用户在使用过程中可能对于数据处理的每一步不如Scanpy那样清晰明了。 3. 灵活性与透明度Scanpy采取的是更模块化的策略,每个处理步骤都由不同的函数完成。这种方法提供了更大的灵活性和透明度,使得用户能够更清楚地看到数据是如何被逐步处理和分析的。这对于需要定制处理流程的研究人员来说是一个优点。 |

![]()
看完记得顺手点个“在看”哦!