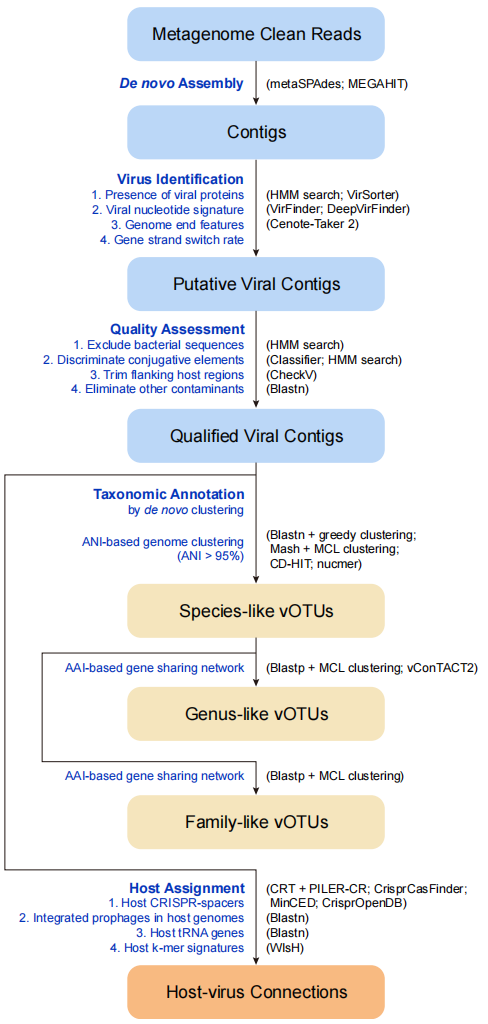
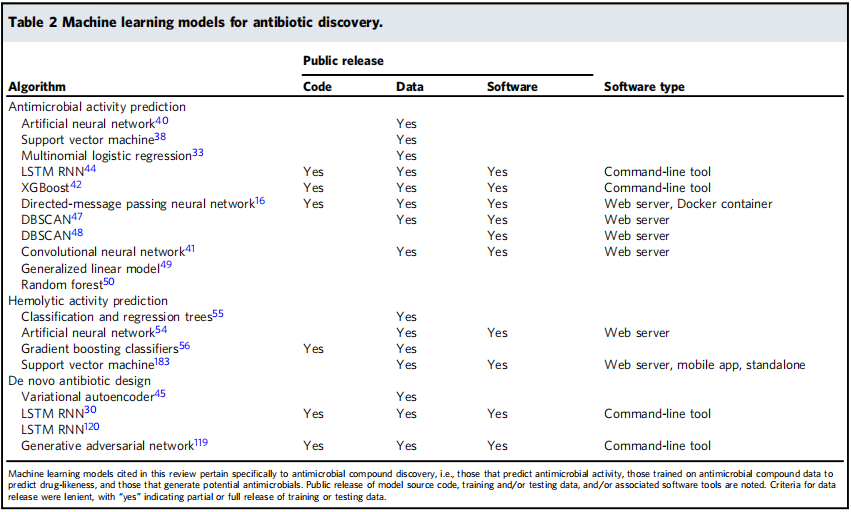
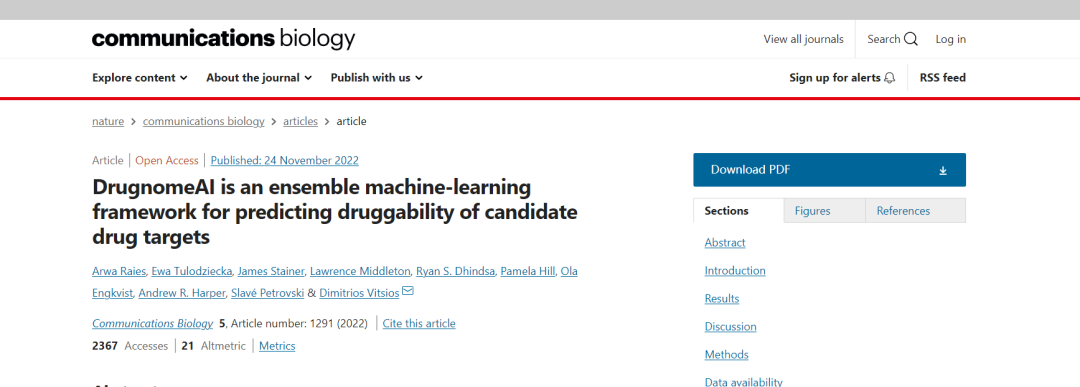
2022年11月,communication biology杂志上刊登了一篇来自英国剑桥大学的名为“DrugnomeAI is an ensemble machine-learning framework for predicting druggability of candidate drug targets”的研究论文。该文章针对靶点成药性预测问题,提出了一个名为DrugnomeAI的预测模型。
概要
研究靶点成药性是药物发现中不可或缺的一部分,其结果会影响到靶点识别,甚至能够左右临床开发的成功与否。本文中,我们将采用(stochastic semi-supervised ML framework)随机半监督机器学习框架来开发DrugnomeAI,用于评估蛋白质编码基因在人类外显子组中的成药性。DrugnomeAI还提供按疾病类型或药物治疗方式分类的通用和专用模型。此外,我们设计了一个web应用程序,以可视化的方式为感兴趣的读者展示我们的方法。
工作介绍
我们的工作扩展了现有的成药性预测方法,如整合了一套全面的成药性特征、提供疾病学和特定领域模型来突出治疗区域和药物形式的基因。具体来讲,我们演示了DrugnomeAI在预测肿瘤及非肿瘤疾病的基因成药性方面的应用。我们的药物模态独立模型预测了哪些基因具有小分子、单克隆抗体和蛋白水解靶向嵌合体(PROTACs)可调节特性,且据我们所知,我们的研究开发了第一个预测基于PROTAC治疗的基因成药性的ML模型。此外,研究人员可以使用DrugnomeAI框架,并利用用户定义的种子基因来训练生成特定的疾病模型。
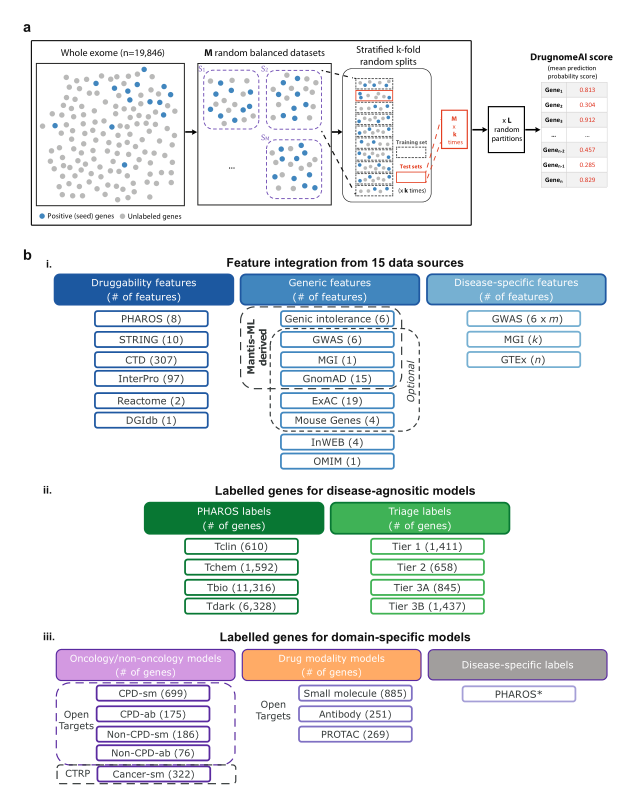
基因成药性分析。基于已知药物靶点的历史数据和关于基因成药性的其他类型数据(如上图a),我们推测了整个人类外显子组的基因成药性。所用的训练数据集来自Pharos库的Tclin和Tchem,以及Triage库的Tier1、Tier2和Tier 3 A。在DrugnomeAI训练期间,我们还测试了一系列不同的成药性和基因级别的特征集(如上图b)。
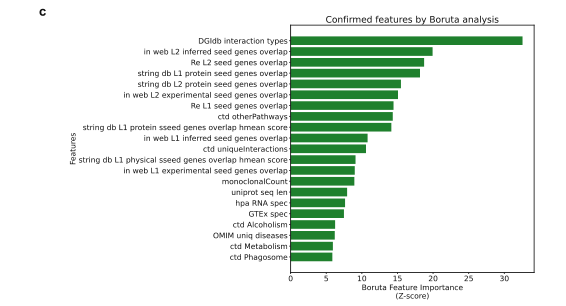
重要成药性特征的消融分析和Boruta分析。首选优选出“Pharos + InerPro”作为DrugnomeAI的默认特征集来消除分信息冗余,然后使用Boruta算法对Tclin(如上图c)和Tier1标记数据集进行特征重要性分析。
验证和探索DrugnomeAI的前列匹配数据。因使用Tclin或Tier1标签集训练的梯度增强模型可以达到最佳性能,于是我们使用这些预测结果作为进一步工作的参考。结果表示,DrugnomeAI-Tclin和DrugnomeAI-Tier1排序中位于前5%的基因有63%是重叠的。
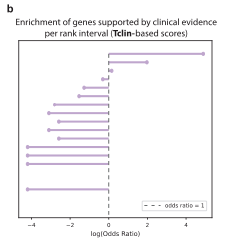
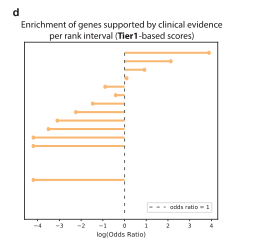
有临床依据的前列匹配数据。基于上面的两组数据,我们发现在临床选择中,DrugnomeAI-Tclin排序前5%的基因显著聚集(如上图b),DrugnomeAI-Tier1也有相似的情况(如上图d),且两者分别有76%和61%的基因被用于临床开发。同时我们也分析了其他无临床依据的基因。
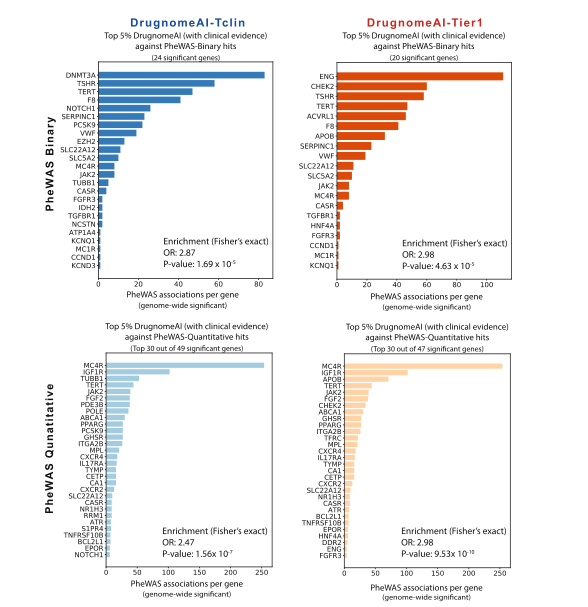
此外,我们还通过大规模表型关联研究(PheWAS)(如上图)、OMIM疾病注释来试验DrugnomeAI模型,以及与其他成药性预测方法进行对照,皆显示出了DrugnomeAI模型的优势。除了通用的DrugnomeAI模型,我们还可开发了针对三种药物模态(小分子、单克隆抗体以及PROTAC)的治疗模态独立模型,以及肿瘤及非肿瘤特异性DrugnomeAI模型和特定领域DrugnomeAI模型。
总结
药物靶点的选择是药物发现过程中非常关键的一步,其对后期临床试验的成功率有极大的影响。因此,我们团队围绕靶点的成药性进行深入研究,推出DrugnomeAI成药性预测模型。不过,DrugnomeAI和其他数据驱动方法都存在一个局限性——即倾向于忽略未研究的基因,这导致模型优先考虑先前已知且充分研究的药物靶点类似的基因,却不一定能识别作用于不同机制的新靶点。
同时,成药性不是基因固有的属性,靶点的成药性有可能取决于疾病和药物。因此,全面了解靶点的成药性、配体性、抑制性和激活性才能进一步帮助我们扩大对成药性基因组的认识,从而发现新的靶点。
参考资料
Raies, A., Tulodziecka, E., Stainer, J. et al. DrugnomeAI is an ensemble machine-learning framework for predicting druggability of candidate drug targets. Commun Biol 5, 1291 (2022).
https://doi.org/10.1038/s42003-022-04245-4