1、环境介绍
python3.6,tensorflow1.4版本,pycharm编译器
2、函数库导入
import cv2
import matplotlib.pyplot as plt
import os, PIL, pathlib
import numpy as np
from tensorflow.keras.layers import Conv2D,MaxPooling2D,Dropout,Dense,Flatten,Activation
import pandas as pd
from tensorflow.keras.models import Sequential
import warnings
from tensorflow import keras
import pathlib
from tensorflow.keras.layers import BatchNormalization
import tensorflow as tf
3、数据集获取
数据集链接,含有猫狗两类图像,图像尺寸不等,猫狗图片各1000张。

猫:

狗:
4、神经网络搭建:
def createModel(num_classes):model = Sequential() # 顺序模型model.add(Conv2D(16, (5, 5), strides=(2, 2), padding="same", input_shape=(256, 256, 3), data_format='channels_last',kernel_initializer='uniform', activation="relu"))model.add(MaxPooling2D(pool_size=(2, 2), strides=(2, 2)))model.add(Conv2D(32, (3, 3), strides=(2, 2), activation="relu"))model.add(MaxPooling2D(pool_size=(2, 2), strides=(2, 2)))model.add(Conv2D(64, (3, 3), activation="relu"))model.add(Conv2D(128, (3, 3), activation="relu"))model.add(MaxPooling2D(pool_size=(2, 2), strides=(2, 2), name="pool5"))model.add(Conv2D(256, (1, 1), activation="relu"))model.add(Dropout(0.25))model.add(Flatten())model.add(Dense(256))model.add(BatchNormalization())model.add(Activation('relu'))model.add(BatchNormalization())model.add(Dropout(0.5))model.add(Dense(num_classes))model.add(BatchNormalization())model.add(Activation("softmax"))model.summary()return model
神经网络结构如下:
Model: "sequential"
_________________________________________________________________
Layer (type) Output Shape Param #
=================================================================
conv2d (Conv2D) (None, 128, 128, 16) 1216
_________________________________________________________________
max_pooling2d (MaxPooling2D) (None, 64, 64, 16) 0
_________________________________________________________________
conv2d_1 (Conv2D) (None, 31, 31, 32) 4640
_________________________________________________________________
max_pooling2d_1 (MaxPooling2 (None, 15, 15, 32) 0
_________________________________________________________________
conv2d_2 (Conv2D) (None, 13, 13, 64) 18496
_________________________________________________________________
conv2d_3 (Conv2D) (None, 11, 11, 128) 73856
_________________________________________________________________
pool5 (MaxPooling2D) (None, 5, 5, 128) 0
_________________________________________________________________
conv2d_4 (Conv2D) (None, 5, 5, 256) 33024
_________________________________________________________________
dropout (Dropout) (None, 5, 5, 256) 0
_________________________________________________________________
flatten (Flatten) (None, 6400) 0
_________________________________________________________________
dense (Dense) (None, 256) 1638656
_________________________________________________________________
batch_normalization (BatchNo (None, 256) 1024
_________________________________________________________________
activation (Activation) (None, 256) 0
_________________________________________________________________
batch_normalization_1 (Batch (None, 256) 1024
_________________________________________________________________
dropout_1 (Dropout) (None, 256) 0
_________________________________________________________________
dense_1 (Dense) (None, 2) 514
_________________________________________________________________
batch_normalization_2 (Batch (None, 2) 8
_________________________________________________________________
activation_1 (Activation) (None, 2) 0
=================================================================
Total params: 1,772,458
Trainable params: 1,771,430
Non-trainable params: 1,028
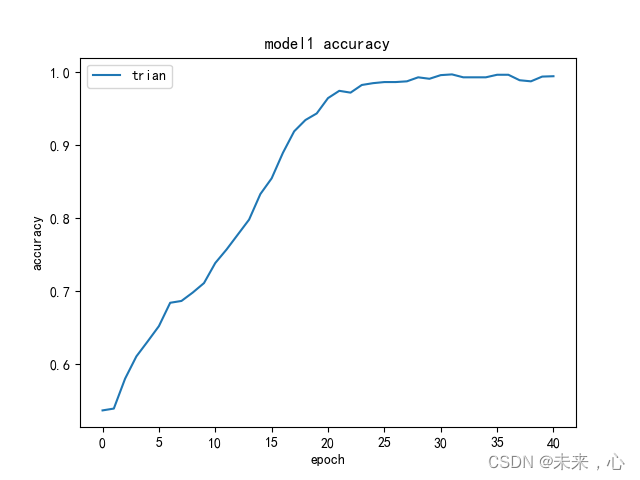
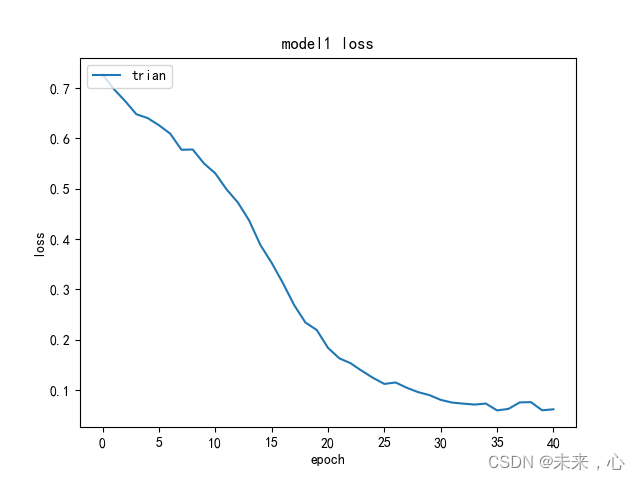
5、训练结果展示:
print(history.history.keys())
plt.plot(history.history['accuracy'])
plt.plot(history.history['val_accuracy'])
plt.title('model1 accuracy')
plt.ylabel('accuracy')
plt.xlabel('epoch')
plt.legend(['trian','test'],loc='upper left')
plt.show()plt.plot(history.history['loss'])
plt.plot(history.history['val_loss'])
plt.title('model1 loss')
plt.ylabel('loss')
plt.xlabel('epoch')
plt.legend(['trian','test'],loc='upper left')
plt.show()
6、完整代码:
import cv2
import matplotlib.pyplot as plt
import os, PIL, pathlib
import numpy as np
from tensorflow.keras.layers import Conv2D,MaxPooling2D,Dropout,Dense,Flatten,Activation
import pandas as pd
from tensorflow.keras.models import Sequential
import warnings
from tensorflow import keras
import pathlib
from tensorflow.keras.layers import BatchNormalization
import tensorflow as tf
os.environ["CUDA_VISIBLE_DEVICES"] = "0"
warnings.filterwarnings("ignore") # 忽略警告信息
plt.rcParams['font.sans-serif'] = ['SimHei'] # 用来正常显示中文标签
plt.rcParams['axes.unicode_minus'] = False # 用来正常显示负号
# 数据集路径
trainpath='.\data\cats_and_dogs_v2\\train'
testpath='.\data\cats_and_dogs_v2\\test'
trainpath1 = pathlib.Path(trainpath)
# testpath1 = pathlib.Path(testpath)
m= len(list(trainpath1.glob('*/*')))
m2= len(list(trainpath1.glob('*/*')))
# 图片尺寸
batch_size = 16
# img_height = 50
# img_width = 50
# 二分类
num_classes=2
def changeDim(img):#扩展维度变4维img=np.expand_dims(img,axis=2)#return img
def Generator(path, batch_size):data = []label = []while True:file = os.listdir(path)i = 0img1 = []img2 = []imgname1 = os.listdir(path + '/' + file[0])imgname2 = os.listdir(path + '/' + file[1])# print(imgname1)for name1 in imgname1:img1.append(path + '/' + file[0] + '/' + name1)for name2 in imgname2:img2.append(path + '/' + file[1] + '/' + name2)imgname = img1 + img2 ######横向连接# print(imgname)# print((len(imgname)))np.random.shuffle(imgname)# 猫是0,狗是1for finame in imgname:im = cv2.imread(finame)im = cv2.resize(im,(256,256))label_a = finame.split('/')[-2]# print(label_a)if label_a=='cats':label_a=0else:label_a = 1# im = changeDim(im)# print(im.shape)data.append(im)# print(data.shape)label.append(label_a)if (len(label) == batch_size):data = np.array(data)data = data.astype('float32')data /= 255.0label = keras.utils.to_categorical(label, 2)yield data, labeldata = []label = []i += 1
def createModel(num_classes):model = Sequential() # 顺序模型model.add(Conv2D(16, (5, 5), strides=(2, 2), padding="same", input_shape=(256, 256, 3), data_format='channels_last',kernel_initializer='uniform', activation="relu"))model.add(MaxPooling2D(pool_size=(2, 2), strides=(2, 2)))model.add(Conv2D(32, (3, 3), strides=(2, 2), activation="relu"))model.add(MaxPooling2D(pool_size=(2, 2), strides=(2, 2)))model.add(Conv2D(64, (3, 3), activation="relu"))model.add(Conv2D(128, (3, 3), activation="relu"))model.add(MaxPooling2D(pool_size=(2, 2), strides=(2, 2), name="pool5"))model.add(Conv2D(256, (1, 1), activation="relu"))model.add(Dropout(0.25))model.add(Flatten())model.add(Dense(256))model.add(BatchNormalization())model.add(Activation('relu'))model.add(BatchNormalization())model.add(Dropout(0.5))model.add(Dense(num_classes))model.add(BatchNormalization())model.add(Activation("softmax"))model.summary()return model
model = createModel(num_classes)
model.compile(optimizer="adam",loss='binary_crossentropy',metrics=['accuracy'])
from tensorflow.keras.callbacks import ModelCheckpoint, Callback, EarlyStopping, ReduceLROnPlateau, LearningRateSchedulerNO_EPOCHS = 50
PATIENCE = 5
VERBOSE = 1# 设置动态学习率
annealer = LearningRateScheduler(lambda x: 1e-3 * 0.99 ** (x+NO_EPOCHS))# 设置早停
earlystopper = EarlyStopping(monitor='loss', patience=PATIENCE, verbose=VERBOSE)#
checkpointer = ModelCheckpoint('best_model.h5',monitor='val_accuracy',verbose=VERBOSE,save_best_only=True,save_weights_only=True)
history=model.fit_generator(Generator(trainpath, batch_size),steps_per_epoch=int(m) // batch_size,epochs=NO_EPOCHS,verbose=1,shuffle=True,callbacks=[earlystopper, checkpointer, annealer])# 保存训练模型model.save('best_model.h5')
# score = model.evaluate(Generator(testpath, batch_size), verbose=0)
score = model.evaluate_generator(Generator(testpath,batch_size),steps=int(m2) // batch_size)
print(history.history.keys())
plt.plot(history.history['acc'])
# plt.plot(history.history['val_accuracy'])
plt.title('model1 accuracy')
plt.ylabel('accuracy')
plt.xlabel('epoch')
plt.legend(['trian','test'],loc='upper left')
plt.show()plt.plot(history.history['loss'])
# plt.plot(history.history['val_loss'])
plt.title('model1 loss')
plt.ylabel('loss')
plt.xlabel('epoch')
plt.legend(['trian','test'],loc='upper left')
plt.show()