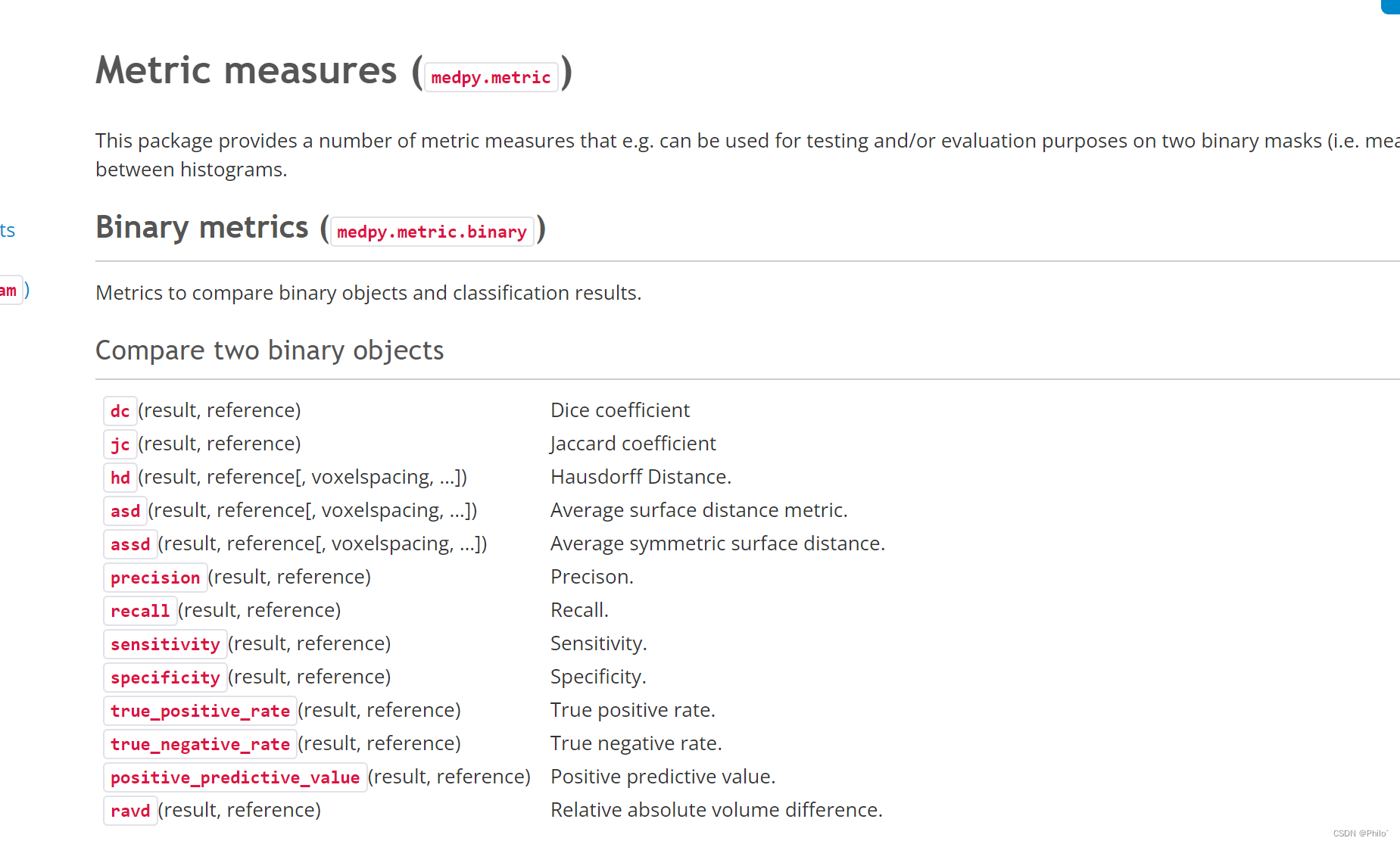
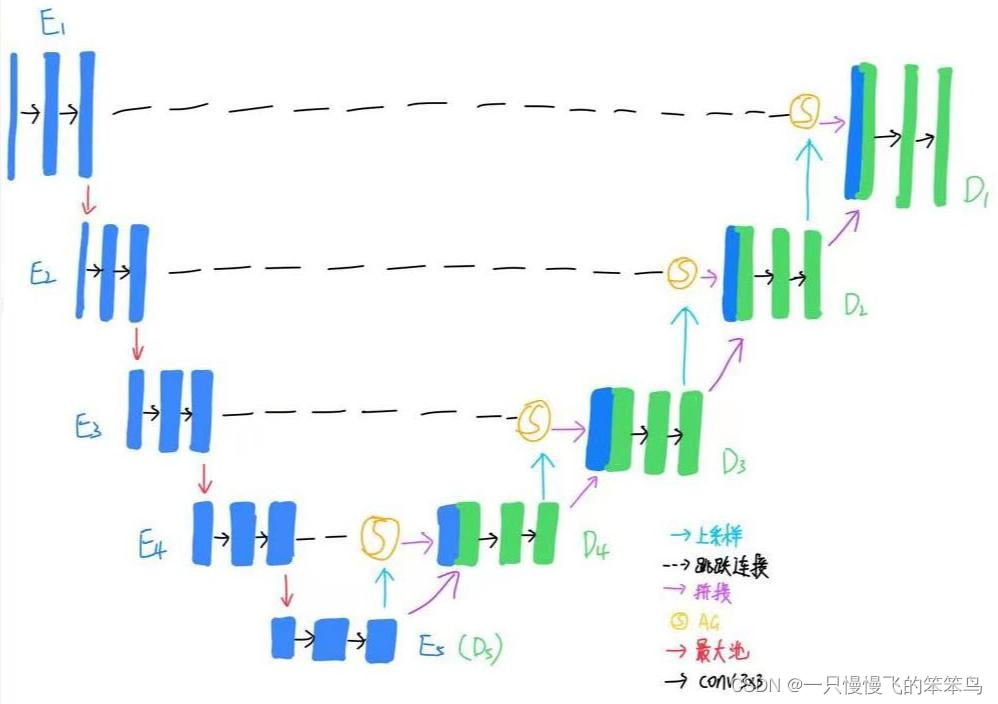
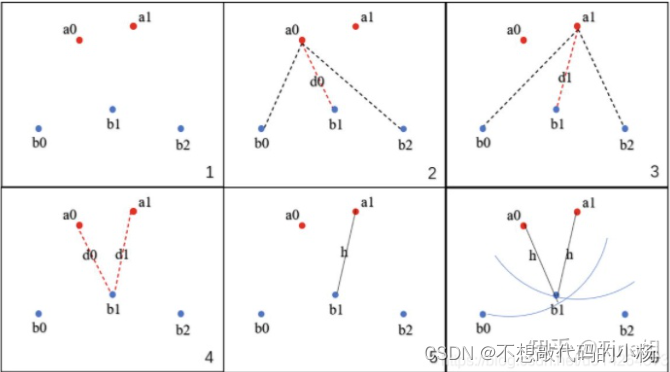
医学图像笔记(四)医学图像分割
- 1、医学图像分割的开源工具
- 2、其他分割
- 2.1、3D VNet
- 2.2、PE-VNet
- 2.3、基于PyTorch的3D 医学图像分割
- 2.4、3D UX-Net:超强的医学图像分割新网络
- 2.5、医学图像分割优质开源代码
- 3、医学图像数据集
- 3.1、百度AI studio 数据集
- 3.2、Github上哈佛 beamandrew机器学习和医学影像研究者贡献的数据集
- 4、医学图像分割优质开源代码
传统图像分割算法:graphcut、growcut、grabcut、、、
基于深度学习的分割算法:
1、医学图像分割的开源工具
nnUNet
MONAI
2、其他分割
2.1、3D VNet
2.2、PE-VNet

2.3、基于PyTorch的3D 医学图像分割
# Python libraries
import argparse
import os# Lib files
import lib.medloaders as medical_loaders
import lib.medzoo as medzoo
import lib.train as train
import lib.utils as utils
from lib.losses3D import DiceLossdef main():args = get_arguments()utils.make_dirs(args.save)training_generator, val_generator, full_volume, affine = medical_loaders.generate_datasets(args,path='.././datasets')model, optimizer = medzoo.create_model(args)criterion = DiceLoss(classes=args.classes)if args.cuda:model = model.cuda()print("Model transferred in GPU.....")trainer = train.Trainer(args, model, criterion, optimizer, train_data_loader=training_generator,valid_data_loader=val_generator, lr_scheduler=None)print("START TRAINING...")trainer.training()def get_arguments():parser = argparse.ArgumentParser()parser.add_argument('--batchSz', type=int, default=4)parser.add_argument('--dataset_name', type=str, default="iseg2017")parser.add_argument('--dim', nargs="+", type=int, default=(64, 64, 64))parser.add_argument('--nEpochs', type=int, default=200)parser.add_argument('--classes', type=int, default=4)parser.add_argument('--samples_train', type=int, default=1024)parser.add_argument('--samples_val', type=int, default=128)parser.add_argument('--inChannels', type=int, default=2)parser.add_argument('--inModalities', type=int, default=2)parser.add_argument('--threshold', default=0.1, type=float)parser.add_argument('--terminal_show_freq', default=50)parser.add_argument('--augmentation', action='store_true', default=False)parser.add_argument('--normalization', default='full_volume_mean', type=str,help='Tensor normalization: options ,max_min,',choices=('max_min', 'full_volume_mean', 'brats', 'max', 'mean'))parser.add_argument('--split', default=0.8, type=float, help='Select percentage of training data(default: 0.8)')parser.add_argument('--lr', default=1e-2, type=float,help='learning rate (default: 1e-3)')parser.add_argument('--cuda', action='store_true', default=True)parser.add_argument('--loadData', default=True)parser.add_argument('--resume', default='', type=str, metavar='PATH',help='path to latest checkpoint (default: none)')parser.add_argument('--model', type=str, default='VNET',choices=('VNET', 'VNET2', 'UNET3D', 'DENSENET1', 'DENSENET2', 'DENSENET3', 'HYPERDENSENET'))parser.add_argument('--opt', type=str, default='sgd',choices=('sgd', 'adam', 'rmsprop'))parser.add_argument('--log_dir', type=str,default='../runs/')args = parser.parse_args()args.save = '../saved_models/' + args.model + '_checkpoints/' + args.model + '_{}_{}_'.format(utils.datestr(), args.dataset_name)return argsif __name__ == '__main__':翻译原稿来自:https://theaisummer.com/medical-image-deep-learning/
iSeg-2017 challenge:https://iseg2017.web.unc.edu/
MedicalZooPytorch:https://github.com/black0017/MedicalZooPytorch
参考:医学成像中的深度学习——基于PyTorch的3D 医学图像分割
2.4、3D UX-Net:超强的医学图像分割新网络
Title: 3D UX-Net: a Large Kernel Volumetric ConvNet Modernizing Hierarchical Transformer for Medical Image Segmentation
Author: Ho Hin Lee et al. (范德堡大学)
Paper: https://arxiv.org/abs/2209.15076
Github: https://github.com/MASILab/3DUX-Net
ICLR 2023 新鲜出炉的 3D 医学图像分割之星——3D UX-Net。
3D UXNet 是一种轻量级3D卷积神经网络,其使用 ConvNet 模块调整分层 Transformer 以实现稳健的体素分割,在三个具有挑战性的脑体积和腹部成像公共数据集与当前的 SOTA 模型如 SwinUNETR 对比,同时在以下三大主流数据集性能均达到了最优:
MICCAI Challenge 2021 FLARE
MICCAI Challenge 2021 FeTA
MICCAI Challenge 2022 AMOS
具体的,相比于 SwinUNETR,3D UX-Net 将 Dice 从 0.929 提高到 0.938 (FLARE2021),Dice 从 0.867 提高到 0.874 (Feta2021)。此外,为了进一步评估 3D UX-Net 的迁移学习能力,作者在 AMOS2022 数据集上训练得到的模型在另一个数据集上取得了 2.27% Dice 的提升(0.880 → 0.900)。
参考:ICLR 2023 | 3D UX-Net:超强的医学图像分割新网络
2.5、医学图像分割优质开源代码
参考:医学图像分割优质开源代码
3、医学图像数据集
3.1、百度AI studio 数据集
医学影像数据集集锦
3.2、Github上哈佛 beamandrew机器学习和医学影像研究者贡献的数据集
beamandrew/medical-data(这是Github上哈佛 beamandrew机器学习和医学影像研究者贡献的数据集,包括了医学影像数据、竞赛数据、来自电子健康记录的数据、医疗数据、UCI数据集、生物医学文献等。)
方法模型
Medical Image Segmentation subtasks
4、医学图像分割优质开源代码
Swin-Unet:Unet形状的纯Transformer的医学图像分割
Bidirectional Projection Network for Cross Dimension Scene Understanding
Paper(Oral): https://arxiv.org/abs/2103.14326
Code: https://github.com/wbhu/BPNet
Semantic Segmentation for Real Point Cloud Scenes via Bilateral Augmentation and Adaptive Fusion
Paper: https://arxiv.org/abs/2103.07074
Code: https://github.com/ShiQiu0419/BAAF-Net
Cylindrical and Asymmetrical 3D Convolution Networks for LiDAR Segmentation
Paper: https://arxiv.org/abs/2011.10033
Code: https://github.com/xinge008/Cylinder3D
Towards Semantic Segmentation of Urban-Scale 3D Point Clouds: A Dataset, Benchmarks and Challenges
Homepage: https://github.com/QingyongHu/SensatUrban
Paper: http://arxiv.org/abs/2009.03137
Code: https://github.com/QingyongHu/SensatUrban
Dataset: https://github.com/QingyongHu/SensatUrban
参考:3D语义分割(3D Semantic Segmentation)
参考:医学图像分割优质开源代码v


![[转]医学图像分割综述](https://img-blog.csdnimg.cn/20190730135615475.png?x-oss-process=image/watermark,type_ZmFuZ3poZW5naGVpdGk,shadow_10,text_aHR0cHM6Ly9ibG9nLmNzZG4ubmV0L3dlaXhpbl80MDc3OTcyNw==,size_16,color_FFFFFF,t_70)